干货| CUT and Tag 技术全方位解答
诺唯赞
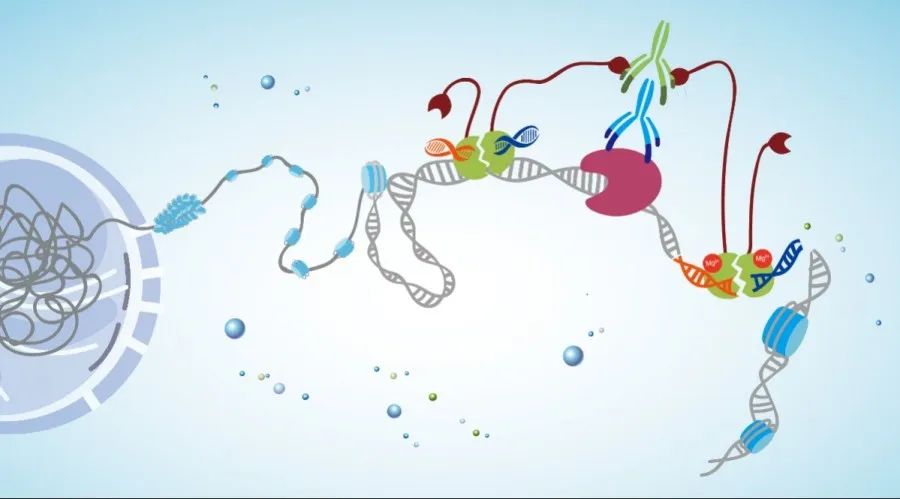
1. CUT&Tag 适用于什么物种?如果需要做植物 CUT&Tag ,需要怎么操作?
CUT&Tag 适用于哺乳动物细胞的蛋白-DNA 互作研究,酵母、植物等细胞需要经过(破除细胞壁或者提取细胞核)来进行实验。CUT&Tag 在哺乳动物细胞系上的应用比较成熟,动物组织经过处理得到悬浮单个细胞同样可以进行实验。植物进行 CUT&Tag 操作可以参照 Low-input chromatin profiling in Arabidopsis endosperm using CUT&RUN (PMID:30719569) 的前端处理。
2. CUT&Tag 技术公布的 Protocol 适用于活细胞,对细胞状态有要求吗?那么经过 PFA 交联过的细胞可以使用吗?
CUT&Tag 在活细胞上确实成功率比较高,在准备细胞时需要保证细胞处于高细胞活性状态,若细胞活性不佳,胞内蛋白质与核酸相互作用的状态会发生改变,对于死亡的细胞,目的蛋白有可能从染色质上脱落,进而影响实验数据。在准备细胞样本时使用台盼蓝进行细胞活性的检测,建议细胞活力大于 90% 以上。经过 PFA 交联过的细胞同样也适用于 CUT&Tag ,但我们不建议使用 PFA 交联的细胞进行实验,因为经过交联的细胞有可能会引起表位覆盖,从而降低数据质量。
3.针对于 Tn5 切割开放染色质区域的特性,CUT&Tag 是否更加适合于开放染色质区域的研究,异染色质上的位点是否适用?
CUT&Tag 是 ChIP-seq 的替代技术,主要用于分析全基因组范围内的 DNA-蛋白质相互作用。容易将 ATAC-seq 技术与CUT&Tag 技术混淆,CUT&Tag 技术在异染色质位点上有很好的结果,Henikoff 推荐的阳性对照就是存在于异染色质区域的 H3K27me3,且我们的客户在 H3K27me3 以及 H3K27AC 上都有很好的结果。
4. ConA beads 的作用是什么?如果没有 ConA beads ,能否用离心法代替?
ConA beads 是经过 Concanavalin A(刀豆蛋白A,ConA)包被的磁珠。ConA 是一种从刀豆和豌豆等籽粒中提取出来的植物凝集素,四聚体球蛋白,含有糖基结合位点,因此 ConA 是一种糖类结合蛋白。ConA 磁珠能与细胞膜上的糖蛋白结合,从而吸附细胞,使对细胞的实验操作可视化,减少在后续实验过程中的细胞的损失。
不建议使用离心法代替 ConA beads,因为离心法不适合于低细胞数,在洗涤的过程中,损失很大,而且大肠杆菌残留的 DNA 无法进行可靠的校准。
5.做 ChIP-seq 的时候不需要二抗,CUT&Tag 二抗的作用是什么?二抗是必须的吗?可以用带修饰(比如 HRP 修饰)的二抗进行实验吗?
ChIP-seq 和 CUT&Tag 都可以研究基因组范围内的 DNA-蛋白质互相作用,但是这个是两个完全不同的技术。ChIP-seq 需要一抗与 pA/pG 磁珠进行结合,钓取目的区域。CUT&Tag 实验中的二抗作用是放大信号,在丰度很高的靶蛋白不使用二抗也可以构建出 CUT&Tag 文库,建议使用二抗。不建议使用带修饰的二抗进行实验,防止因为表位的覆盖影响二抗与 pA/pG-Tn5 结合的效率。
6.做 CUT&Tag 实验的时候,阴性对照和阳性对照怎么设置,IgG 的阴性对照有意义么?
针对抗体的阳性对照可以采用 RNA Pol II/ 组蛋白,证明系统没有问题。阴性对照使用与一抗相同种属的 IgG ,阳性对照,阴性对照证明抗体特异,实验结果可信。
7.如何区分 DNA 提取步骤里提取到的目的区域和非目的区域?
被提取的 DNA 片段,只有加上了接头的 DNA 片段才能在后续的 PCR 扩增中富集,通过 PCR 扩增可以特异性的富集目的片段。
8. CUT&Tag 实验完成后,怎么进行质控?
可以用 Qubit 进行浓度检测,Agilent2100 等可以检测片段分布的仪器进行文库峰型检测(如果研究组蛋白修饰,可以得到梯状条带分布)。如果没有检测片段分布的仪器,可以考虑使用 2% 的琼脂糖凝胶电泳代替,但是灵敏度会低,组蛋白修饰实验也有可能看不到典型的梯状条带。如果做转录因子,上机之前可以根据 ChP-seq 数据库里面的 peak 设计引物检测与 GAPDH 等内参位点对照,通过 qPCR 来确定目的靶点的富集程度。
9. CUT&RUN 与 CUT&Tag 实验的主要区别是什么?
CUT&RUN 中的功能酶是融合酶 pA-MNase ,使用 Ca2+ 激活,在 0℃ 切割。CUT&RUN 实验中的二抗为选择项。回收到的 DNA 为片段化的 DNA ,后续需要接传统的 DNA 文库构建,才能得到 CUT&RUN 文库。CUT&Tag 中的功能酶是融合酶 pA/pG-Tn5 ,该酶由 Mg2+ 在 37℃ 激活转座酶切割 DNA ,实验时需要加入二抗进行信号放大。回收到的 DNA 已经带有测序引物结合接头,可直接进行 PCR 扩增得到 CUT&Tag 文库。
10.对于 CUT&Tag 的实验样本,二代测序上机应该如何选择 ?测序公司反馈文库有大片段分布怎么处理?需要做文库分选吗?
使用常规的 PE150 即可,主流的测序平台如 HiSeq X10 以及 Novaseq 均可。CUT&Tag 文库分布会有一定的大片段,组蛋白修饰呈现典型的梯子状或做转录因子经过了 qPCR 验证有富集,可以选择风险上机。不需要做文库分选,数据产生方面会有些波动。
文章图文来源:诺唯赞生物