重磅 | CUT and Tag 成功应用于植物领域!
诺唯赞
CUT&Tag(Cleavage Under Targets and Tagmentation)【1】是一种研究蛋白质-DNA 相互作用的新技术。与传统的染色质免疫沉淀(Chromatin Immunoprecipitation, ChIP)相比,CUT&Tag 具有信噪比高、可重复性好、实验周期短(1 天实现从细胞到文库构建)、细胞投入量低等显著优势。该方法一经问世,便受到广大科研工作者的青睐。
在过去的一年时间里,科研工作者在动物、植物细胞中成功应用 CUT&Tag 的成果已经在线发表。2020 年 1 月 13 日,北京大学系统生物医学研究所尹玉新课题组在国际免疫学领域顶级期刊 Nature immunology 发表了题为 The phosphatase PAC1 acts as a T cell suppressor and attenuates host antitumor immunity 的研究论文,研究人员初时使用 ChIP-Seq实验时无法得到正常的结果,通过尝试,最终利用 CUT&Tag 技术(Vazyme #S603)成功获得了 NuRD 复合体核心成分CHD4 与 DNA 的结合谱【2】。
由于植物存在细胞壁、大液泡以及复杂次生代谢产物,在进行 CUT&Tag 实验之前,需要分离出高质量的细胞核,所以植物组织中 CUT&Tag 的应用非常具有挑战性。近日,浙江大学农学院的关雪莹教授团队根据动物细胞中 CUT&Tag 的实验,开发了一套在植物中进行 CUT&Tag 的方法,为植物中染色质状态的表观遗传研究提供了一个比 ChIP 更具优势、操作性更强的方法。相关研究成果以 Efficient Chromatin Profiling of H3K4me3 Modification in Cotton Using CUT&Tag 为题发表在 Plant Methods 上。

1.研究过程
该研究以棉花为模型系统,棉花植物(棉属)带有种皮毛(棉纤维),是全世界重要的商品。由于 ChIP-Seq 对起始量需求高( 20µg 染色质),棉纤维中表观基因组修饰的概况分析比较困难。关雪莹教授团队在这次研究中使用了 CUT&Tag(Vazyme #S602/S603),仅需要常规ChIP策略所需原料的约 1/20,成功获得了棉纤维中表观基因组修饰的分析。并且,在实验操作过程中不需要进行实验材料的交联和 DNA 超声处理的步骤,提取的 DNA 直接进行 PCR 扩增便可得到完整的文库,大大简化了操作流程(见图 1 ),节省了实验时间,两天内即可完成实验。
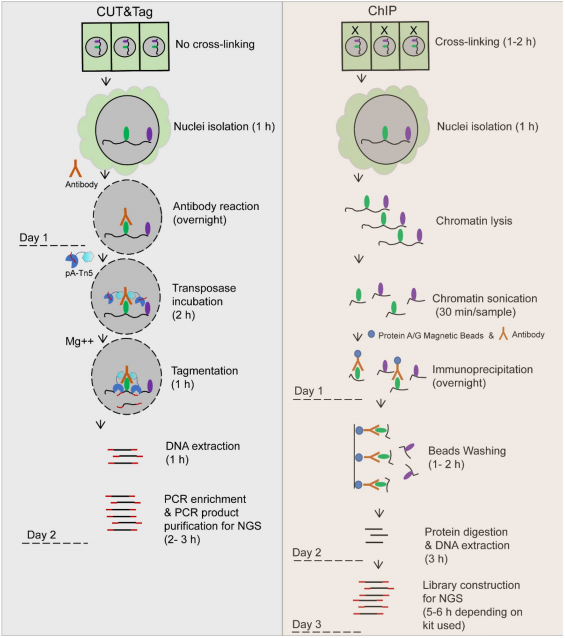
图 1. CUT&Tag 与 ChIP-Seq 流程比较图【3】
2.结果分析——CUT&Tag VS ChIP-Seq
以 H3K4me3 组蛋白修饰为例,研究人员比较了相同材料同时进行 CUT&Tag与ChIP-Seq 的数据表现。该课题组首先对 CUT&Tag 和 ChIP 组进行了相关分析(见图2),发现 CUT&Tag 与 IgG 对照的相关性非常低(r = 0.01),而 ChIP-Seq 组与 mock 对照组的相关性高(r=0.89),表明 CUT&Tag 数据信噪比高。同时研究人员进行了 CUT&Tag 样本重复相关性分析,发现 CUT&Tag 样本之间的重复性非常高(r=0.97)。
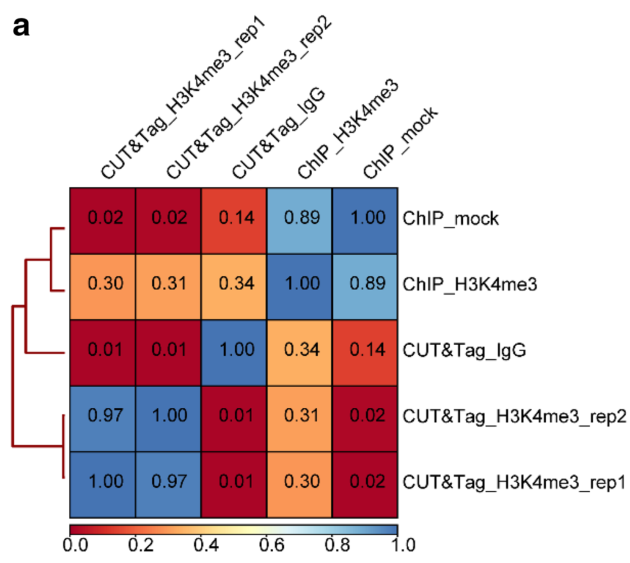
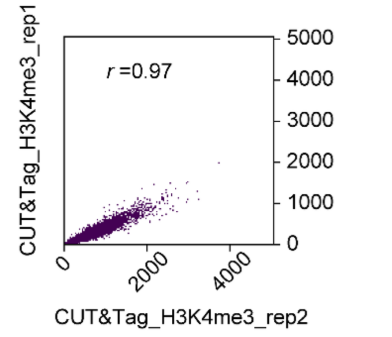
图 2. CUT&Tag 与 ChIP-Seq 相关性比较【3】
同时为了比较 CUT&Tag 与 ChIP-Seq 的数据信噪比,研究人员从每个样本中随机选取了 6-24 M Clean reads 进行 Peak calling 分析,发现截取 6 M Clean Reads 的 CUT&Tag 数据得到的 peak 数目与截取 24 M Clean Reads 的 ChIP-Seq 数据得到的 peak 数目基本一致。
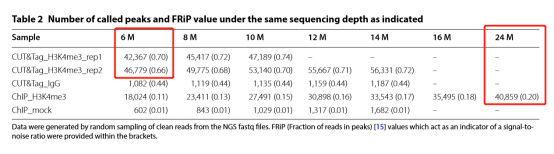
图 3. CUT&Tag 与 ChIP-Seq 不同 Clean Reads 获得的 peaks 数目比较【3】
研究人员截取 6 M Clean reads(CUT&Tag)和 24 M Clean reads(ChIP-Seq)进行数据分析,有 25,597 个 peak 是两种方法结果共有的,重复比率为 54.7–62.6%。而 CUT&Tag 的两个重复实验之间共有 37,168 个 peak ,比率高达 79.5–87.7%,这表明 CUT&Tag 数据的两个重复具有很高的重复性。
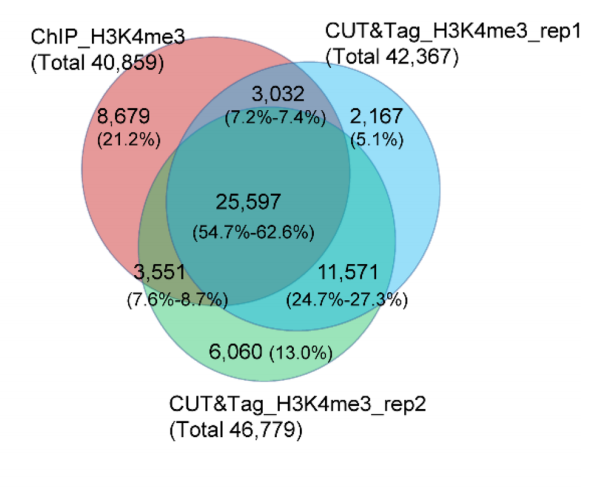
图 4. CUT&Tag 与 ChIP-Seq 共有 peak 分析【3】
研究人员进一步的分析了 1600kb 左右的基因组区域的 H3K4me3 信号,发现 CUT&Tag 信号在分辨率和灵敏度上均优于 ChIP-Seq 信号。
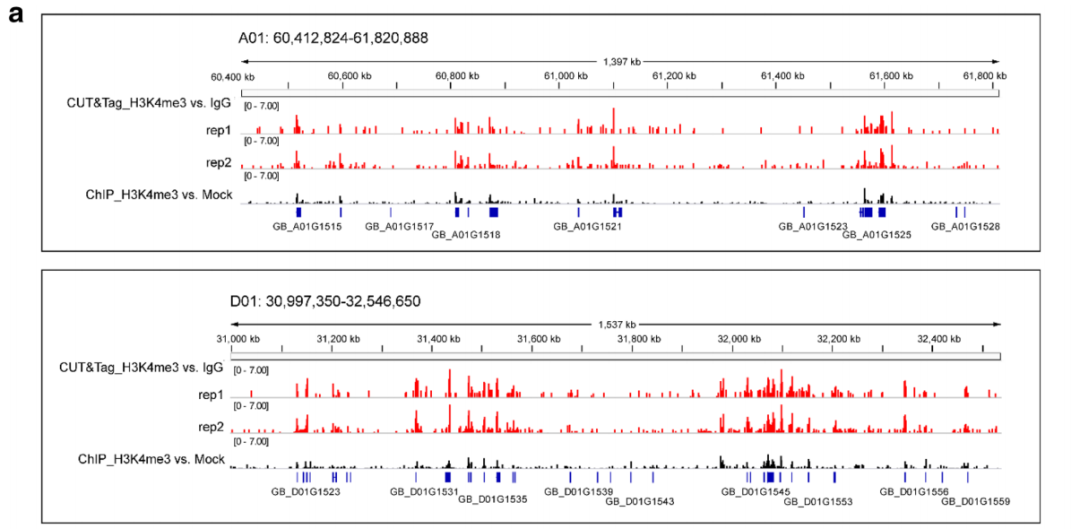
图 5. CUT&Tag 与 ChIP-Seq IGV 视图比较【3】
该研究成果为进一步研究植物中的表观基因组学提供了一个有前途的策略。在操作简单性和所需的时间方面,CUT&Tag 优于传统染色质分析方法。更重要的是 CUT&Tag 需要更少的起始材料,更少的测序数据得到更高质量的数据。
参考文献:
【1】 Kaya-Okur HS, Wu SJ, Codomo CA, Pledger ES, Bryson TD, Henikoff JG, Ahmad K, Henikoff S: CUT&Tag for efficient epigenomic profiling of small samples and single cells. Nat Commun 2019, 10(1):1930.
【2】 Lu D, Liu L, Sun Y, et al. The phosphatase PAC1 acts as a T cell suppressor and attenuates host antitumor immunity[J]. Nature Immunology, 2020: 1-11.
【3】 Tao X, Feng S, Zhao T, et al. Efficient chromatin profiling of H3K4me3 modification in cotton using CUT&Tag[J]. Plant Methods, 2020, 16(1): 1-15.
文章图文来源:诺唯赞生物