CRISPR/Cas 是进行基因编辑的强大工具,可以对基因进行定点的精确编辑。在向导 RNA(guide RNA, gRNA)和 Cas9 蛋白的参与下,待编辑的细胞基因组 DNA 将被看作病毒或外源 DNA,被精确剪切。
一、寻找目的基因的靶标
使用在线设计网站 CRISPR direct,如需直接复制网址,可在生物学霸后台对话框回复 direct即可。
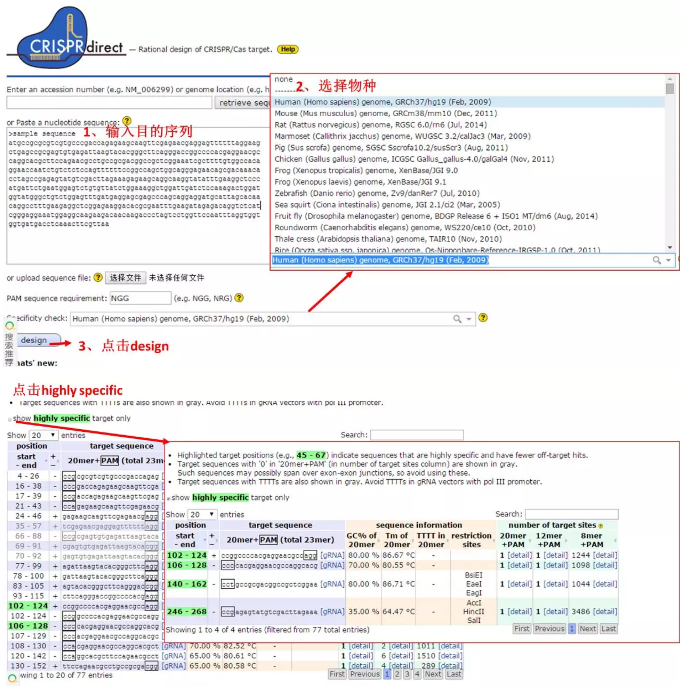
靶点挑选要点:
基因敲除靶点应设计在起始密码子附近(包括起始密码子)或者起始密码子下游的外显子范围内。
不同 Cas9/gRNA 靶点在基因敲除效率上有较大差异,因此同时设计构建 2~3 个靶点的基因敲除载体再从中选出敲减效果较佳的靶点。
N1-N20 NGG 靠近 PAM 的碱基对靶点的特异性很重要,前 7~12 个碱基的错配对 Cas9 切割效率影响较小。设计好的靶点序列应在基因库中进行 BLAST 检测。
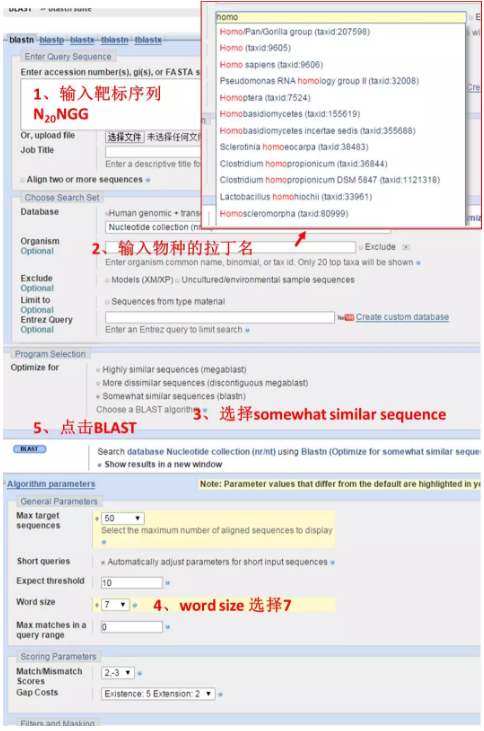
Cas9Nicknase 需要挑选成对的靶点。一般在正义链和反义链上分别挑选相距 20~30bp 的靶点配对。多对靶点的敲除效率常有较大差异。由于基因敲除实验时间长,在正式对目的细胞进行敲除前对靶点进行验证和挑选非常必要。
二、 插入片段设计

插入寡核苷酸序列设计(必须 PAGE 纯化寡核苷酸):
正向序列
5’ACACCGNNNNNNNNNNNNNNNNNNNGTTTTAGAGCTAGAAATAGCAAGTTAAAATAAGGCTAGTCCGTT3’
反向序列
3’TGTGGCNNNNNNNNNNNNNNNNNNNCAAAATCTCGATCTTTATCGTTCAATTTTATTCCGATCAGGCAA5’
插入片段的合成
用水将寡核苷酸稀释为 100 μM。按以下体系配制退火反应体系:
正义寡核苷酸(100 μM)5μl
反义寡核苷酸(100 μM)5μl
NaCl 100 mM(终浓度)
Tris‐Cl pH7.4 50 mM(终浓度)
加水补足 50 μl
将配制好的退火反应缓冲液重复混合,短暂离心后放置 PCR 仪上,运行以下程序: 90℃ 4 min, 70℃ 10 min, 55℃ 10 min, 40℃ 10 min, 25℃ 10 min。退火后的寡核苷酸可以立刻使用或者在 ‐20℃ 长期保存。
三、 pCas9/gRNA 基因敲除载体的构建
用 EcoRV 酶切 2 μg pCas9/gRNA 载体(inovogen)。通常情况下用大约 20~30 单位的酶大约 3 小时可以酶切完全。酶切后我们建议用琼脂糖凝胶回收线性化载体。将回收后的线性化载体定量,通常线性化载体的工作浓度为 50~100ng/μl。连接用水将退火后双链寡核苷酸 (10 μM) 稀释 100 倍备用。
连接反应体系:
T4 DNA 连接酶 5U
EcoRV 5U
线性化载体 2 μl
稀释 100 倍后双链寡核苷酸 1 μl
10× 连接酶 Buffer 1 μl
50% PEG4000 1 μl
加水补足 10 μl
反应条件: 22℃ 30 min, 37℃ 15 min。
注:
平末端连接效率较低,在连接体系中添加 PEG4000 可以提高连接效率。
在连接体系中添加 EcoRV 酶可以显著提高阳性率。
四、 转化大肠杆菌感受态细胞及通过载体上的引物进行 PCR 鉴定阳性克隆
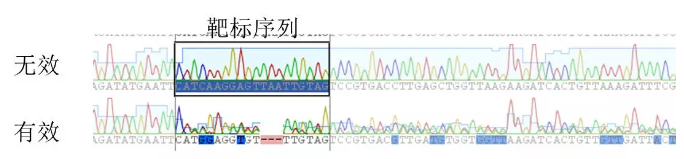
挑选阳性克隆进行测序验证, 验证正确后, 提取质粒进行转染试验, 关于 sgRNA 是否有效。可以转染之后,直接提取 DNA,通过测序验证,如果在靶标位点附近开始出现杂乱的多峰说明是 sgRNA 有效,如果没有则该 sgRNA 无效。