以最火热的 p53 为例
Step1 先找 p53 基因序列
1. NCBI--输入 p53 并选择物种 human:tumor protein p53 [Homo sapiens (human)]
https://www.ncbi.nlm.nih.gov/gene
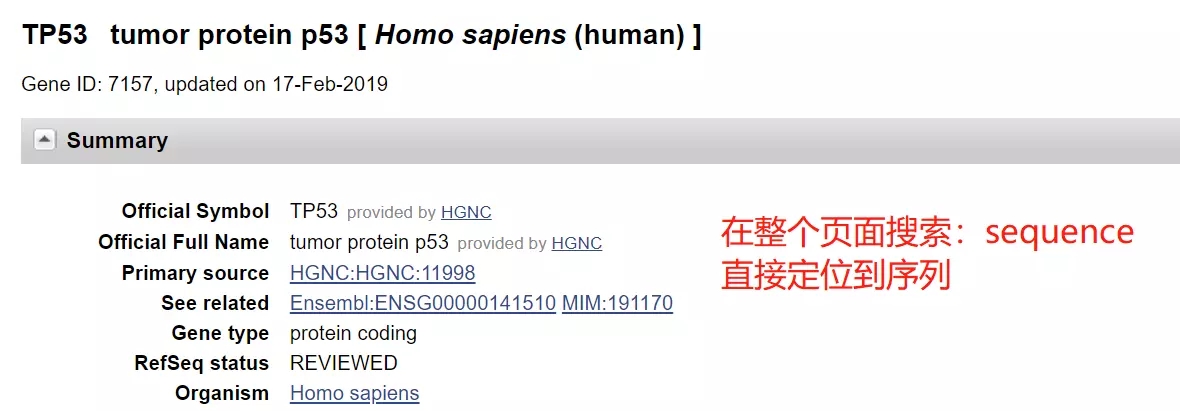
2. Ctrl+F,输入 sequence,快速定位(当然其实拉下去就是了),点击 Genebank
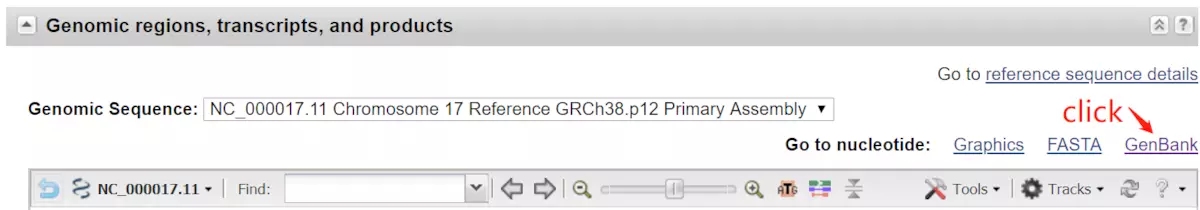
3. 下载 p53 基因序列

4. 用 SnapGene 软件打开序列,并简单了解每个元件
最好是可以学一下 SnapGene 这个软件的使用方法(有视频),个人认为,自行查资料理解每一个元件的含义和作用,可以帮助更快的补充背景知识。通过将散落的知识点集合起来,最后拼凑成自己的知识地图。
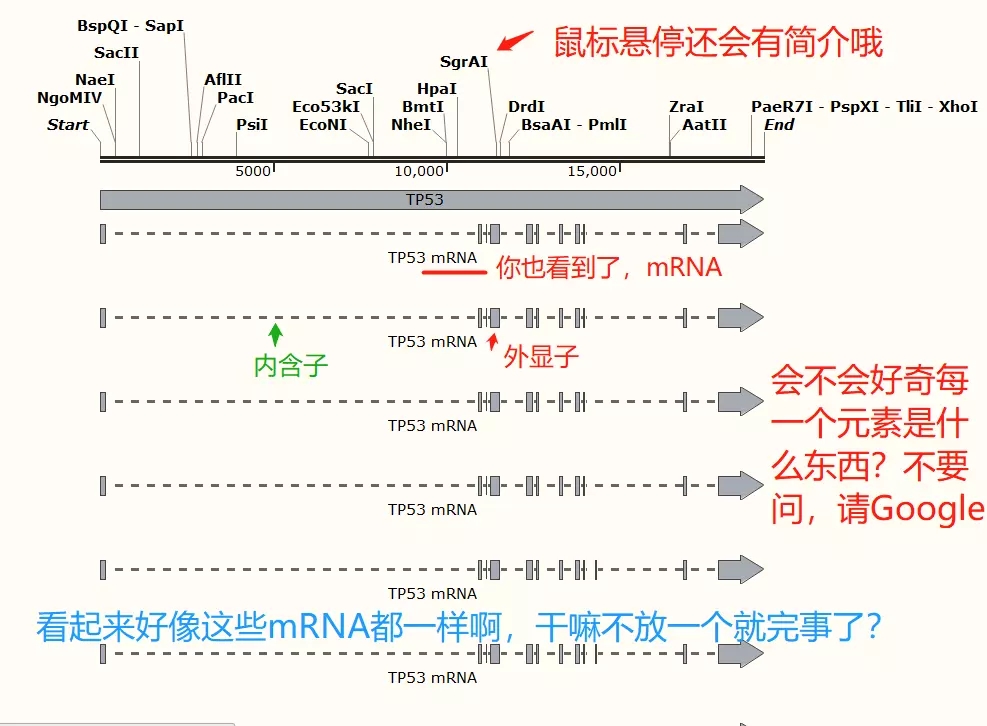
那么,为什么看起来好像这些 mRNA 都一样,怎么不只放一条完事了?不是滴。。。还有不一样的 mRNA。
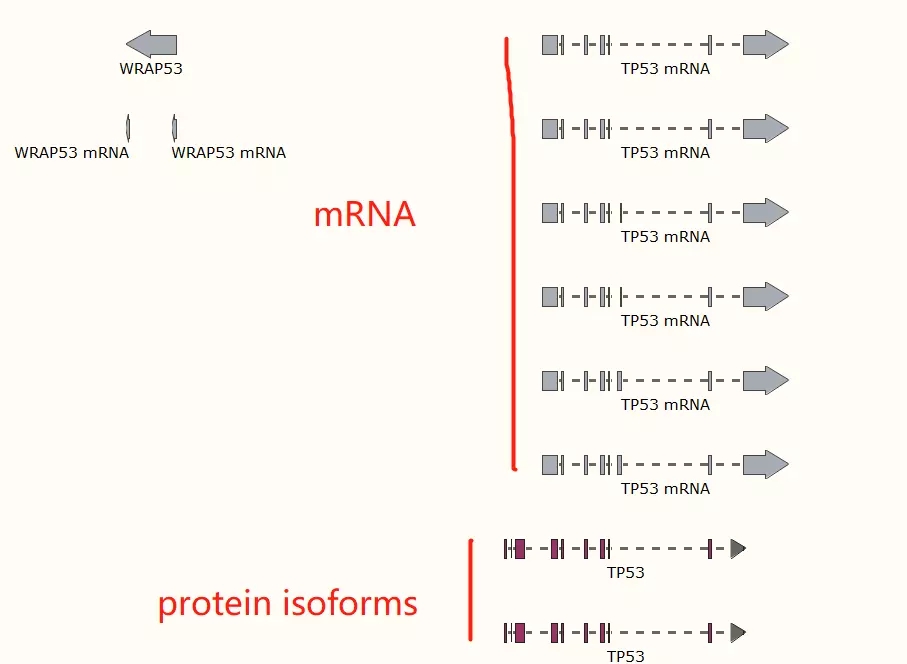
在学生信的时候,听到过一个词,「可变剪接」,有了初步印象之后,在这里才算是实实在在看到了,我认真的数了数,mRNA 画了 15 条,TP53 蛋白也画了 15 条。那么我们可以理解为,TP53 有 15 个亚型,当然,每个亚型都有自己的 mRNA。
Step2 分析敲除位点(找到合适的敲除位置)
怎么样才能通过尽量少的 sgNRA 数量,去敲除尽量多的 TP53 蛋白。既然是做 knockout,那一定是要所有的亚型都不能表达。那么既然如此,就是要敲除掉这些亚型共有的区域,而且越前面越好(如果只敲除尾巴,TP53 合成了半个身子,半个身子的 TP53 说不定也能起作用),另外,也不能敲除掉内含子(说是,如果敲掉了,可能会形成进的剪接,就有新的蛋白出现)----知识点好伐~
1. 发现所有亚型共有区域
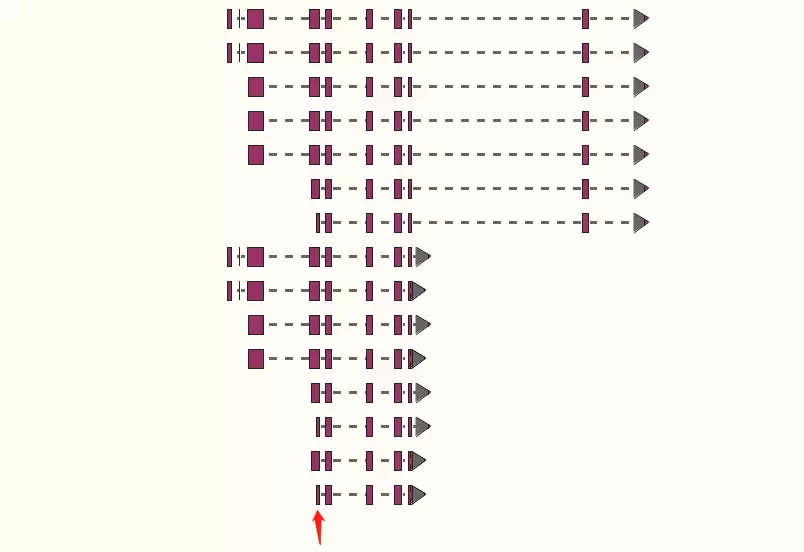
2. 查看详细信息,得知第一个 segment(片段)是从 12417 - 12498,一共 82 bp

3. 在 Sequence 中查看,复制一整个 segment 的碱基序列如下
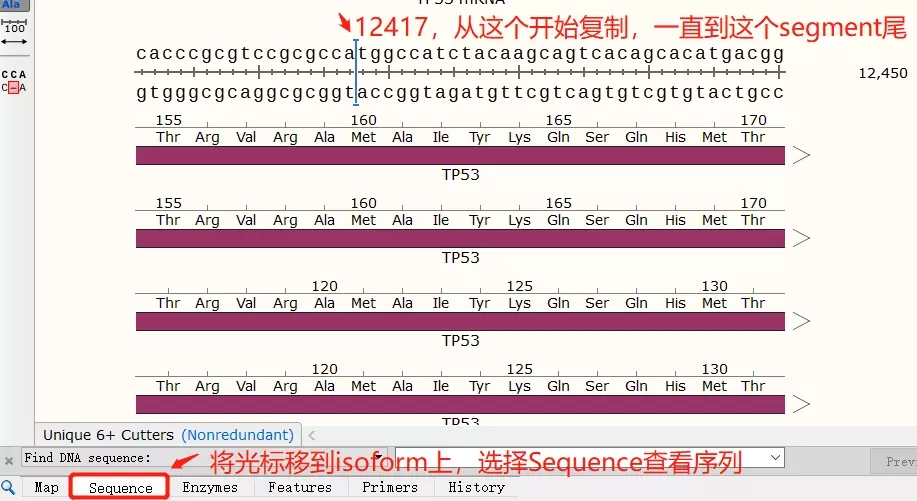
segment 碱基序列
atggccatctacaagcagtcacagcacatgacggaggttgtgaggcgctgcccccaccatgagcgctgctcagatagcgatg
Step3 设计 sgRNA
首先,先附上鼎鼎大名的张峰实验室提供的网页
https://zlab.bio/guide-design-resources
1. 打开网页,选择网页工具--博德研究所

2. 粘贴序列,一步到位

3. 查看结果
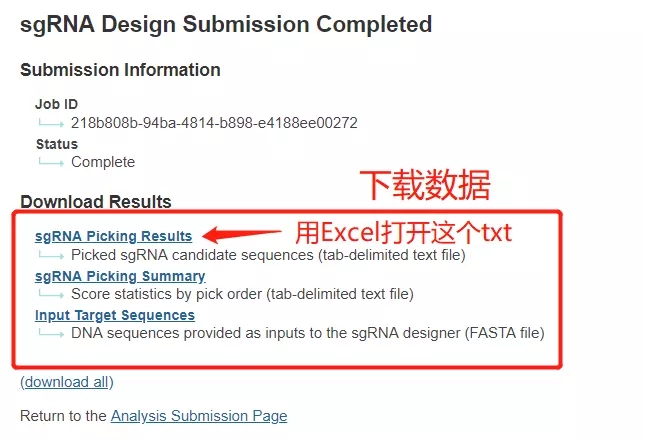
我没有写结果怎么查看,或者分析结果耶,因为。。。因为做到这里,真正想学的人,感兴趣的人早就去看了。
4. 选择目的序列
略