CRRSPR/Cas9基因敲除系统之sgRNA设计
百奥迈科生物
sgRNA设计网站介绍
sgRNA的在线设计网站有:
2、http://www.e-crisp.org/E-CRISP/;
等网站。这些网站虽然一定程度上满足了部分研究 者的需要,但仍然存在很多不足和缺陷。例如:
1、数据分析周期长;
2、无具体的脱靶数据;
3、可供选择物 种十分有限。
极大的影响了科研工作者的工作效率。鉴于此Biomics及时推出了本地化运行sgRNA软件设计服务,2个工作日内可给出sgRNA设计结果,包括详细的off‐target数据,且不限物种,提高科研工作效率!
设计要点
一、设计选项
1、基因信息或基因ID;
2、种属信息;
3、Cas9/sgRNA类型,比如spCas9 nickase需要设计一对sgRNA;
4、CRISPR类型,例如敲入、敲除、抑制、激活;
5、PAM类型,NGG或者NAG等。
二、基因敲除设计思路
1、根据相应基因及种属信息,在NCBI或ENSEMBLE中进行查找,并明确CDS的外显子部分;
2、确定待敲除位点,对于蛋白编码基因,如果该蛋白具 重要结构功能域,可考虑将基因敲除位点设计在编码该 结构域的外显子;如果不能确定基因产物性质,可选择将 待敲除位点放在起始密码子ATG后的外显子上。如果是microRNA, 可以将待敲除位点设计在编码成熟microRNA的外显子或在 编码成熟microRNA的外显子的5’和3’侧翼序列。
Biomics本地化设计软件
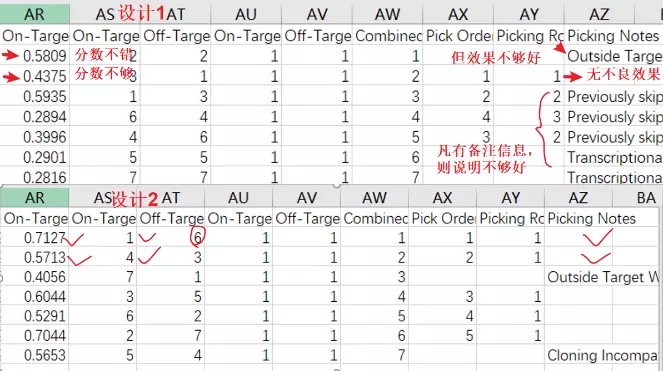
一、潜在靶点输出
百奥迈科sgRNA 设计软件数据输出包括sgRNA靶点序列,靶点全基因组潜在脱靶分析、外显子等信息。
二、脱靶数据分析
尽管现有sgRNA设计软件总结了一些规律,但仍然非常有限,因此预测sgRNA 的敲除效率仍然不尽如人意。Biomics sgRNA设计软件主要关注避免Cas9脱靶效应,以确保成功设计的sgRNA尽可能的减少脱靶效应。在此基础上通过试验筛选设计的sgRNA靶点,选择敲除效率高的sgRNA靶点用于最终的下游试验。