CRISPR-Cas9 基因敲除 sgRNA 设计好之后
春卷00
设计了 sgRNA 之后怎么办呢?还是这篇文章

先讲一下这里的框架
首先是实验设计

Experimental design
1. Target selection for sgRNA--选择靶点
详情见:CRISPR-Cas9 基因敲除 sgRNA 设计 https://www.jianshu.com/p/ 8222f449cb44
2. Approaches for sgRNA construction and delivery.
sgRNA 的合成和投递有两种方式:A)PCR;B)单独的 sgRNA 的表达质粒。
A)PCR 扩增的方法
U6:来源于人 U6 小核启动子,常用小 RNA 表达,转录本为 shRNA。
将 sgRNA 放到 U6 后面,利用 U6 的启动来扩增 sgRNA。
将这部分内容整合到 Cas9 expression plasmid pSpCas9(Cas9 表达质粒)中,一起共转。

B)sgRNA 表达质粒的方法
现在我要用这个方法来举例



设计 p53 引物
找到包括 sgRNA 序列的前后 1000 个碱基的序列(2000 bp)

找到的序列如下:

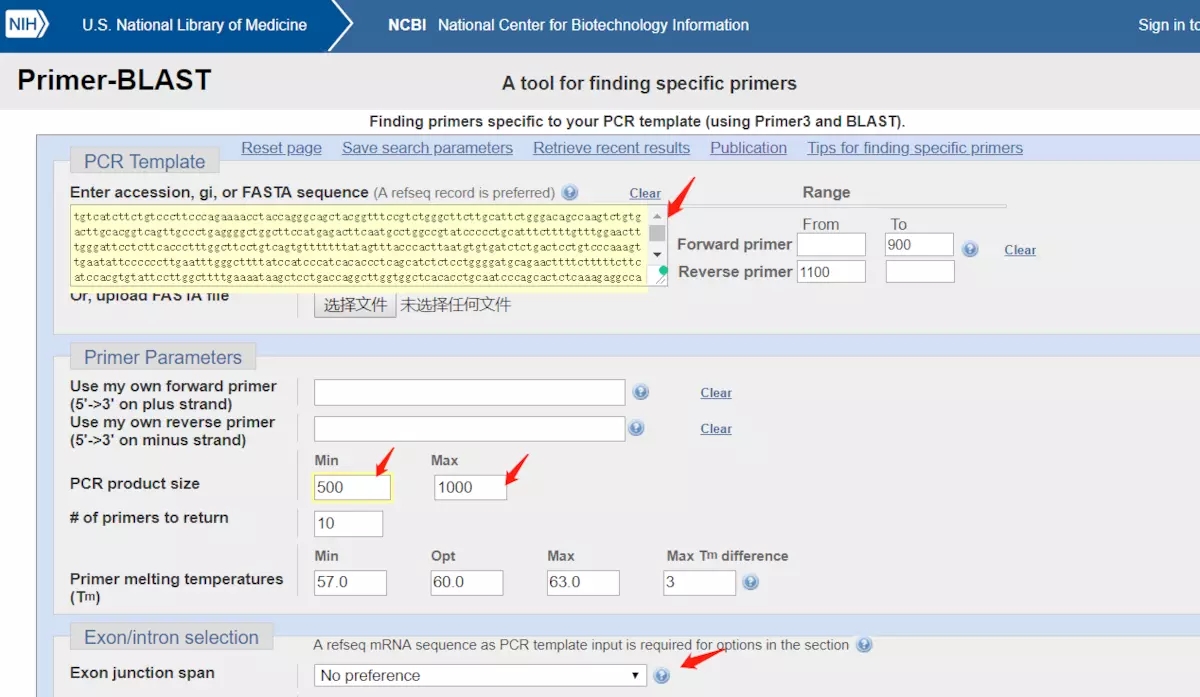
将上面复制的序列粘贴到 blast 中
https://www.ncbi.nlm.nih.gov/tools/primer-blast/
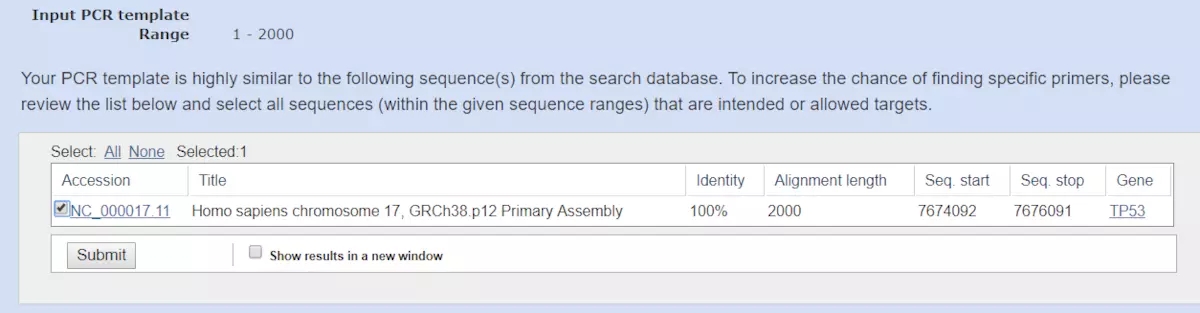
系统反馈说我提交的序列和数据中的某基因很像,问我是不是就用下面这个。
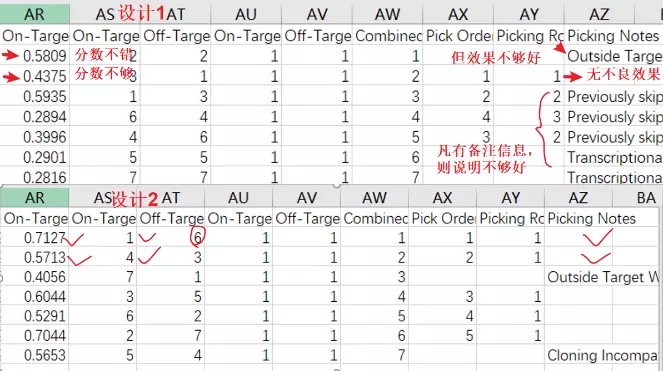
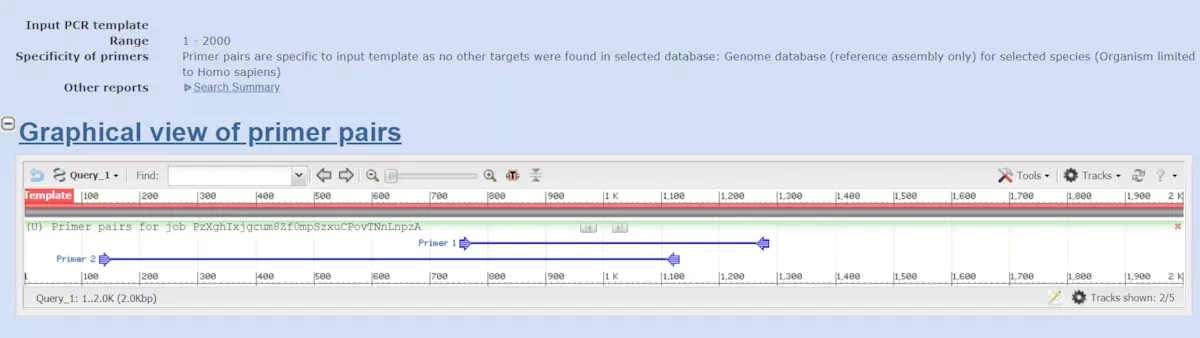
选和不选我都试过了,选择的话,得到的引物有两对,如下(摘选了其中一个):
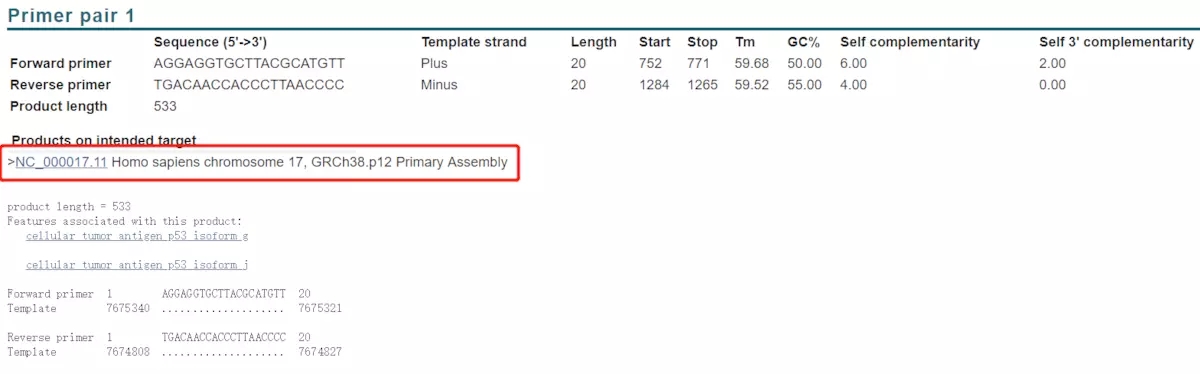
如果不选的话
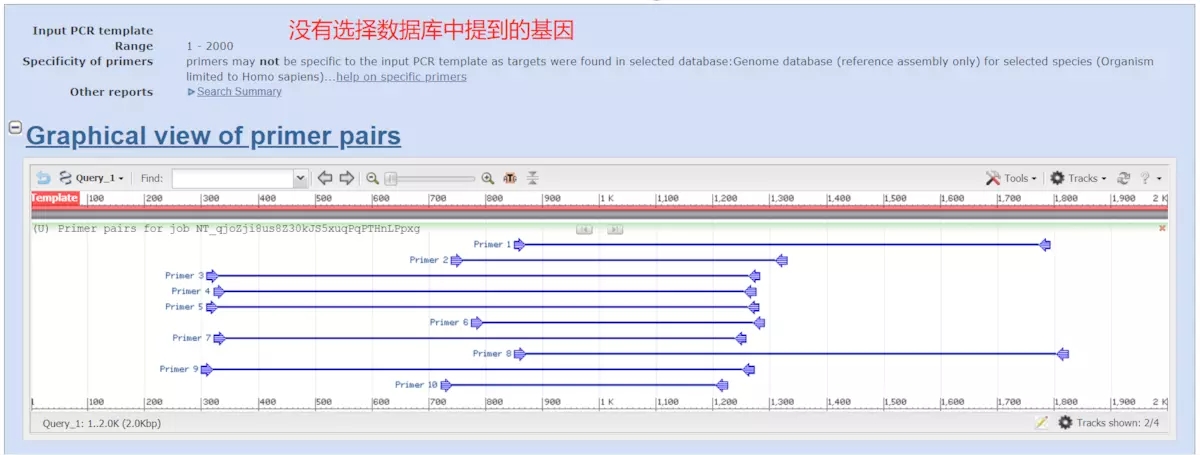
摘选一个引物:发现两种都可以得到比较好的引物,但是后者的引物有不少错配,前者的引物质量看起来更好(而且可以看到 primer 几乎是均匀的横跨 1 kb 大小,说明 map 到 target 上很好,后续验证 knockout 的时候也会好做)。

3. Design of repair template. 设计修复模板
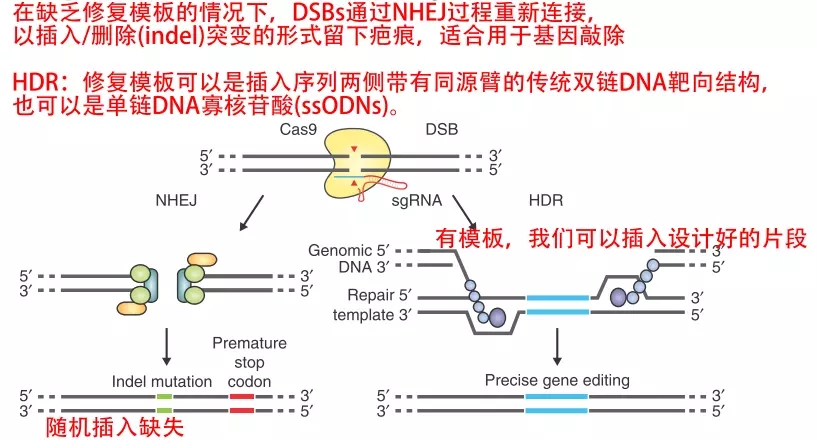
这几个词不知道要看多少遍,我承认,我不止一次看到它,但是我每次都无法精确理解其中的含义
error-prone NHEJ pathway
都说修复方式有两种
(1)NHEJ:nonhomologous end joining,非同源末端结合,这个方式在没有修复模板的情况下发生的。
(2)HDR:homology-directed repair,同源直接修复。这个方式是在有修复模板的情况下。
修复模板:
(1)plasmid-based donor repair templates that contain homology arms flanking the site of alteration,也就是有一个供体质粒,质粒有两端同源序列(互补 DSB 用),两段同源序列之间就是我们要插入或者修改的内容。同源臂通常大于 500 bp
(2)single-stranded DNA oligonucleotides (ssODNs),emm。。。就是需要两条单链寡核苷酸(ssODN),两臂也是带有同源序列(30~60 个碱基,但为了提高效率,最好是大于 40 个),在 Cas9 切开双链之后,模板的两臂同源序列直接互补(HDR,同源修复),从而完成了基因编辑。聪不聪明!
上传一张自己的理解图
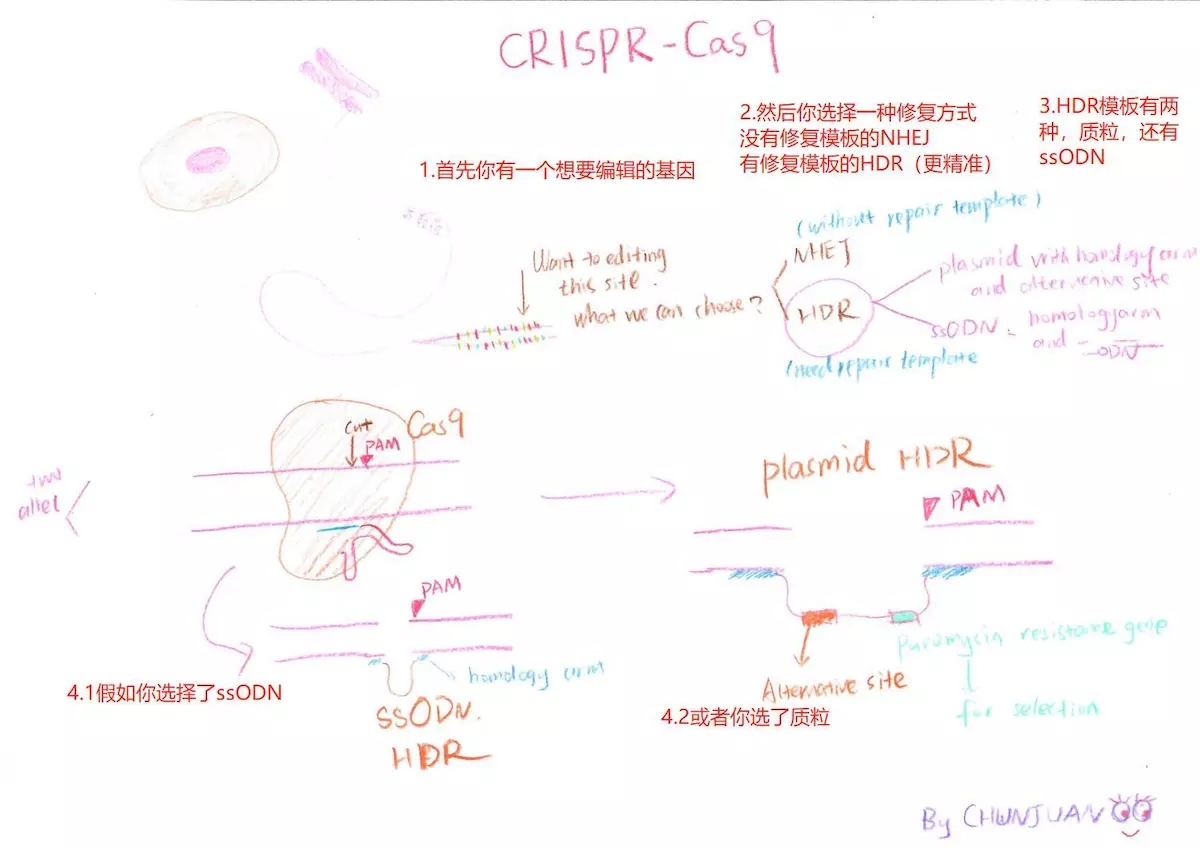
4. Clonal isolation of cell lines 将已编辑的细胞分离开来
这个很好理解,在编辑片段中加入抗生素抗性基因或者荧光基因后,可通过抗生素筛选或者 FACS 分离。
5. Functional testing 功能验证

(1)SURVEYOR nuclease assay
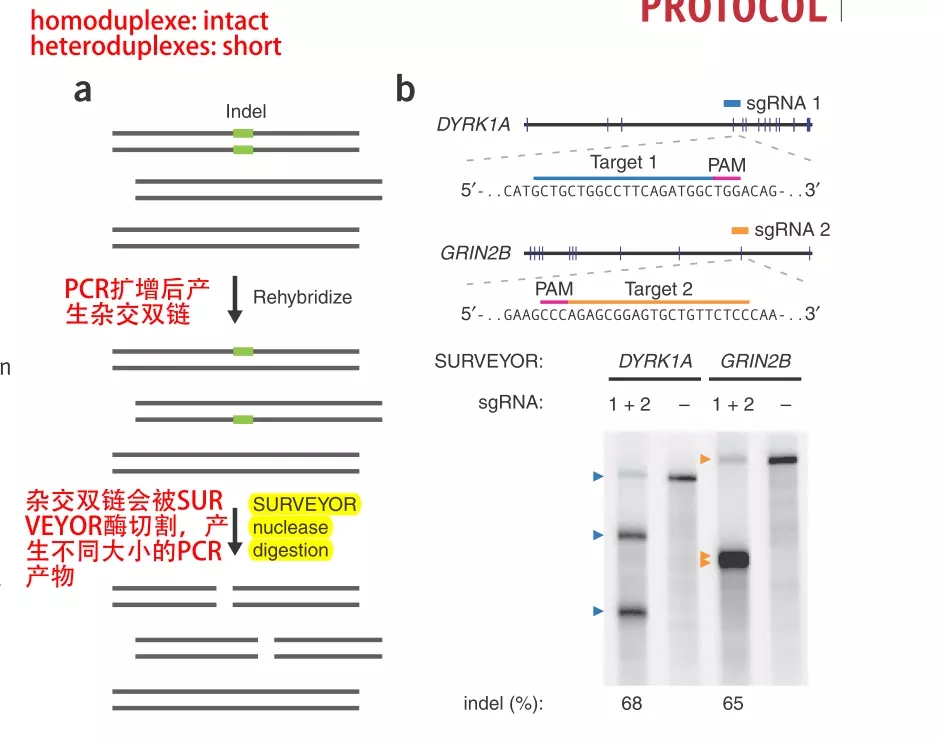
(2)Detection of indels or HDR by sequencing.