3 个回答
loveliufudan
主要有以下几点可能原因:
1. 引物设计的目标基因在NCBI的小黑麦数据库中没有存档记录。NCBI数据库中小黑麦的序列信息还不是非常完整。
2. 引物的特异性不高,除了目标基因,也能识别其他非特异性序列。可以调整引物设计,增强其特异性。
3. 目标基因在小黑麦中的表达丰度较低,NCBI中没有收录到这一基因的序列信息。可以考虑在更广泛的数据库或转录组数据库中检索。
4. 引物设计的参数需要优化,如Tm值、GC含量、自交联倾向等,应该设计一对Tm值接近、特异性更高的引物。
5. 检索的数据库范围需要扩大,不要限定在小黑麦的机构,可以在所有植物或更广泛的物种中检索。
龙大人驾到
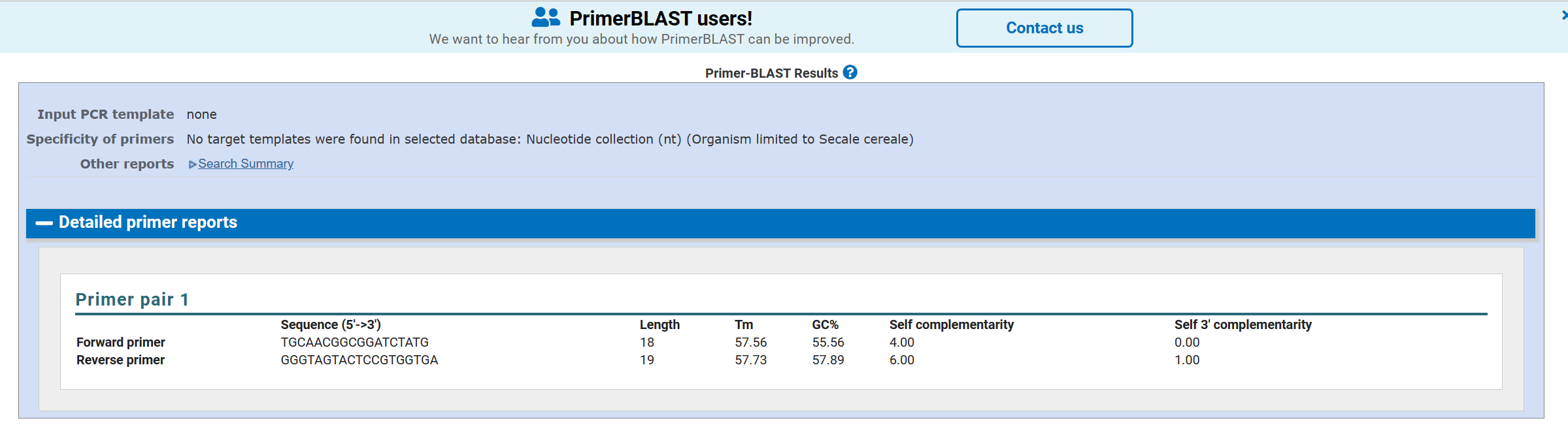
"Input PCR template none"的错误提示意味着在选择的数据库中没有找到目标模板。"Specificity of primers No target templates were found in selected database"的错误提示表示在选择的数据库中未找到目标模板。
这可能有以下几个可能的原因:
1. 数据库中没有包含目标模板:您选择的数据库可能不包含您所期望的目标模板。确保选择适当的数据库,其中包含您感兴趣的生物物种或相关序列。
2. 引物特异性问题:引物的特异性可能存在问题。确认所使用的引物序列是否与预期的目标序列完全匹配,并排除引物序列错误或非特异性扩增的可能性。您可以再次检查引物序列并进行引物特异性分析,以确认其特异性。
3. 引物设计问题:引物的设计可能存在问题,导致无法在选择的数据库中找到目标模板。确保引物的设计合理,并使用合适的引物设计工具进行设计,以最大程度地提高特异性和特异性。
解决这些问题的方法包括:
1. 检查数据库:确认所选择的数据库是否包含您感兴趣的目标模板。如果数据库不包含您想要的目标模板,您可能需要选择其他数据库或扩展搜索范围。
2. 重新评估引物:重新检查引物的特异性和设计。确保引物序列的准确性,并使用引物特异性分析工具进行验证。如果发现问题,考虑重新设计引物或选择其他合适的引物。
3. 扩展搜索范围:如果合适,尝试在其他数据库中搜索目标模板,或者使用更广泛的数据库进行搜索,以增加找到目标模板的可能性。
总之,通过仔细检查引物设计和验证特异性,并选择合适的数据库,您可以解决"Input PCR template none"和"Specificity of primers No target templates were found in selected database"的问题。
高山云初
引物的特异性在选定的数据库中没有发现目标模板
1.首先你根据什么物种设计的序列,检索选项中填写的是否是该物种?
2.pcr产物长度是多少?默认值能否cover全部序列?
相关产品推荐
相关问答