浅析染色质免疫沉淀(ChIP)技术在 DNA 与蛋白质相互作用研究中的重要性
丁香实验
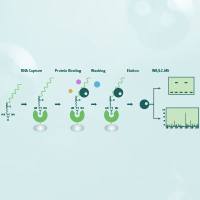
染色质免疫沉淀(ChIP)是研究蛋白质-DNA 相互作用的一项强大技术,广泛用于多个领域的染色质相关蛋白的研究(如组蛋白及其异构体,转录因子等),特别适用于已知启动子序列或整个基因位点的组蛋白修饰分析研究。这项技术采用特定抗体来富集存在组蛋白修饰或者转录调控的 DNA 片段,通过多种下游检测技术(定量 PCR ,芯片,测序等)来检测此富集片段的 DNA 序列。
ChIP 技术自诞生之后,已成功的应用于人或动物细胞和组织 [1] 、植物组织 [2] 、酵母 [3] 以及细菌、质粒 [4] 。由于在信号转导和表观遗传研究中的突出作用,ChIP 在肿瘤 [5-7] 、神经科学 [8-10] 、植物发育 [11-13] 等领域中应用非常广泛,同时有关细胞凋亡 [14] 、雌激素信号转导 [15] 、胰岛素抵抗 [16] 、组织发育 [1] 的文献中也用到ChIP。
目前,最常见的有以下两种 ChIP 实验技术:
- nChIP:用来研究 DNA 及高结合力蛋白,采用微球菌核酸酶(micrococcal nuclease)消化染色质,然后进行片段富集及后续分析,适用于组蛋白及其异构体,例如 [17-19];
- xChIP:用来研究DNA及低结合力蛋白,采用甲醛或紫外线进行 DNA 和蛋白交联,超声波片段化染色质,然后进行片段富集及后续分析,适用于多数非组蛋白的蛋白,例如 [9, 15, 20]。
X-ChIP 试验的一般过程
以上两种方法在分离 DNA-蛋白质复合物之后,对 DNA 进行 PCR 扩增,验证目标序列的存在。除验证实验外,ChIP DNA也可以进行测序分析,这种方法被称为 ChIP-seq [22] ;也可做芯片分析,这种方法被称为 ChIP on CHIP 或 ChIP-CHIP [23] 。这两种方法都可用于分析感兴趣蛋白结合的未知序列,而不需要知道目标序列的详细信息,因此可以进行探索性的研究。当需要对 DNA 结合的蛋白复合物(两个或两个以上蛋白共同结合在 DNA 上)进行研究时,可以采用 reChIP 技术对DNA 蛋白复合物进行再次富集,从而分析两种蛋白同时结合的 DNA 片段,例如转录调控因子及其受体复合物 [24] 。
相比其他 DNA 与蛋白相互作用的研究工具,ChIP 的突出优势在于通过检测蛋白(in vivo)与 DNA 的物理结合,研究体内真实情况下的染色质结构,因此得出确凿的诱导或阻碍转录的证据,这对构架信号调控网络至关重要。同时,通过此工具也可以区分转录调控的直接作用和非直接作用 [21] ,从而使研究者对转录调控过程有更精确而深刻的认识。
ChIP 试验的结果示例
基于上述的技术优势,ChIP 首先被用来研究分子层面上的调控机制,区别于其他研究转录调控相关性的实验手段。这类分子机制研究多数是关注于 DNA 结合位点是否为启动子区或其他转录调控位点。例如,癌基因 myc 上游调控机制的研究非常之多,然而大多停留于假说阶段。Sotelo 等人 [20] 于近期首先发现转录因子 Tcf-4 和 β-catenin 共同上调 c-Myc 基因调控因子 enhance E 的表达,然而 luciferase reporter 实验结果仅能证明 β-catenin 和转录因子 Tcf-4与enhance E 的相关性,无法提供证据表明这两种细胞因子如何作用于 enhance E 基因上。因此,他们采用了 ChIP 技术,证明前列腺癌 LnCAP 细胞中是 Tcf-4 而不是 β-catenin 直接结合到 enhance E 基因上,这就表明 Tcf-4 直接参与到 enhance E 的调控中。此结果与 Hatzis 等人 [25] 对直肠癌细胞系 LS174T 做的 ChIP-CHIP 测得的结果一致,证实了一直以来研究者关于 myc 存在远端增强子的猜测。类似地,有研究表明 β-catenin 与 FGF2 相互作用,影响神经干细胞增殖或分化,然而具体如何协同调控增殖或分化进程尚未知,直到 Israsena 等人[10]用 ChIP 试验证明了 β-catenin 仅在 FGF2 存在时与 neurogenin 启动子直接结合,从而调控 FGF2 下游信号通路。这就解释了 FGF2 在该信号通路中的主导作用。另一种常见的分子机制研究则是关注于 DNA 结合位点下游的基因,用此类基因的表达活化或抑制来解释表型上的差异。例如,Seo 等人 [26] 采用 ChIP 技术分析了 NeuroD 结合位点的下游基因,发现了多个转录调控因子,因此得出结论,NeuroD 则位于该神经发育调控网络的重要位置。
其次,ChIP 也往往被用来验证其他 DNA-蛋白质相互作用研究方法所得的结果。由于其他研究方法并非在体内(in vivo)实现或者尚不能得出确切结论,因此需要 ChIP 试验进行有力的支持。有研究 [6] 通过 WB, MSP,EMSA 等技术发现乳腺癌细胞的高度恶性与抑制肿瘤转移相关的 TFPI-2 基因启动子区过度甲基化有关,之后用 ChIP 试验则证实了该区域高度甲基化后确实转录水平因此受到影响,为上述的结论提供更充分的论据。类似的,Peng 等人 [13] 用 EMSA 技术得出水稻转录因子 OsLFL1 结合于 Ehd1 基因的启动子区的初步结论后,采用 ChIP 在体内重复出了这个结果,因此最终能确定该转录因子的结合位置。
ChIP 是染色质结构改变,特别是组蛋白修饰后,研究全基因组活化或抑制作用的少数工具之一。ChIP 可以针对乙酰化或者甲基化的组蛋白,富集该区域的 DNA ,因此可以进行基因组的多样性分析。2008 年,Xiang 等人 [7] 发现硒(Se)处理与前列腺癌细胞的 DNA 甲基化水平降低、组蛋白乙酰化水平升高、GSTP1(前列腺癌相关蛋白)活化有关。试验中,他们用 ChIP 技术富集了乙酰化 H3-k9 和甲基化 H3-K9,对分离获得的 DNA 进行多个基因的 PCR 扩增。结果显示 GSTP1 基因在处理组和非处理组之间有显著差异,这表明 Se 处理的癌细胞 GSTP1 启动子相关的 H3-K9 乙酰化水平提高、 H3-K3 甲基化水平降低,预示 Se 可能具有调节染色质表观结构的功能。
正因为 ChIP 技术对于表观遗传学及信号转导研究是如此之重要,默克密理博特别为该领域的科学家们专门开发了的 ChIP 解决方案,其中完备的 ChIP 试剂盒免去提前准备各种试剂(包括鲑鱼精子 DNA 包被的琼脂糖珠子、Protein A/G 等)的麻烦,专用的 ChIP 抗体节省了抗体测试的时间和精力,优化的 protocol 大大降低了 ChIP 实验的难度、减少摸索实验条件的时间。经过不断的改良及完善,新一代的 EZ-ChIP 能够提供阴性、阳性对照和 DNA 纯化柱,使 ChIP 试验更为高效可靠,大量经典文献的引用即是其品质的保证 [27-29] 。另外,对于应用越来越多的 ChIP on CHIP,默克密理博还提供 EZ-Magna ChIP 试剂盒,磁珠分离技术让 ChIP 跨入高通量时代,为全基因组的探索插上翱翔的翅膀。
参考文献:
[1] Zhang, Y., et al., GATA and Nkx factors synergistically regulate tissue-specific gene expression and development in vivo. Development,2007. 134(1): p. 189-98.
[2] Rossi, V., et al., A maize histone deacetylase and retinoblastoma-related protein physically interact and cooperate in repressing gene transcription. Plant Mol Biol, 2003. 51(3): p. 401-13.
[3] Han, Q., et al., Gcn5- and Elp3-induced histone H3 acetylation regulates hsp70 gene transcription in yeast. Biochem J, 2008. 409(3): p. 779-88.
[4] Grainger, D.C., et al., Genomic studies with Escherichia coli MelR protein: applications of chromatin immunoprecipitation and microarrays. J Bacteriol, 2004. 186(20): p. 6938-43.
[5] Wu, C.H., et al., Combined analysis of murine and human microarrays and ChIP analysis reveals genes associated with the ability of MYC to maintain tumorigenesis. PLoS Genet, 2008. 4(6): p. e1000090.
[6] Guo, H., et al., Tissue factor pathway inhibitor-2 was repressed by CpG hypermethylation through inhibition of KLF6 binding in highly invasive breast cancer cells. BMC Mol Biol, 2007. 8: p. 110.
[7] Xiang, N., et al., Selenite reactivates silenced genes by modifying DNA methylation and histones in prostate cancer cells. Carcinogenesis, 2008. 29(11): p. 2175-81.
[8] Sun, G., et al., Orphan nuclear receptor TLX recruits histone deacetylases to repress transcription and regulate neural stem cell proliferation. Proc Natl Acad Sci U S A, 2007. 104(39): p. 15282-7.
[9] Zhao, C., et al., A feedback regulatory loop involving microRNA-9 and nuclear receptor TLX in neural stem cell fate determination. Nat Struct Mol Biol, 2009. 16(4): p. 365-71.
[10] Israsena, N., et al., The presence of FGF2 signaling determines whether beta-catenin exerts effects on proliferation or neuronal differentiation of neural stem cells. Dev Biol, 2004. 268(1): p. 220-31.
[11] Kwon, C.S., C. Chen, and D. Wagner, WUSCHEL is a primary target for transcriptional regulation by SPLAYED in dynamic control of stem cell fate in Arabidopsis. Genes Dev, 2005. 19(8): p. 992-1003.
[12] Rossi, V., et al., Maize histone deacetylase hda101 is involved in plant development, gene transcription, and sequence-specific modulation of histone modification of genes and repeats. Plant Cell, 2007. 19(4): p. 1145-62.
[13] Peng, L.T., et al., Ectopic expression of OsLFL1 in rice represses Ehd1 by binding on its promoter. Biochem Biophys Res Commun, 2007. 360(1): p. 251-6.
[14] Omata, K., et al., Identification and characterization of the human inhibitor of caspase-activated DNase gene promoter. Apoptosis, 2008. 13(7): p. 929-37.
[15] Metivier, R., et al., Estrogen receptor-alpha directs ordered, cyclical, and combinatorial recruitment of cofactors on a natural target promoter. Cell, 2003. 115(6): p. 751-63.
[16] Shi, H., et al., TLR4 links innate immunity and fatty acid-induced insulin resistance. J Clin Invest, 2006. 116(11): p. 3015-25.
[17] Tsukuda, T., et al., Chromatin remodelling at a DNA double-strand break site in Saccharomyces cerevisiae. Nature, 2005. 438(7066): p. 379-83.
[18] Bernstein, B.E., et al., A bivalent chromatin structure marks key developmental genes in embryonic stem cells. Cell, 2006. 125(2): p. 315-26.
[19] Topp, C.N., C.X. Zhong, and R.K. Dawe, Centromere-encoded RNAs are integral components of the maize kinetochore. Proc Natl Acad Sci U S A, 2004. 101(45): p. 15986-91.
[20] Sotelo, J., et al., Long-range enhancers on 8q24 regulate c-Myc. Proc Natl Acad Sci U S A, 2010. 107(7): p. 3001-5.
[21] Weinmann, A.S., Novel ChIP-based strategies to uncover transcription factor target genes in the immune system. Nat Rev Immunol, 2004. 4(5): p. 381-6.
[22] Barski, A. and K. Zhao, Genomic location analysis by ChIP-Seq. J Cell Biochem, 2009. 107(1): p. 11-8.
[23] Kim, T.H., L.O. Barrera, and B. Ren, ChIP-chip for genome-wide analysis of protein binding in mammalian cells. Curr Protoc Mol Biol, 2007. Chapter 21: p. Unit 21 13.
[24] Metzger, E., et al., LSD1 demethylates repressive histone marks to promote androgen-receptor-dependent transcription. Nature, 2005. 437(7057): p. 436-9.
[25] Hatzis, P., et al., Genome-wide pattern of TCF7L2/TCF4 chromatin occupancy in colorectal cancer cells. Mol Cell Biol, 2008. 28(8): p. 2732-44.
[26] Seo, S., et al., Neurogenin and NeuroD direct transcriptional targets and their regulatory enhancers. EMBO J, 2007. 26(24): p. 5093-108.
[27] Loffler, D., et al., Interleukin-6 dependent survival of multiple myeloma cells involves the Stat3-mediated induction of microRNA-21 through a highly conserved enhancer. Blood, 2007. 110(4): p. 1330-3.
[28] Kim, D.H., et al., MicroRNA-directed transcriptional gene silencing in mammalian cells. Proc Natl Acad Sci U S A, 2008. 105(42): p. 16230-5.
[29] Borchert, G.M., W. Lanier, and B.L. Davidson, RNA polymerase III transcribes human microRNAs. Nat Struct Mol Biol, 2006. 13(12): p. 1097-101.