求多个基因变体如何设计引物
相守RGXS
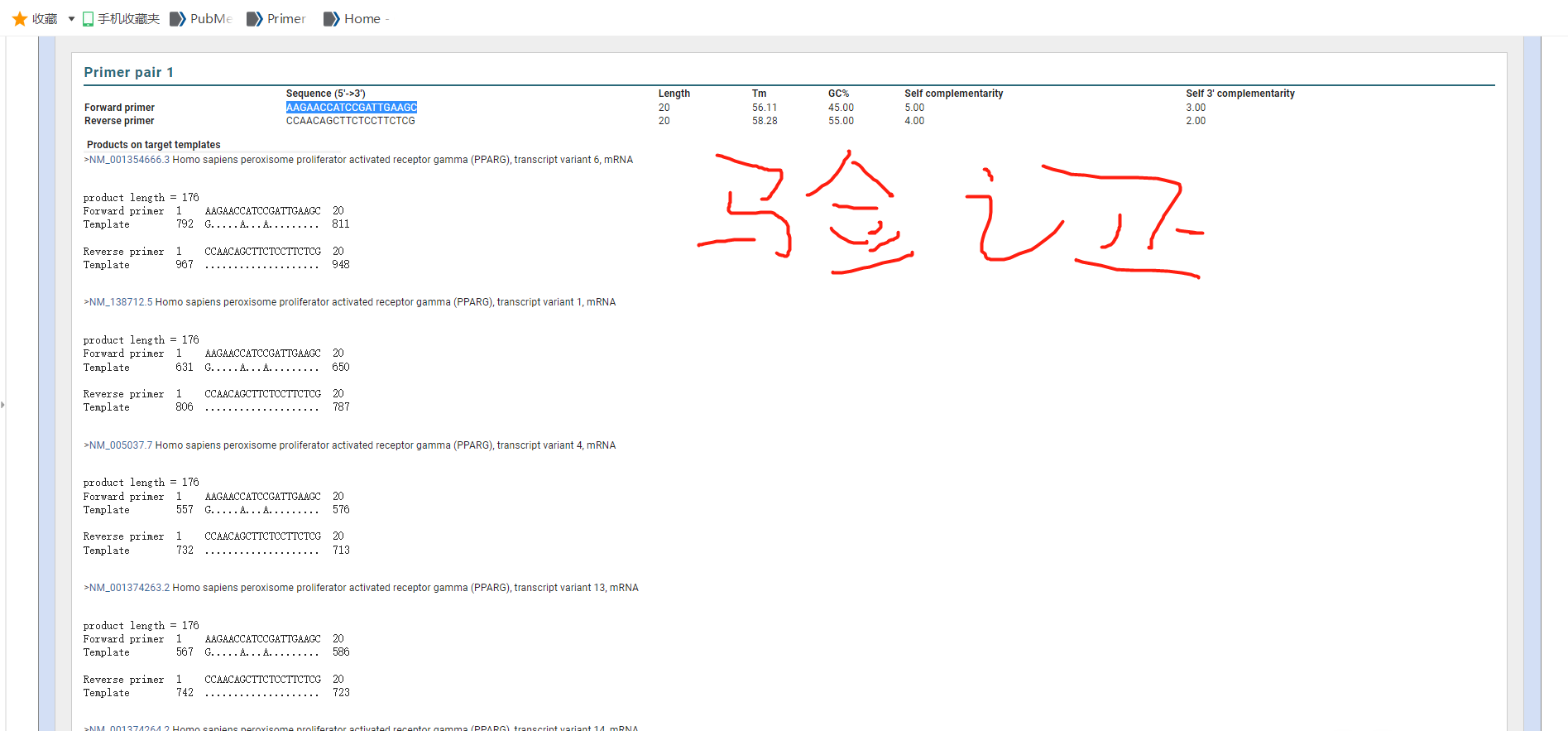
如图,想设计一个rat的PPARγ引物,第一:NCBI里面把这个的基因分成了2大块(见2张截图),这有啥区别吗。第二,据我所知他有γ1、2、3三种蛋白亚型,所以可能变体比较多,但是仔细看每种基因对应的蛋白并不是简单的γ1、2、3,而是命名为,举个例子:isoform 2或1这样子,感觉有点混乱;第三:我拿人家文章里面的序列验证了(AAGAACCATCCGATTGAAGC和 CCAACAGCTTCTCCTTCTCG),出来的结果对应多种变体。。。额,更乱了感觉(见第三张截图)总之,求大佬解惑或者给个学习的链接也可,想搞搞清楚,感谢!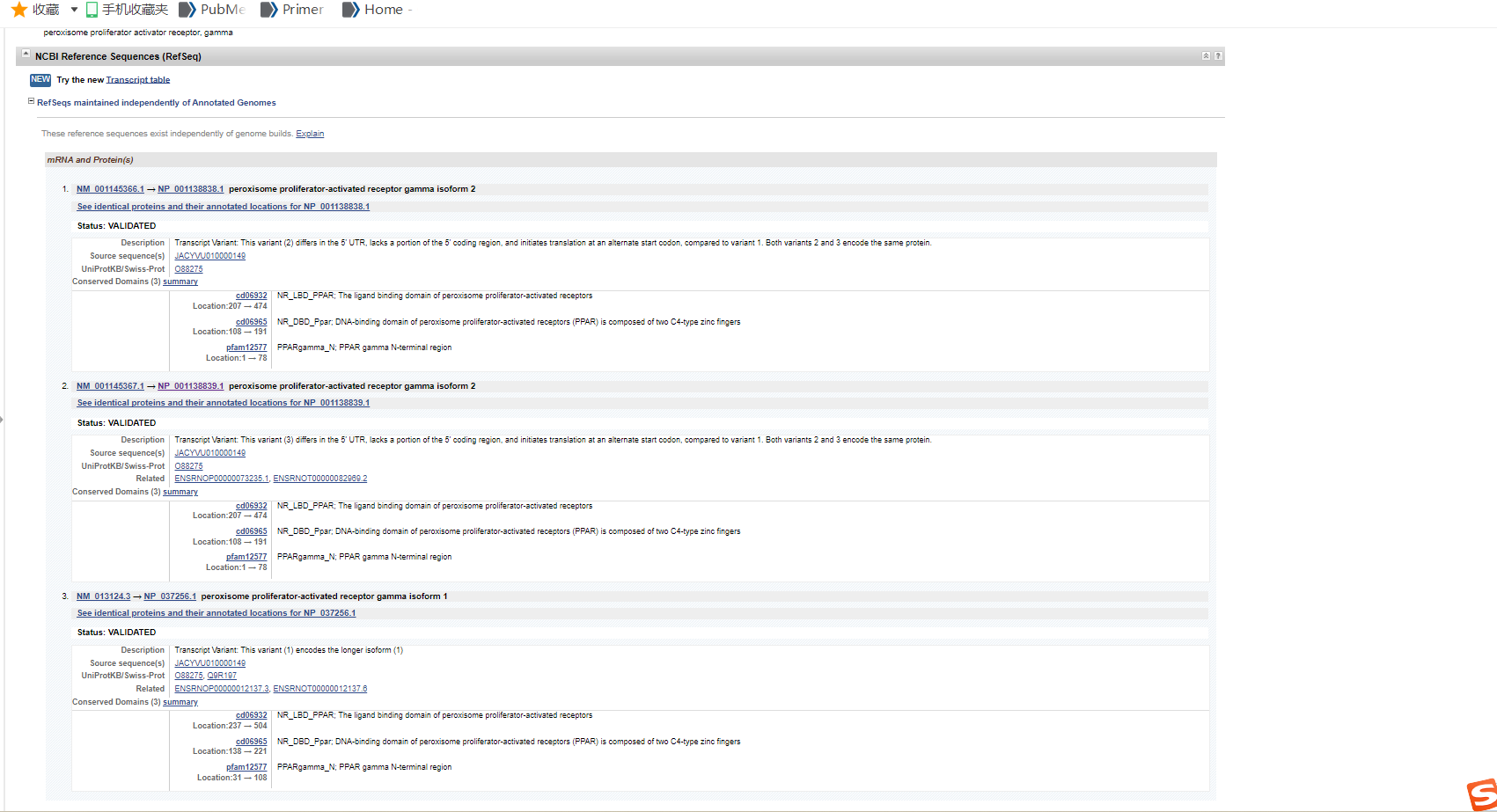

6 个回答
小芙fu
最好设计三对引物,一对为同源序列,另两对分别针对特异序列,这样分析出来的结果更丰富。
如果有一个Transcript variant不能设计特异的序列,就设计另一个Transcript variant的特异性序列和两个Transcript variant的同源序列即可。
我认为,分别测定两具Transcript variant的表达水平比较好,因为两个Transcript variant的干扰效果可能不一样。
dxyc42u
对于有多个变体的基因,看具体哪个变体有功能就设计这个的
相守RGXS
请问怎么查看哪个基因有功能呢,会存在功能不一样的情况么?
sswei
如果仅仅检测这个基因的表达量,可以将引物设计在这多种变体的保守区域内,就是所有变体都包含的区域。如果明确要检测哪一个变体,就设计在这个变体独有的区域内。
2引物blast后的产物有多种
应该是在NCBI上进行的primer blast。只要看到引物扩增出来的都是要的目的基因就好了。在NCBI上有很多标记,一般认为NM的是NCBI上认可的reference的序列,NX是用户上传还未认证的序列,忽略也是可以的。predicted是指用户预测的序列,建议还是用NCBI认证的序列。
于小鱼鱼1998
你列举的引物如果对应单一基因可以得到特异性序列,所以可以分别设计引物,然后再进行序列对比
土井挞克树
多个基因变体设计引物要分别设计,像你是共同设计就会混乱,应该分开设计
huarenqiang5
你如果仅仅检测这个基因的表达量,你可以将引物设计在这多种变体的保守区域内,就是所有变体都包含的区域。如果你明确要检测哪一个变体,就设计在这个变体独有的区域内。
相守RGXS
好的 非常感谢,明白了。请问有操作的教程不,我只会在pubmed的基因里面找到具体序列后pick primarys,哈哈。。。
相关产品推荐
相关问答