文献速递|低丰度基因和 IncRNAs 表达的动态示踪
EVIDENT
CRISPR 是一种基因编辑技术,由 Cas 核酸酶和 sgRNA 组成,可以对生物的 DNA 序列进行定向切割、修改,被 Nature 杂志列为 2013 年年度十大科技进展之一。
而将 CRISPR 与荧光成像结合,就可以建立一个活体成像系统,实现对特定基因位点标记成像,并进一步在基因的生物学功能研究中发挥着重要的作用。

在活细胞中实时监测内源基因活性对于研究基因的生物学功能并调控其表达水平至关重要。近年来越来越多证据表明,低表达基因虽然表达量较低,但是在各种生物学过程中往往起着重要的调控作用;
此外,基因组中编码蛋白质的基因只有 1%-2%,大部分为非编码 RNA,其中长链非编码 RNA(Long non-coding RNAs)具有非常重要的调控功能(图 1),多达 78% 的 lncRNAs 表达具有组织特异性 [1]。
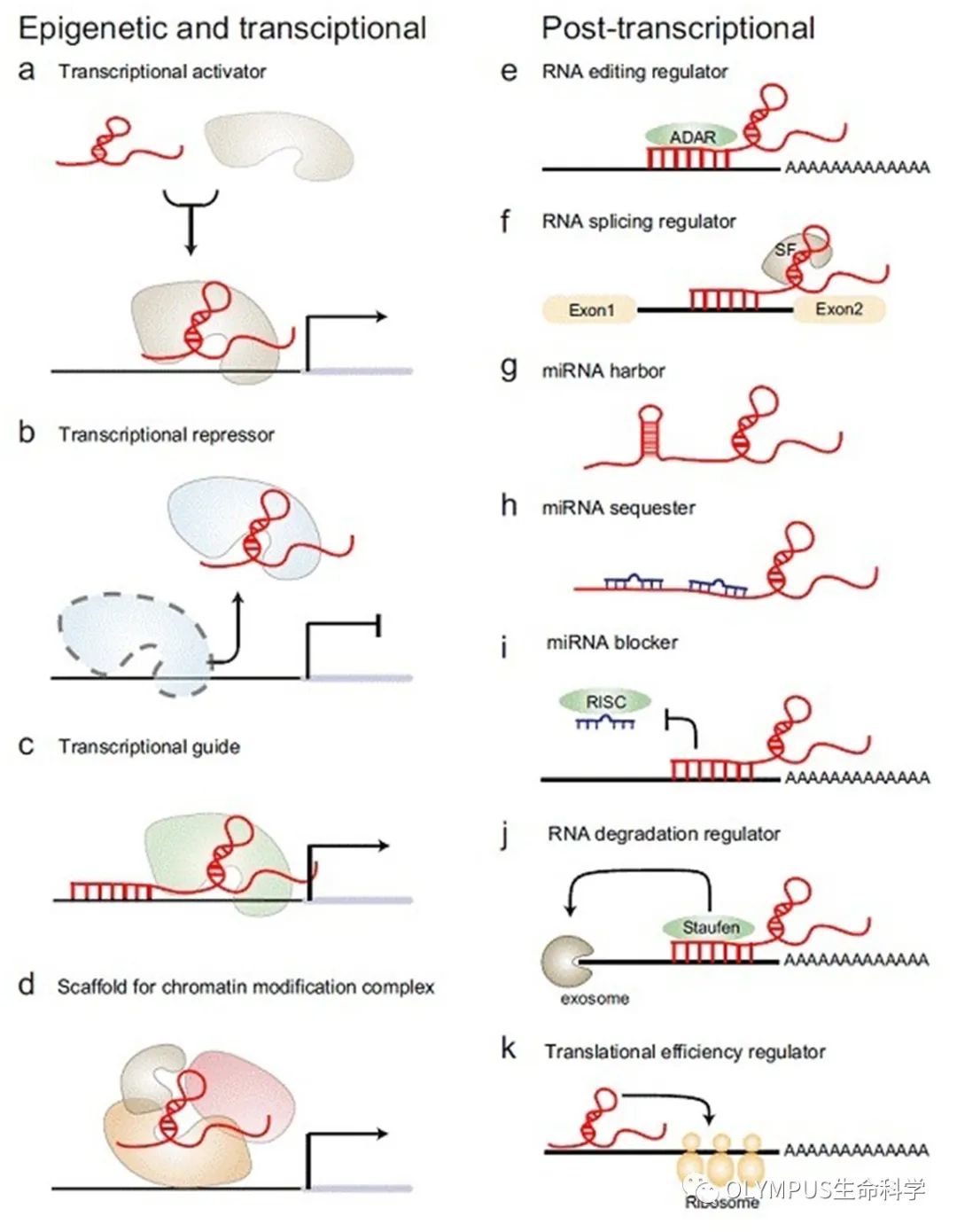
图 1:lncRNA 功能示意图
近年来越来越多证据表明,lncRNAs 不仅参与各种生物学过程和通路,而且与各种疾病的发生发展紧密相关。但是受现有标记技术的限制,对低表达基因和 lncRNAs 的功能注释极具挑战性。

近日,《Nature Cell Biology》期刊在线发表了题为《通过内源性启动子驱动的 sgRNA 监测低丰度转录本和 lncRNAs 的表达》的研究论文,该研究通过内源基因的启动子驱动 sgRNA(single-guide RNAs)表达,结合 SPH-OminiCMV(CRISPR-activator Suntag-P65-HSF1 and OminiCMV-mCherry)荧光报告系统,成功实现低丰度基因和 lncRNAs(Long non-coding RNAs) 基因表达的动态示踪。
该研究建立了一种通用的内源基因转录门控系统,为活细胞中实时标记低表达基因和 lncRNAs,研究其生物学功能提供了有效工具。
Part1 监测内源基因表达的技术概述
监测内源基因活性的常规方法是将荧光蛋白精确插入蛋白编码框中 [3],但是,这种方法并不适用于非编码基因和低丰度转录的基因。
sgRNA,类似于一个 GPS 可以将 Cas 核酸酶直接导向目标核酸位点,具有高特异性、高效率和多功能性 [4]。
尽管各种诱导性 sgRNA 已经被开发用来接收活细胞中的内源信号,但这些方法只能用于某些功能明确的小 RNA 的反应 [5-7]。
目前在活细胞里面标记内源基因表达的方法,都具有一定的局限性,而且,对于低表达的基因以及 lncRNA,目前缺乏很好的活细胞内的标记技术。
建立适用范围更广,能增强内源信号,并且信噪比更低的新型活细胞标记技术,是该研究领域急需解决的问题。
Part2 建立并优化 SPH-OminiCMV-Ents 荧光报告系统
我们开发了一种广谱的内源性转录门控开关 Ents(Endogenous Transcription-Gated Switch),利用内源性启动子表达 sgRNA 前体,之后 sgRNA 前体上的 tRNA 序列可以被内源的自剪切机制识别并且切割,进而释放出能够执行功能的 sgRNA,当 Ents 与高度敏感的 CRISPR 激活相关的报告系统 SPH-OminiCMV 结合,可以监测到内源基因的表达(图 2a)。
值得注意的是,相比 P2A-mcherry 策略,SPH-OminiCMV-Ents 能够放大内源信号,从而实现低丰度转录本表达的可视化;此外,该系统理论上可以处理任何转录本的信息,所以也适用于监测非编码 RNA 的转录。
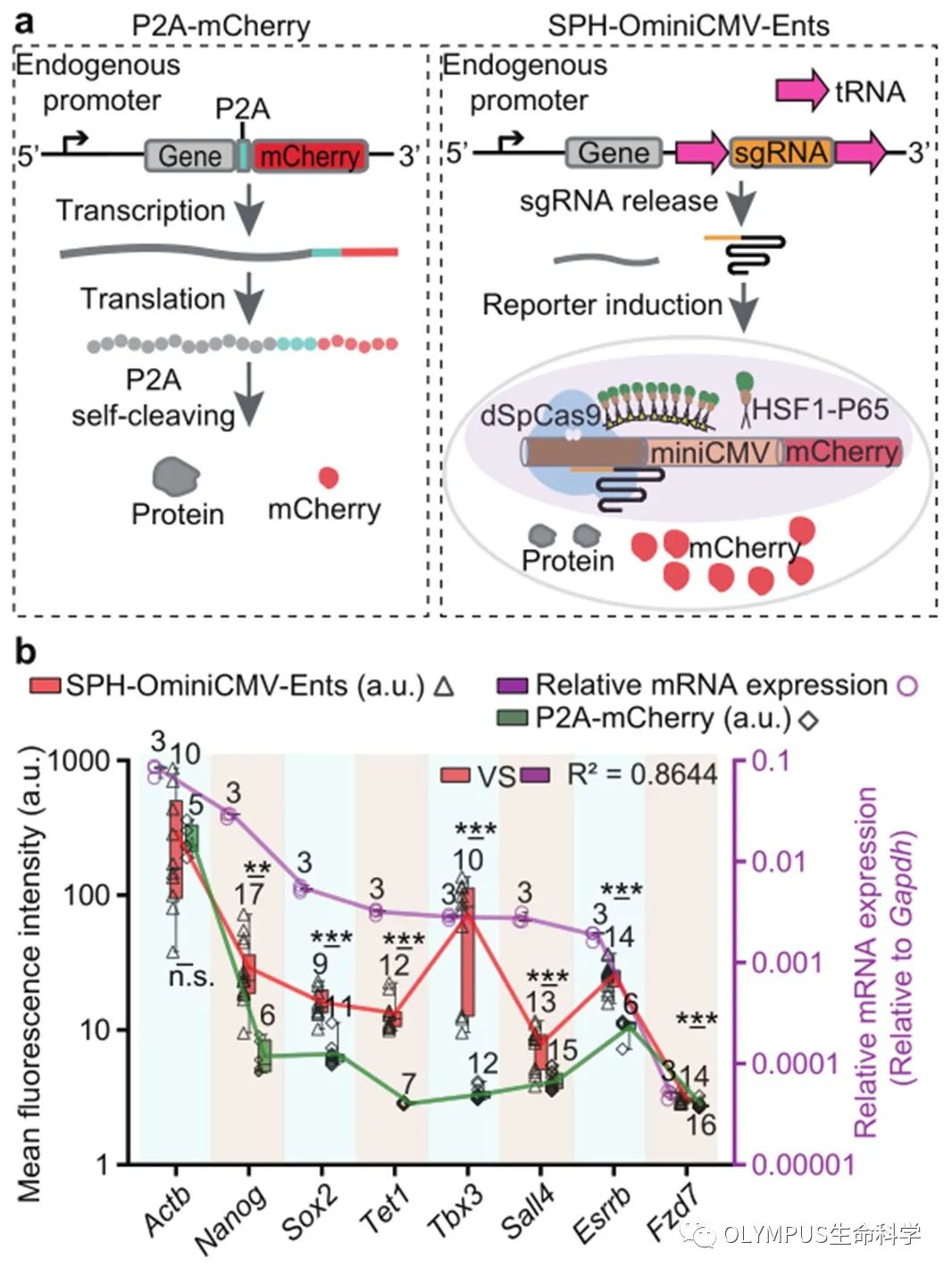
图 2:(a)示意图显示 P2A-mcherry 和 SPH-OminiCMV-Ents 两种标记基因的策略。
(b)在 mESC 细胞中用 SPH-OminiCMV-Ents 策略(红色三角形)和 P2A- mCherry 策略(绿色菱形)标记不同的基因,统计其 mCherry 荧光信号强度以及用 qPCR 分析这些基因的表达水平(紫色圆圈,紫色 y 轴)。
为探讨增加 sgRNA 拷贝数是否会进一步增强荧光蛋白的产生,团队构建了一个 sgRNA 前体,包含六到八个串联的 sgRNA 拷贝,每个 sgRNA 的两侧分布着 tRNA 序列(图 3a)。研究表明,插入 sgRNA 阵列可以对低丰度基因实现更好的监测,比如 lncRNA Tug1,当仅用一个拷贝的 sgRNA 的时候不能监测到其表达,但使用 sgRNA 阵列则可以(图 3b,c)。
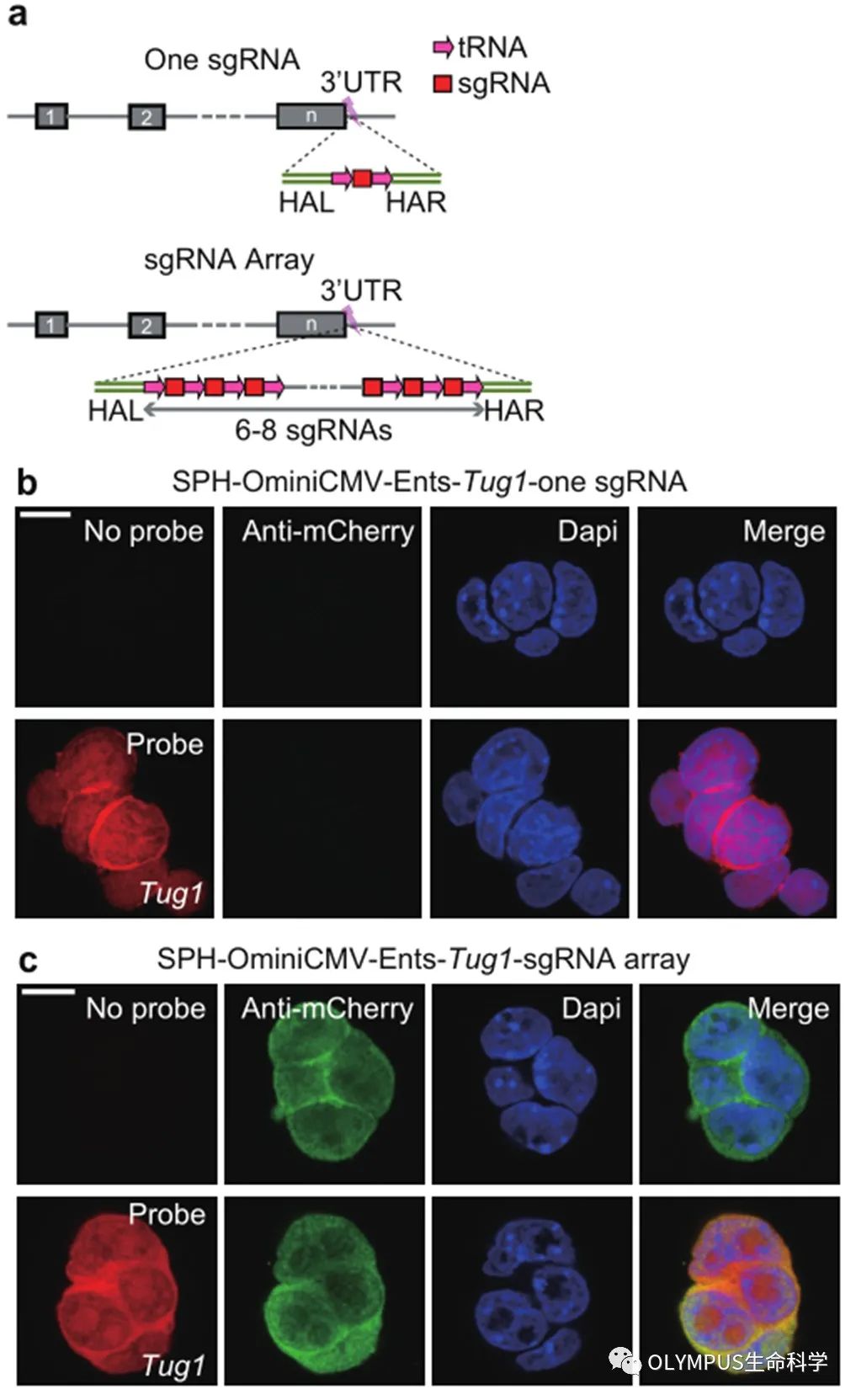
图 3:(a)示意图显示插入一个 sgRNA 或一个 sgRNA 阵列的标记策略。
(b,c)Fish 图像显示对 lncRNA Tug1 在 3'UTR 中插入一个 sgRNA 或一个 sgRNA 阵列后,细胞系中 mCherry 报告荧光信号的表达情况。
Part3 通过显微镜观察到的荧光信号反映目标基因转录水平
为进一步探讨荧光报告系统的特性,我们建立了一个可控的 Tet-on 系统,该系统可以通过调节 Dox 的浓度,实现 EGFP 基因不同程度的表达量,进而用来研究报告系统的荧光信号强度是否可以很好地反映目标基因的表达水平。
研究发现,报告系统的 mCherry 表达量和 EGFP 基因的表达量有很强的相关性,并且进一步探索了此报告系统和目标基因表达在转录本水平和蛋白翻译水平存在的时间差(图 4)。
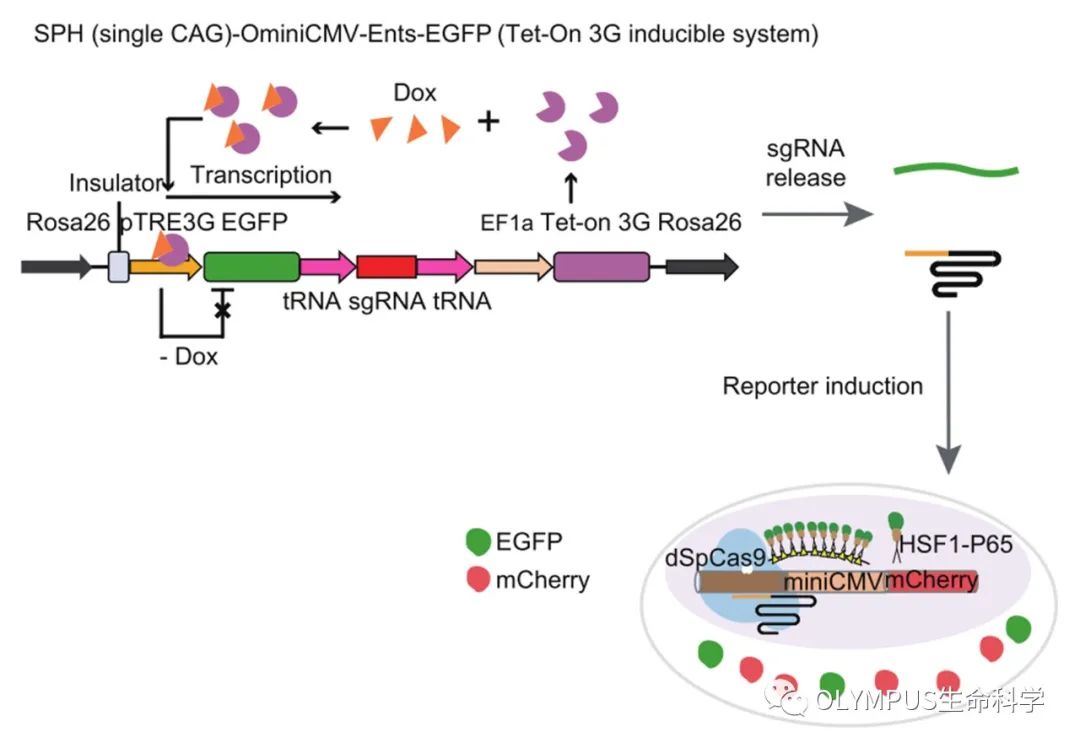
图 4 : 可控的 Tet-on 系统示意图,Dox 诱导 EGFP-sgRNA 前体转录,成熟的 sgRNA 释放出来激活 mcherry 报告系统。
Part4 SPH-OminiCMV-Ents 荧光报告系统的应用
我们将 Ents 与 SPH-OminiCMV 结合使用,在小鼠胚胎干细胞水平上实现了对低丰度基因和 lncRNA 的可视化监测及其动态变化的监测。
该研究为在活细胞中研究基因元件的功能开辟了新途径,在动物个体水平可用于描绘基因表达的时空图谱和标记、鉴定特定的细胞类群,同时可用于研究天使综合症等疾病中 lncRNA 的表达模式,构建 lncRNA 体内表达的完整图谱。
Part5 对显微镜的要求,样品制备技巧
首先,该研究中的活细胞样品需要继续培养,因此要求尽可能简化显微镜成像过程,提高成像速度。
Evident 的 FV3000 软件简化了从采集到分析的整个共聚焦成像及分析工作流程,将多位点延时(MATL)模块与多孔板导航器模块配合使用,可以设置同一孔板甚至同一孔内多点拍摄,无需手动调整细胞培养板,非常方便,可以尽量减少细胞在培养箱外待的时间。
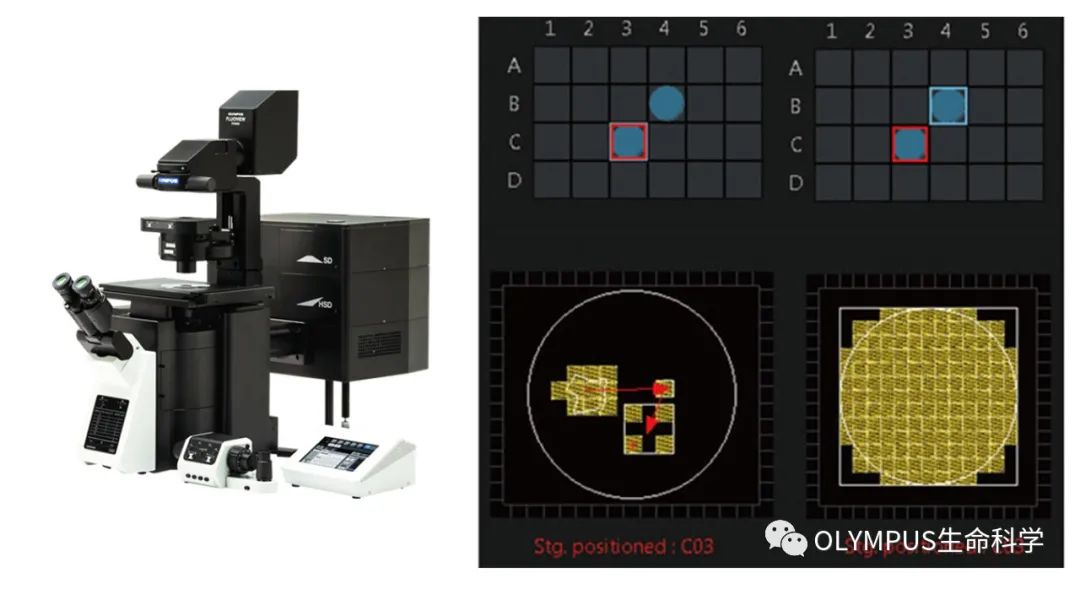
此外,本研究中使用的聚苯乙烯的细胞培养板与常规的共聚焦玻底皿不同,这种差异使得在成像过程中不但工作距离有所改变,还需要额外的光学校准。
Evident 全新的 X Line 物镜,依靠超薄透镜打磨技术和超薄透镜组装技术,使图像平场性、数值孔径和色差校正范围三大核心参数同步得到提升,还提供盖玻片厚度、工作距离、浸油折射率等多种校正,为实验提供更专业可靠的支持。