干货 | 蛋白质-DNA 互作研究那些你不知道的技术更迭!
诺唯赞
蛋白质与 DNA 互作是基因转录调控的关键,也是启动基因转录的前提。蛋白质与 DNA 互作主要包括组蛋白、转录因子、DNA 甲基化酶和染色质重塑复合物等。为了研究蛋白质-DNA 互作,科学家发明了很多方法:凝胶阻滞、DNaseⅠ足迹实验、甲基化干扰、体内足迹、酵母杂交、ChIP-Seq 等。其中 ChIP-Seq 可以真实、完整地反映结合在 DNA 序列上的靶蛋白,是目前研究蛋白质-DNA 互作的经典方法。但该技术需要大量细胞,且步骤繁琐,耗时较长,因此,研究者不断在寻找新的替代方法。
2019 年 4 月 Thomas G Fazzio 教授等发表在 Cell 期刊的 uliCUT&RUN 技术,将蛋白质-DNA 互作研究升级到单细胞水平。近几个月来,又相继有文章报道了 CUT&Tag 等技术,进一步优化了蛋白质-DNA 互作研究方法,本文将对系列方法一并总结介绍。
1.蛋白质-DNA 互作新技术发展历程
Orlando等研发出ChIP[1],主要用于研究转录因子与 DNA 结合位点的序列信息。
Dominic Schmidt 等将 ChIP 与高通量测序技术结合,发明了 ChIP-seq ,能够在全基因组范围内检测与蛋白相互作用的 DNA 区域。
Henikoff 等发布 CUT&RUN 技术,使用微球菌核酸酶(micrococcal nuclease, MNase)进行染色质切割替代 ChIP-seq中的超声打断,使得蛋白-DNA 互作研究从 ChIP-seq 所需的 104 级别降到 100-1000 个细胞。
Thomas G Fazzio 教授等发布 uliCUT&RUN ,将 CUT&RUN 技术升级到单细胞水平,并应用于早期胚胎研究。
Henikoff 等发明 CUT&Tag ,使用带有接头的 Tn5 转座酶替代 MNase ,简化实验操作,将细胞量从 104 级别降到 60 个细胞甚至单细胞。
2.蛋白质-DNA 互作技术概述
✦染色质免疫共沉淀(ChIP):
ChIP 是全基因组范围内检测蛋白-DNA 互作的标准方法,该技术由 Orlando 等于 1997 年创立[1]。
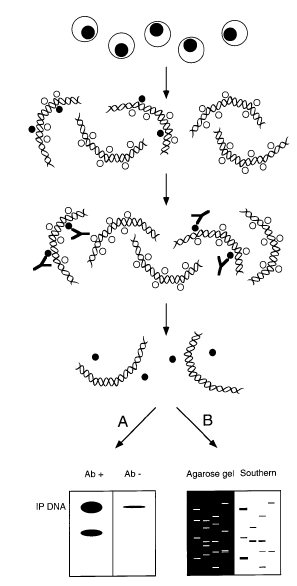
图 1:ChIP 基本原理图[1]
✦ ChIP 原理:
将染色质和与之相互作用的转录因子和组蛋白通过甲醛等物质交联起来,然后通过超声将染色质打碎成小片段,加入针对特定转录因子或特殊修饰的组蛋白抗体,通过 Protein A/Protein G 微球或磁珠将抗体-转录因子-染色质复合物拖下来,通过 PCR 或测序的方法检测与目的蛋白相结合的 DNA 序列,进而研究这些转录因子在细胞发育或者生长中的作用位点。
✦ ChIP-seq:
ChIP-seq 将 ChIP 技术与二代测序相结合,将 ChIP 下来的 DNA 进行二代测序文库构建,能够获取全基因组范围内组蛋白 DNA 及染色质修饰等 DNA 区段信息。
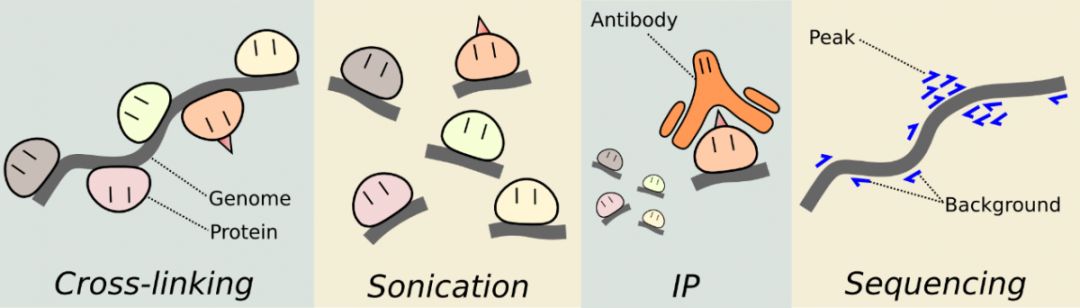
图 2:ChIP-seq 流程
✦ ChIP-seq 原理:
将通过 ChIP 特异性收集到的与目的蛋白结合的 DNA 片段进行纯化与文库构建,ChIP-seq 继承了 ChIP 的技术难点,需要先用甲醛将与 DNA 互作的蛋白固定,这个过程会造成一些非相关蛋白交联,形成假阳性。一些作用力小的转录因子或者由于甲醛交联不充分,在超声破碎时会造成假阴性。为了消除背景噪音,则需加大细胞投入量。ChIP 以及 ChIP-seq 无法捕获少量细胞中发生的关键表观基因组学过程, 包括影响胚胎发育,发育疾病,干细胞的过程分化和某些癌症的发生。
✦ CUT&RUN (Cleavage Under Targets and Release Using Nuclease ):
Henikoff 实验室于 2017 年 1 月 16 日发表在 elife 上关于 CUT&RUN 的文章,使得蛋白-DNA 互作研究从 ChIP-seq 所需的 104 级别降到 100-1,000 个细胞[2]。

图 3:CUT&RUN 文章 [2]
✦ CUT&RUN 原理:
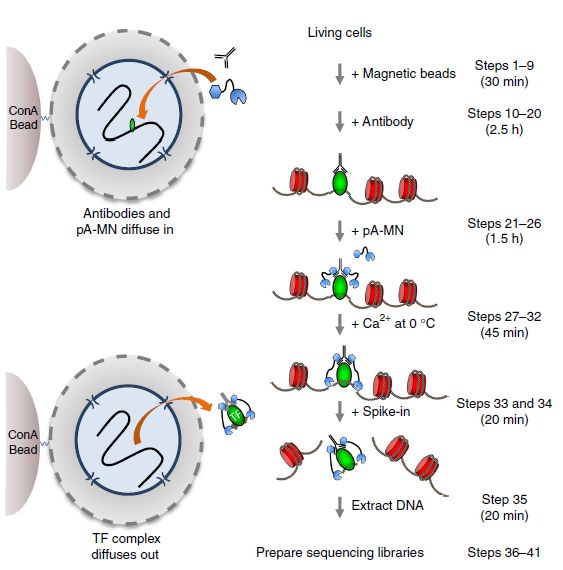
图 4:CUT&RUN 原理 [3]
利用连有刀豆蛋白 A 的磁珠(concanavalin A-coated magnetic beads)结合细胞。使用非离子去污剂洋地黄皂苷进行细胞膜通透。然后孵育靶蛋白(如转录因子, TF )的抗体和 Protein A-MNase 。抗体和 Protein A-MNase 能够通过核孔进入细胞核,MNase 通过 Protein A 和抗体的介导切割靶蛋白附近的 DNA 序列。 MNase 的活化需要 Ca2+,可通过加入 Ca2+ 或者螯合剂来启动或终止反应,进而将靶蛋白结合的 DNA 序列从细胞核中释放到上清中,用以建库。
✦ uliCUT&RUN :
2019 年 4 月 4 日,发表在 Cell 期刊,文章进一步将该技术升级到单细胞水平,并应用于早期胚胎。升级版 CUT&RUN 被称为超低量 CUT&RUN(ultra-low input CUT&RUN, uliCUT&RUN)。

图 5:uliCUT&RUN 文章 [4]
相对于 CUT&RUN 技术,uliCUT&RUN 的改进包括:Buffer、体系、孵育时间、spike-in DNA 量以及建库制备和纯化过程。
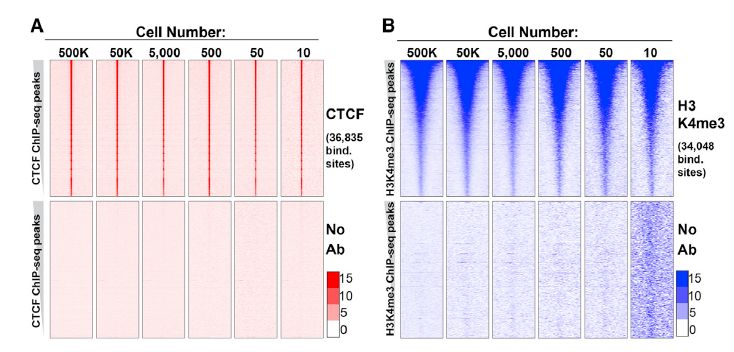
图 6:不同细胞量 mESCs 的 uliCUT&RUN 数据结果[4]
将不同细胞量 mESC 的 uliCUT&RUN 数据中的 CTCF 和 H3K4me3 测序结果与已有文献发表的 ChIP-seq 数据进行 read 密度可视化比较,发现 10 个细胞起始量的测序结果可以展现出类似 50 万细胞量的结合位点富集热图。其中 CTCF 的结合区更为狭窄集中,而 H3K4me3 则占据相对宽的区段。
✦ CUT&Tag(Cleavage Under Targets and Tagmentation):
Henikoff博士于 2019 年 4 月 29 日发表在 Nature Communication 上关于 CUT&Tag 的文章,让细胞量从 104 级别降到 60 个细胞甚至单细胞。
 图 7:CUT&Tag 文献[5]
图 7:CUT&Tag 文献[5]
✦CUT&Tag 原理:
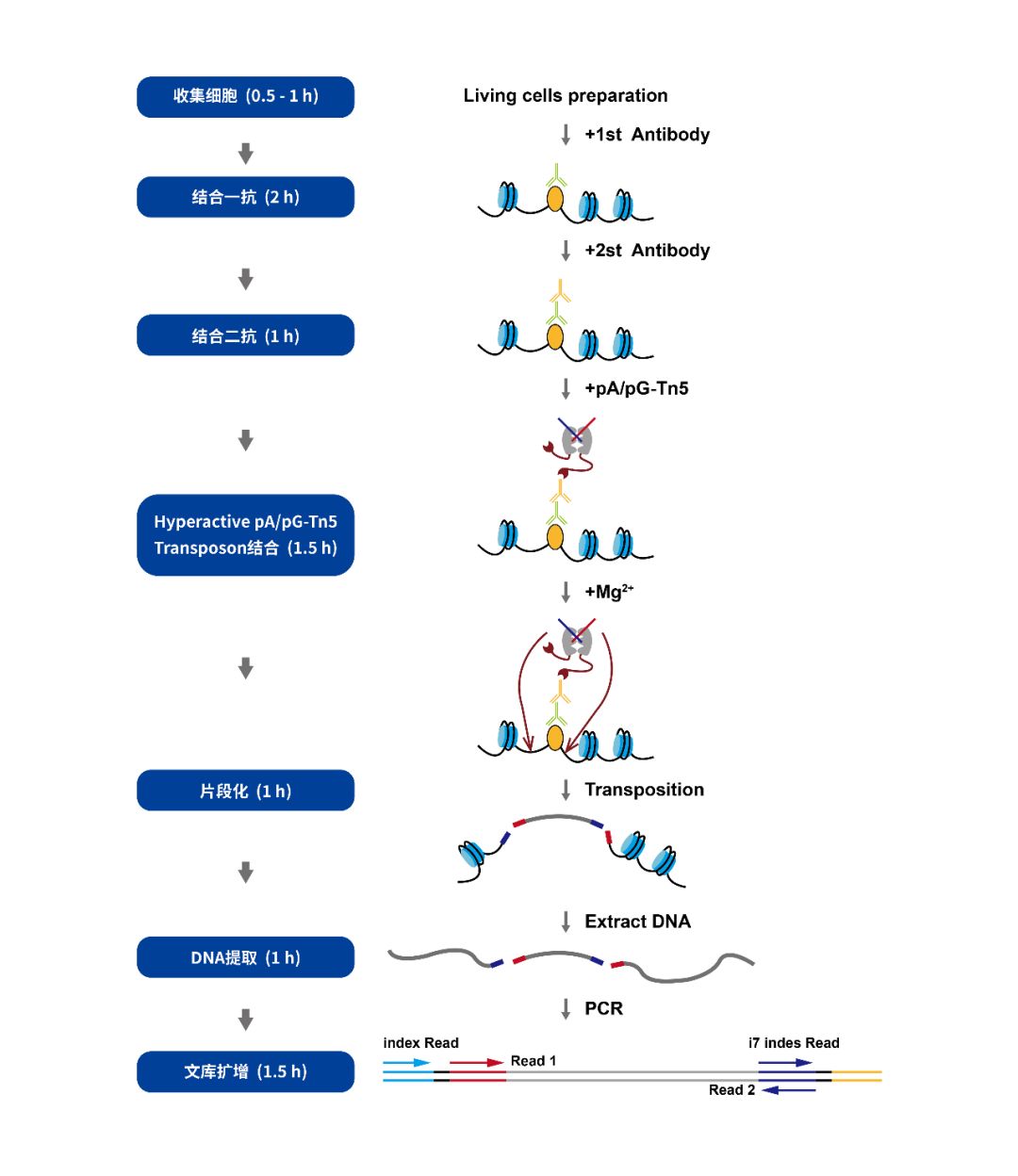
图 8:CUT&Tag 原理图[5]
利用连有刀豆蛋白 A 的磁珠(concanavalin A-coated magnetic beads)结合细胞(刀豆蛋白 A 能与细胞膜上的糖蛋白结合)。使用非离子去污剂洋地黄皂苷进行细胞膜通透。然后分步孵育靶蛋白(如转录因子, TF)的抗体(一抗)、二抗、和 Protein A-Tn5 ,抗体和 Protein A-Tn5 能够通过细胞膜、核孔进入细胞核,Tn5 通过 Protein A 和抗体的介导切割靶蛋白附近的 DNA 序列。使用 Tn5 在进行切割的时候,会在被切割的片段两端加上接头序列,通过 PCR 扩增直接可以得到二代测序文库。
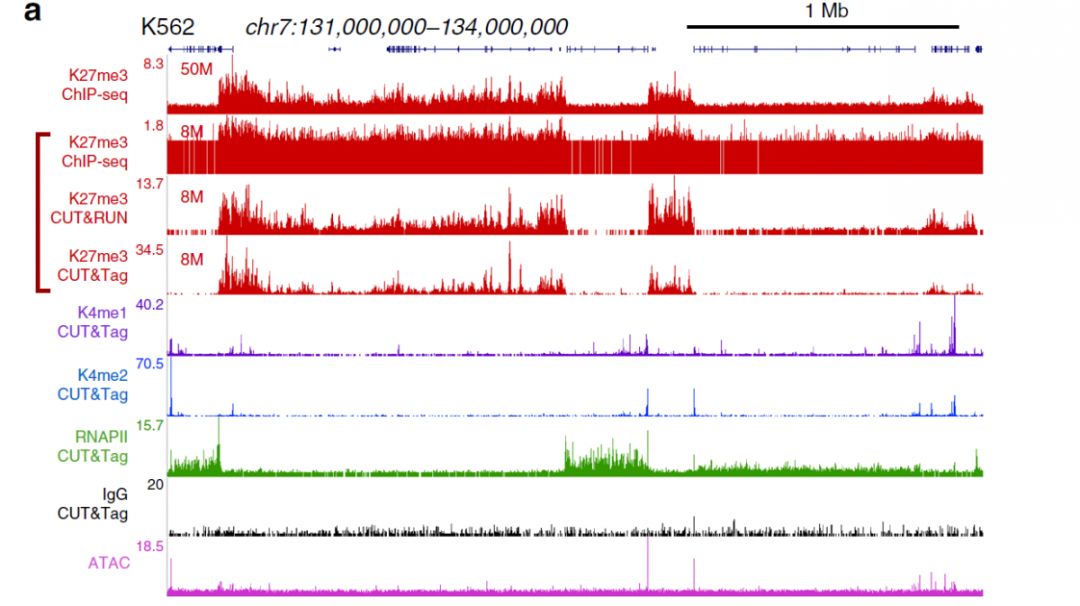
图 9: CUT&Tag、CUT&RUN 的 peaks 鉴定结果与 ChIP-seq 的对比[5]
Henikoff 等使用了 ChIP-seq 、CUT&RUN 、CUT&Tag 三种方法来对组蛋白 H3K27me3 进行研究。通过相同的数据量(8M Reads)进行比较分析发现,三种方法对应的染色体景观相似,但是 ChIP-seq 需要更高的测序深度才能达到去背景噪音的效果,CUT&RUN 和 CUT&Tag 的信噪比远远高于 ChIP-seq 。
✦ CUT&Tag 单细胞操作流程:
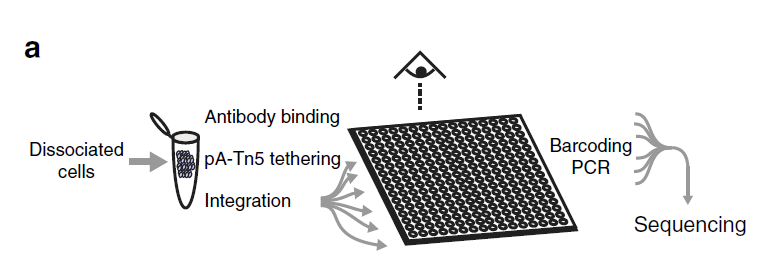
图 10:单细胞 CUT&Tag 操作流程[5]
CUT&Tag 能够对极少量细胞( 60 个细胞)进行操作,还可以进行单细胞操作。进行单细胞操作需要将细胞进行分装,由于 Protein A-Tn5 在细胞内进行,PCR 反应之前,切割的核酸均在细胞内, Henikoff 博士通过分配单个细胞到 5184 纳米孔,再加入带随机标签的引物进行扩增的办法,实现单细胞测序。
✦其他蛋白质-DNA 互作研究方法:
2019 年 3 月以来,bioRxiv 上传了数篇蛋白-DNA 互作研究的预印文章,包括 CoBATCH [5]和 ACT-seq [6]技术等,同样基于 Tn5 与融合蛋白进行 ChIP 技术的创新,原理基本相同。
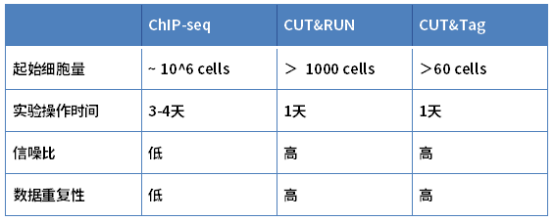
3. ChIP-seq、CUT&RUN、CUT&Tag技术比较
4. CUT&Tag、CUT&RUN技术核心
Protein A-Tn5、Protein G-Tn5 以及 Protein A-MNase 三种酶是 CUT&Tag、CUT&RUN 技术中的关键核心酶。由于CUT&Tag、CUT&RUN 均是针对极低起始量细胞进行实验,因此对核心酶原料有极高要求。
首先是 Tn5 转座酶需要具有高活性,才能保证对微量 DNA 的高灵敏度和高亲和力,有效抓取数十个细胞中的有限结合位点;第二,由于 Tn5 转座酶对 DNA 的高亲和力,纯化过程中易产生非特异核酸残留。应用在仅有数十个细胞的 CUT&Tag实验中,非特异核酸残留会对建库效果和测序数据产生较大影响。因此,选择 CUT&Tag 用酶时,一方面需要对 Tn5 转座酶进行定向进化和突变,选择高活性突变株;另一方面,需要有较高的蛋白纯化工艺,尽可能降低非特异核酸残留。
参考文献
[1] Orlando, V., Strutt, H., & Paro, R. (1997). Analysis of chromatin structure byin VivoFormaldehyde cross-linking. Methods, 11(2), 205-214.
[2] Skene, P.J., and Henikoff, S. (2017). An efficient targeted nuclease strategy for high-resolution mapping of DNA binding sites. eLife 6, e21856.
[3] Skene, P.J., Henikoff, J.G., and Henikoff, S. (2018). Targeted in situ genomewide profiling with high efficiency for low cell numbers. Nat. Protoc. 13, 1006–1019.
[4] Kaya-Okur, H. S., Wu, S. J., Codomo, C. A., Pledger, E. S., Bryson, T. D., Henikoff, J. G., ... & Henikoff, S. (2019). CUT&Tag for efficient epigenomic profiling of small samples and single cells. Nature communications, 10(1), 1930.
[5] Wang, Q., Xioong, H., Ai, S., Yu, X., Liu, Y., Zhang, J., & He, A. (2019). CoBATCH for high-throughput single-cell epigenomic profiling. bioRxiv, 590661.
[6] Carter, B., Ku, W. L., Tang, Q., Kang, J. Y., & Zhao, K. (2019). Mapping Histone Modifications in Low Cell Number and Single Cells Using Antibody-guided Chromatin Tagmentation (ACT-seq). bioRxiv, 571208.
文章图文来源:诺唯赞生物