比较基因组杂交(CGH)
互联网
9124
比较基因组杂交 (CGH)
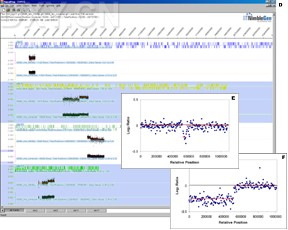
比较基因 组杂交主要应用于肿瘤染色体变异等研究。这里主要介绍采用基因芯片的CGH,其原理是:先分别将肿瘤与正常细胞的基因组DNA打碎,再利用双色荧光杂交的策略,对肿瘤细胞基因组DNA片段进行红色荧光(Cy5荧光素)标记,而对照的正常基因组DNA片段用绿色荧光(Cy3荧光素)标记,再将这些标记后的片段作为探针,与高密度的涵盖整个人类基因组的DNA芯片杂交。通过这两组探针的竞争杂交,再利用共聚焦显微镜等数码摄像系统对结果进行扫描,并对芯片上每个点(对应于人类基因组的不同位置)的发光强度进行比较。红绿色均等的区域是肿瘤细胞与正常细胞均具有的正常部分,而红色较强的部分即肿瘤细胞染色体区域产生扩增的部分;反之,以绿色为主的部分则是肿瘤细胞染色体缺失的部分。分离到的肿瘤组织DNA可成功地用于检测整个基因组的DNA缺失或扩增。
CGH应用于肿瘤遗传学的研究,可以提供一个全基因组的扫描图,提示肿瘤DNA在整个染色体组的哪个特定位置存在扩增或缺失,那些发生扩增的区域,极有可能存在癌基因,而缺失区域即可能包含一些抑癌基因。至今以此方法已发现了多种肿瘤的抑癌基因和癌基因。现今CGH主要是采用高分辨率的DNA芯片,如SMART阵列(submegabase resolution ntiling set forarray CGH)和MA(representational oligonucleotide microarray analysis)和SNP芯片。SMART阵列由32 433个细菌人工染色体(BAC)克隆制成,涵盖了人类的全基因组,覆盖率达到1.5倍,分辨率约为80kb。Ishkanian等人使用SMART发现了一些新的肿瘤细胞扩增区域和缺失区域,并且所确定的异常染色体区域比其他技术所确定的更小,因而更有利于寻找疾病基因。ROMA是Lucito和Wigler等人电子克隆了人全基因组BgIⅡ片段而设计的,含85 000个70bp长度的探针,分辨率达到30kb,综合了代表性差别分析(representative difference analysis,RDA)、CGH和微阵列等技术。而分辨率最高的显然要数Affymetrix公司的GeneChip Mapping 500KArraySet。利用这些高分辨率的DNA芯片,将能找到更为细小的DNA扩增或缺失区域。