Active Motif ChIP 实验专家技术经验分享:如何判断 ChIP 成功与否?
Active Motif
上一篇分享了做好 ChIP 实验需要注意的一些细节。但是注意了这些细节,按照流程完成了实验就结束了吗?当然没有,接下来的问题又来了。得到了目的片段 DNA,但是怎么判断得到的片段是不是自己想要的呢?
换句话说,怎么判断 ChIP 是不是成功呢?
IP 之后的染色质经过洗脱,解交联,RNase A 和 Protease K 处理,抽提或 DNA 纯化柱回收(这些步骤类似于前面的检测超声效果)就可以获得目的蛋白结合的 DNA。后续的这些步骤对于有分子生物学基础的人来说并不会存在太大的问题。需要说明的是,很多刚开始做 ChIP 的新手会以 ChIP 得到的 DNA 总量来判断 ChIP 好坏,这实际上是不正确的。虽然很多测序公司会要求提供特定量的 ChIP DNA 用于建库,但是 ChIP 得到的 DNA 量跟 ChIP 成功与否没有直接关系,因为很多蛋白的 ChIP 得到的总 DNA 大部分为背景 DNA。一般只有通过 ChIP-qPCR 或 ChIP-PCR 验证才能确定 ChIP 成功与否。
如何判断 ChIP 是否成功?这包含了两个层次的问题。
第一,整个实验是否 work?
一般来说,不管所用抗体是否是 ChIP 级,首先都要确定抗体是否能做你的 ChIP。如果是 ChIP 级抗体,就严格遵照产品说明书做,或根据别人文章报道的方法做。如果不是 ChIP 级抗体,需要先确定是否可以做 IPIF。如果可行,那再进一步尝试是否可以用于 ChIP。比如可以尝试根据别人文章报道的方法来做。如果没有被报道过,那就只能去推测可能的结合区域,设计引物去做 qPCR 验证。如果都不能确定,那只能通过测序判断了。如果你是新手,可以同时做一个阳性对照 ChIP,比如 RNA pol II 的 ChIP(用于对照转录因子)或 H3K4me3 的 ChIP(用于对照组蛋白修饰)。即使确定抗体可以用于做 ChIP,但对于目的细胞样本,如果之前没有做过,而且不确定目的蛋白结合的区域,一般也可以用别人报道过的对照细胞株作为对照,与目的细胞样本同时做 ChIP,确定 ChIP 是否成功。
第二,整个实验是否真的 work?
也就是如何排除假阳性?这就需要做阴性对照。阴性对照包括阴性对照抗体 IgG 和阴性对照区域(即阴性对照引物)。阴性对照 IgG 用于排除目的蛋白结合的 DNA 区域是抗体非特异性结合的,区域对照排除目的蛋白结合的 DNA 是背景 DNA。
如何通过 ChIP-qPCR 判断 ChIP 是否成功?
ChIP 成功与否一般是通过 ChIP-qPCR 计算富集度的方式来判断的。
一般来说,通过 ChIP-qPCR 判断 ChIP 成功与否是通过比较目的蛋白在阳性区域和阴性区域的富集度差异来判断的。大体上,阳性区域比阴性区域富集度高 4~8 倍以下(也就是 2~3 个 cycles),我们可以认为 ChIP 没有成功;如果在 4~8 倍以上,说明目的蛋白在阳性区域有富集,可以初步认为 ChIP 成功了,但具体还要依蛋白而定。比如 H3K4me3 的 ChIP,通常阳性区域比阴性区域的富集度要高 100 倍以上才可以认为做的不错,但对于很多与染色质结合不是很紧密的蛋白,比如一些转录因子,可能富集度有 10 倍差异就不错了。一般对于已有报道的蛋白的 ChIP,可以参考别人的报道来判断;而对于没有报道的蛋白 ChIP,一般能做出有 4~8 倍以上的差异,就可以初步认为成功了。
具体富集度是怎么计算的呢?举个例子来说明:
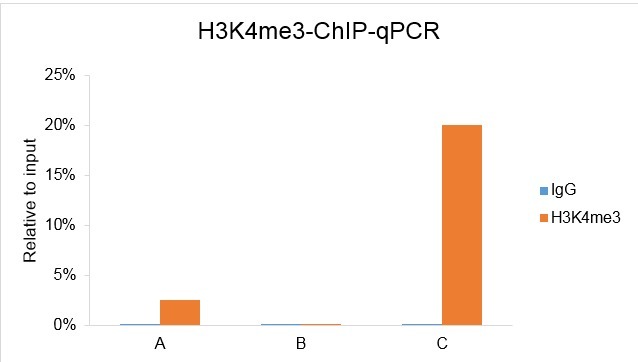
用作一个 ChIP 反应的起始样品叫做 Input,比如你用 20 ug 染色质去做一个 ChIP,那你的 Input 就是 20 ug。在做 ChIP 之前我们一般会取出一定比例的 Input 作为对照,我们通常称之为% 的 Input。如我们一共有 41 ug 的染色质,准备用 20 ug 做一个 H3K4me3 的 ChIP,同时再用 20 ug 做一个 IgG 的对照 ChIP。最后用剩余的 1 ug 染色质做对照,这 1 ug 的染色质相当于一个 ChIP 反应所用染色质(20 ug)的 5%,我们称之为 5% 的 Input。那么就说我们用了 5% 的 Input(1 ug/20 ug=0.5%)作为对照。
ChIP 结束之后,将 5% Input DNA,H3K4me3 ChIP DNA 以及 IgG ChIP DNA 纯化之后都溶解到等体积的水、10 mM Tris pH 8.0 或 TE buffer 中。分别设计目的区域 A,阴性区域 B 和阳性区域 C 的引物。 引物扩增片段大小以小于超声后 DNA 大小为宜(一般设计 70~200 bp),因为扩增片段太大,上下游引物结合的模板有可能不是位于同一个 DNA 片段上,导致扩增效率较低,甚至会影响真实富集度。准备好之后分别用 5% Input,H3K4me3 ChIP,IgG ChIP 的 DNA 作为模板,qPCR 分别扩增 A,B,C 三个区域。H3K4me3 在任何一个区域的富集都都可以用这个公式计算:5% × 2[CT value (Input-H3K4me3)],同样 IgG 的富集度的计算公式是:5% × 2[CT value (Input-IgG)]。
如下表所示:
| CT values | 5% Input | IgG | H3K4me3 |
| A (Target region) | 26 | 32 | 27 |
| B (Negative region) | 25 | 32 | 33 |
| C (Positive region) | 25 | 31 | 23 |
表 1,CT 值汇总
根据上面的公式推导出富集度如下表格所示:
| Enrichment | IgG | H3K4me3 |
| A (Target region) | 5%×2(26-32)≈0.08% | 5%×2(26-27)=2.5% |
| B (Negative region) | 5%×2(25-32)≈0.04% | 5%×2(25-33)≈0.02% |
| C (Positive region) | 5%×2(25-31)≈0.08% | 5%×2(25-23)=20% |
表 2,富集度汇总
通过计算富集度,我们就可以清楚地知道目的蛋白在感兴趣的 DNA 区域的结合强弱。如下图所示:
确定 ChIP 成功之后可以将样品建库,测序用于全基因组分析。
相关链接:Active Motif 技术专家经验分享:如何才能做好 ChIP 实验?
Active Motif ChIP 实验相关对照产品:
>> ChIP-IT® Control Kits
>> ChIP Control qPCR Primer Sets
>> ChIP-IT® qPCR Analysis Kit
更多产品详情欢迎咨询 Active Motif
Tel:021-20926090
Email:Techchina@activemotif.com