高通量测序(NGS)的原理简介
互联网
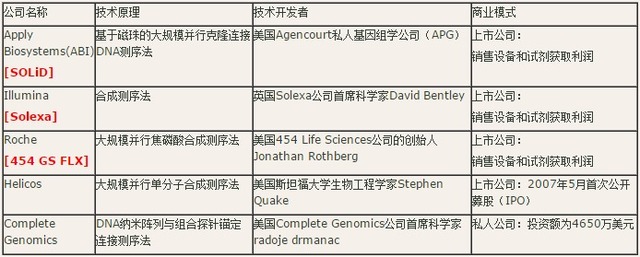
高通量测序(High-Throughput Sequencing)又名下一代测序(Next Generation Sequencing,NGS),是相对于传统的桑格测序(Sanger Sequencing)而言的。目前高通量测序的主要平台代表有罗氏公司(Roche)的454测序仪(Roch GS FLX sequencer),Illumina公司的Solexa基因组分析仪(Illumina Genome Analyzer)和ABI的SOLiD测序仪(ABI SOLiD sequencer)。
454公司首选将焦磷酸测序应用在测序技术上,之后便被罗氏诊断收购,形成了目前的Roche 454。待测DNA样品被打断成300-800bp的片段后,3' 和5'端分别加上接头,这些接头会使DNA片段结合到微珠上。测序PCR反应就发生在固相的微珠上,并且整个PCR反应和相关的酶被油包水的液滴包裹,每个油滴系统只包含1个DNA模板。扩增后,每个DNA分子可以得到富集,每个微珠只能形成一个克隆集落。454测序仪的测序通道体积非常狭小,只能容纳一个微珠。测序过程中,GS FLX系统会将引物上dNTP的聚合与荧光信号释放偶联起来。通过检测荧光信号,就可以达到DNA测序的目的。相对于Sanger测序,Solexa和Solid测序而言,454可以提供中等的读长和适中的价格,适合de novo 测序、转录组测序、宏基因组研究等。
Solexa的测序原理是可逆终止化学反应。DNA片段加上接头之后,可以随机的附着于玻璃表面(Flow cell),并且在固相的表面经过桥式扩增。这样就形成了数千份相同的单分子簇,被用做测序模板。测序采取边合成边测序的方法,和模板配对的ddNTP原料被添加上去,不配对的ddNTP原料被洗去,成像系统能够捕捉荧光标记的核苷酸。随着DNA 3'端的阻断剂的去除,下一轮的延伸就可以进行。和焦磷酸测序不同,每次DNA的只能延伸一个核苷酸。Solexa的读长在100-150bp之间,适合小RNA鉴定、甲基化和表观遗传学研究。
ABI Solid技术的核心是4种荧光标记寡核苷酸的连接反应测序。测序之前,DNA模板通过乳化PCR扩增,和Roche 454的基本相同,只是Solid的微珠更小,只有1μm。3'端修饰的微珠可以沉淀在玻片上。连接测序所用的底物是8个碱基荧光探针混合物,根据序列的位置,样品DNA就可以被探针标记。DNA连接酶优先连接和模板配对的探针,并引发该位点的荧光信号的产生。Solid的读长只有50-75bp,精确度可达Q40,适于基因组重测序和SNP检测。
三个厂家的高通量测序仪器虽然各具特点,在原理上很多的共同之处:
1 将目标DNA剪切为小片段
2 单个小片段DNA分子结合到固相表面
3 单分子独立扩增
4 每次只复制一个碱基(A,C,T,G)并检测信号
5 高分辨率的成像系统
高通量测序以其高输出量与高解析度的特性,不仅为我们提供了丰富的遗传学信息,而且使得测序的费用和时间大大缩短。在高通量测序发展的过程中,也有很多的问题需要我们去解决:数据在临床诊断上的作用,测序数据的储存和分析,数据的安全和信息隐私等。