基因敲除技术
互联网
1137
一.概述:
基因敲除是自80年代末以来发展起来的一种新型分子生物学技术,是通过一定的途径使机体特定的基因失活或缺失的技术。通常意义上的基因敲除主要是应用DNA 同源重组原理,用设计的同源片段替代靶基因片段,从而达到基因敲除的目的。随着基因敲除技术的发展,除了同源重组外,新的原理和技术也逐渐被应用,比较成功的有基因的插入突变和iRNA ,它们同样可以达到基因敲除的目的。
二.实现基因敲除的多种原理和方法:
1.利用基因同源重组进行基因敲除
基因敲除是80年代后半期应用DNA 同源重组原理发展起来的。80年代初,胚胎干细胞(ES细胞)分离和体外培养的成功奠定了基因敲除的技术基础。1985年,首次证实的哺乳动物细胞中同源重组的存在奠定了基因敲除的理论基础。到1987年,Thompsson首次建立了完整的ES细胞基因敲除的小鼠模型[1]。直到现在,运用基因同源重组进行基因敲除依然是构建基因敲除动物模型中最普遍的使用方法。
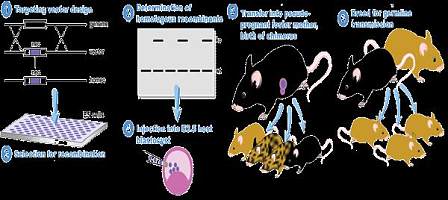
(1)利用同源重组构建基因敲除动物模型的基本步骤:
①. 基因载体的构建:把目的基因和与细胞内靶基因特异片段同源的DNA 分子都重组到带有标记基因(如neo 基因,TK 基因等)的载体上,成为重组载体。基因敲除是为了使某一基因失去其生理功能,所以一般设计为替换型载体。
②.ES 细胞的获得:现在基因敲除一般采用是胚胎干细胞,最常用的是鼠,而兔,猪,鸡等的胚胎干细胞也有使用。常用的鼠的种系是129及其杂合体,因为这类小鼠具有自发突变形成畸胎瘤和畸胎肉瘤的倾向,是基因敲除的理想实验动物。而其他遗传背景的胚胎干细胞系也逐渐被发展应用。[2,3]
③.同源重组:将重组载体通过一定的方式(电穿孔法或显微注射)导入同源的胚胎干细胞(ES cell)中,使外源DNA 与胚胎干细胞基因组中相应部分发生同源重组,将重组载体中的DNA 序列整合到内源基因组中,从而得以表达。一般地,显微注射命中率较高,但技术难度较大,电穿孔命中率比显微注射低,但便于使用。[4,5]
④.选择筛选已击中的细胞:由于基因转移的同源重组自然发生率极低,动物的重组概率为10-2 ~10-5 ,植物的概率为10-4 ~10-5 。因此如何从众多细胞中筛出真正发生了同源重组的胚胎干细胞非常重要。目前常用的方法是正负筛选法(PNS法),标记基因的特异位点表达法以及PCR 法。其中应用最多的是PNS法。[6]
⑤.表型研究:通过观察嵌和体小鼠的生物学形状的变化进而了解目的基因变化前后对小鼠的生物学形状的改变,达到研究目的基因的目的。[2,3,7]
⑥.得到纯合体:由于同源重组常常发生在一对染色体上中一条染色体中,所以如果要得到稳定遗传的纯合体基因敲除模型,需要进行至少两代遗传。[8]
(2)条件性基因敲除法
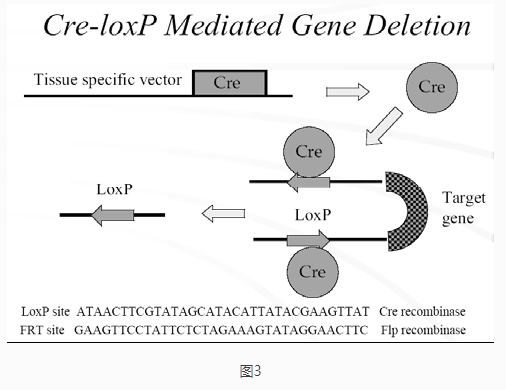
条件性基因敲除法可定义为将某个基因的修饰限制于小鼠某些特定类型的细胞或发育的某一特定阶段的一种特殊的基因敲除方法[2]。它实际上是在常规的基因敲除的基础上,利用重组酶Cre介导的位点特异性重组技术,在对小鼠基因修饰的时空范围上设置一个可调控的“按钮”,从而使对小鼠基因组的修饰的范围和时间处于一种可控状态。
条件性敲除的原理:
利用Cre/LoxP 和来自酵母的FLP—frt 系统可以研究特定组织器官或特定细胞中靶基因灭活所导致的表型[7]。通过常规基因打靶在基因组的靶位点上装上两个同向排列的1oxP,并以此两侧装接上loxP 的(“loxP floxed”)ES 细胞产生“loxPfloxed”小鼠,然后,通过将“loxP floxed”小鼠与Cre 转基因鼠杂交(也可以其他方式向小鼠中引入Cre 重组酶),产生靶基因发生特定方式(如特定的组织特异性)修饰的条件性突变小鼠。在“loxP floxed”小鼠,虽然靶基因的两侧已各装上了一个loxP,但靶基因并没有发生其他的变化,故“1oxP noxed”小鼠表型仍同野生型的一样。但当它与Cre 转基因小鼠杂交时,产生的子代中将同时带有“loxP floxed”靶基因和Cre 基因。Cre 基因表达产生的Cre 重组酶就会介导靶基因两侧的1oxP 间发生切除反应,结果将一个loxP 和靶基因切除。这样,靶基因的修饰(切除)是以Cre 的表达为前提的。Cre 的表达特性决定了靶基因的修饰(切除)持性:即Cre 在哪一种组织细胞中表达,靶基因的修饰(切除)就发生在哪种组织细胞;而Cre 的表达水平将影响靶基因在此种组织细胞中进行修饰的效率。所以只要控制Cre 的表达特异性和表达水平就可实现对小鼠中靶基因修饰的特异性和程度[9,10]。


(3)诱导性基因敲除法
诱导性基因敲除也是以Cre/loxp 系统为基础,但却是利用控制Cre 表达的启动子的活性或所表达的Cre 酶活性具有可诱导的特点,通过对诱导剂给予时间的控制或利用Cre 基因定位表达系统中载体的宿主细胞特异性和将该表达系统转移到动物体内的过程在时间上的可控性,从而在1oxP 动物的一定发育阶段和一定组织细胞中实现对特定基因进行遗传修饰之目的的基因敲除技术。人们可以通过对诱导剂给予时间的预先设计的方式来对动物基因突变的时空特异性进行人为控制、以避免出现死胎或动物出生后不久即死亡的现象。常见的几种诱导性类型如下:四环素诱导型(图4);干扰素诱导型;激素诱导型;腺病毒介导型。
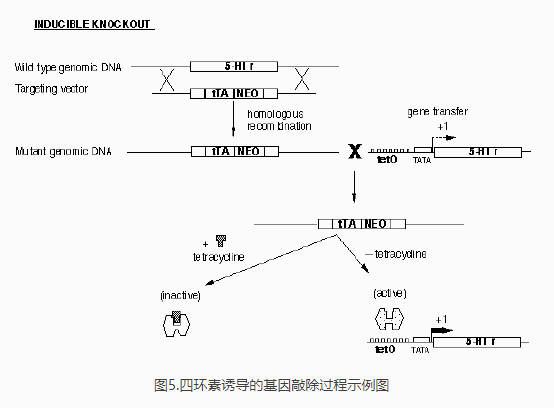
诱导性基因敲除优点:
① 诱导基因突变的时间可人为控制;
② 可避免因基因突变而致死胎的问题
③ 在2 个loxP 位点之间的重组率较高;
④如用病毒或配体/DNA 复合物等基因转移系统来介导Cre 的表达,则可省去建立携带Cre 的转基因动物的过程。[10]
2.2 利用随机插入突变进行基因敲除。
2.2.1 原理:
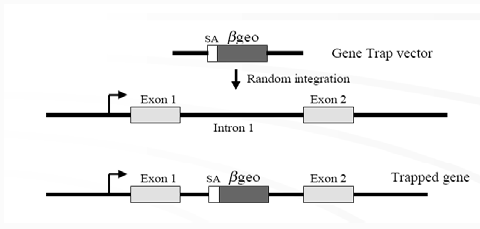
此法利用某些能随机插入基因序列的病毒,细菌或其他基因载体,在目标细胞基因组中进行随机插入突变,建立一个携带随机插入突变的细胞库,然后通过相应的标记进行筛选获得相应的基因敲除细胞(原理见图5)[11,12] 。根据细胞的不同,插入载体的选择也有所不同。逆转率病毒可用于动植物细胞的插入;对于植物细胞而言农杆菌介导的T-DNA转化和转座子比较常用;噬菌体可用于细菌基因敲除。
插入了靶目标的基因转录翻译出无活性的目标蛋白随机插入内含子中
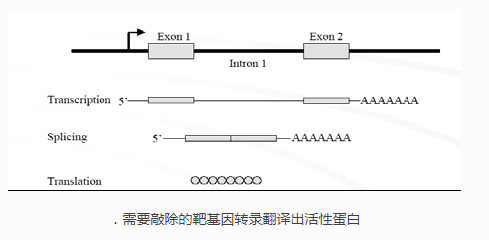
.需要敲除的靶基因转录翻译出活性蛋白
图6:基因捕获法的基本原理图
2.2.2基因捕获法
基因捕获法是最近发展起来的利用随机插入突变进行基因敲除的新型方法,其原理可见图6。通常基因捕获载体还包括一个无启动子的报道基因,通常是neo 基因,neo 基因插入到ES 细胞染色体组中,并利用捕获基因的转录调控元件实现表达的ES 克隆可以很容易地在含G418 的选择培养基中筛选出来,从理论上讲,在选择培养基中存活的克隆应该100%地含有中靶基因。中靶基因的信息可以通过筛选标记基因侧翼cDNA或染色体组序列分析来获得[13]
2.2.3基因捕获法的优缺点
用常规方法进行基因敲除研究需耗费大量的时间和人力,研究者必须针对靶位点在染色体组文库中筛选相关的染色体组克隆,绘制相应的物理图谱,构建特异性的基因敲除载体以及筛选中靶ES 细胞等,通常一个基因剔除纯合子小鼠的获得需要一年或更长的时间。面对人类基因组计划产生出来的巨大的功能未知的遗传信息,传统的基因敲除方法显得有些力不从心。因此,基因捕获法应运而生,利用基因捕获可以建立一个携带随机插入突变的ES 细胞库,节省大量筛选染色体组文库以及构建特异打靶载体的工作及费用,更有效和更迅速地进行小鼠染色体组的功能分析。
此方法的缺点是只能剔除在Es 细胞中表达的基因.单种的细胞类型中表达的基因数目约为I04,现在的基因捕获载体从理论上来讲应能剔除所有在ES细胞表达的基因,因此,在ES 细胞中进行基因捕获还是大有可为的。用基因捕获法进行基因剔除的另一个缺点是无法对基因进行精细的遗传修饰,
2.3.RNAi引起的基因敲除。
由于少量的双链RNA就能阻断基因的表达,并且这种效应可以传递到子代细胞中,所以RNAi的反应过程也可以用于基因敲除。近年来,越来越多的基因敲除采用了RNAi这种更为简单方便的方法。[13-15]
2.3.1 RNAi阻断基因表达的机理
双链RNA进入细胞后,能够在Dicer酶的作用下被裂解成siRNA,而另一方面双链RNA还能在RdRP (以RNA为模板指导RNA合成的聚合酶RNA-directed RNA
polymerase,RdRP)的作用下自身扩增后,再被Dicer酶裂解成siRNA。SiRNA的双链解开变成单链,并和某些蛋白形成复合物,Argonaute2是目前唯一已知的参与复合物形成的蛋白。此复合物同与siRNA互补的mRNA结合,一方面使mRNA被RNA酶裂解,另一方面以SiRNA作为引物,以mRNA为模板,在RdRP作用下合成出mRNA的互补链。结果mRNA也变成了双链RNA,它在Dicer酶的作用下也被裂解成siRNA。这些新生成的siRNA也具有诱发RNAi的作用,通过这个聚合酶链式反应,细胞内的siRNA大大增加,显著增加了对基因表达的抑制。从21到23个核苷酸的siRNA到几百个核苷酸的双链RNA都能诱发RNAi,但长的双链RNA阻断基因表达的效果明显强于短的双链RNA[13]。
2.3.2 RNAi基因敲除的优点及应用
①.比用同源重组法更加简便,周期大大缩短。
②.对于哺乳动物,如对于一些敲除后小鼠在胚胎时就会死亡的基因,可以在体外培养的细胞中利用RNAi技术研究它的功能。
③.由于RNAi能高效特异的阻断基因的表达,它成为研究信号传导通路的良好工具。
④.RNAi还被用来研究在发育过程中起作用的基因,如可用RNAi来阻断某些基因的表达,来研究他们是否在胚胎干细胞的增殖和分化过程中其起着关键作用。
2.4实现基因敲除的其他原理。
除上述几种已经比较成熟并且普遍使用了的基因敲除原理外,还有一些基于其他原理的敲除技术正处于研究和完善过程中,如TFOs(Triple helix forming oligonucleotides)引导的基因敲除术[16]以及反义技术在基因敲除技术中的运用等[17]。随着遗传学,分子生物学理论的发展,新的基因敲除原理也在不断的发现和发掘中。
3.基因敲除技术的应用及前景:
①.建立生物模型。在基因功能,代谢途径等研究中模型生物的建立非常重要。基因敲除技术就常常用于建立某种特定基因缺失的生物模型,从而进行相关的研究。这些模型可以是细胞,也可以是完整的动植物或微生物个体。最常见的是小鼠,家兔、猪、线虫、酵母和拟南芥等的基因敲除模型也常见于报道。
②.疾病的分子机理研究和疾病的基因治疗。通过基因敲除技术可以确定特定基因的性质以及研究它对机体的影响。这无论是对了解疾病的根源或者是寻找基因治疗的靶目标都有重大的意义。
③.提供廉价的异种移植器官。众所周知,器官来源稀少往往是人体器官移植的一大制约因素,而大量廉价的异种生物如猪等的器官却不能用于人体。这是因为异源生物的基因会产生一些能引起人体强烈免疫排斥的异源分子,如果能将产生这些异源分子的基因敲除,那么动物的器官将能用于人体的疾病治疗,这将为患者带来具大的福音。如:PPL Therapeutics 公司于1999 年已成功地在猪的体细胞中用基因敲除技术敲除了α-1,3GT 基因。使每只猪都缺乏产生a1-3半乳糖基转移酶的基因的2个拷贝。这些酶在细胞表面产生一种糖分子,人体的免疫系统可以立即辨认出这种糖分子为异源性,从而引发超急性免疫排斥反应。在缺乏这种酶的情况下,超急性排斥反应即不会再发生[10]。
④.免疫学中的应用。同异源器官移植相似,异源的抗体用于人体时或多或少会有一定的免疫排斥,使得人用抗体类药物的生产和应用受阻。而如果将动物免疫分子基因敲除,换以人的相应基因,那么将产生人的抗体,从而解决人源抗体的生产问题。
⑤改造生物、培育新的生物品种。细菌的基因工程技术是本世纪分子生物学史上的一个重大突破,而基因敲除技术则可能是遗传工程中的另一重大飞跃。它为定向改造生物,培育新型生物提供了重要的技术支持。
4.基因敲除技术的缺陷
随着基因敲除技术的发展,早期技术中的许多不足和缺陷都已经解决,但基因敲除技术始终存在着一个难以克服的缺点,即敲掉一个基因并不一定就能获知该基因的功能,其原因包括:一方面,许多基因在功能上是冗余的,敲掉一个
在功能上冗余的基因,并不能造成容易识别的表型,因为基因家族的其他成员可以提供同样的功能;另一方面,对于某些必需基因,敲除后会造成细胞的致死性,也就无法对这些必需基因进行相应的研究了。
参考文献:
1.王又红 基因敲除技术的应用现状与发展前景 国外医学 1999 26-5
2.Muller,-U.Ten years of gene targeting: targeted mouse mutants,from vector
design to phenotype analysis.Mech-Dev.1999 Apr; 82(1-2): 3-21
3.Ledermann,-B.Embryonic stem cells and gene targeting.Exp-Physiol.2000 Nov;
85(6): 603-13
4.ARMINHALLMANundefined,ANNETTE RAPPEL,AND MANFRED SUMPER Gene replacement by homologous recombination in the multicellular green alga Volvox carteri
Proc.Natl.Acad.Sci.USA Vol.94,pp.7469–7474,July 1997
5.Thomas P.homologous recombination in human ES cell Nature publish on line 2003 10 Feb
6.刘德培方福德 基因敲除 生理科学进展 1995 26-1
7.Nelson RJ,Young KA.Behavior in mice with targeted disruption of
single genes.Neurosci Biobehav Rev 1998;22:453±62.
8.Andrew Holmes Targeted gene mutation approaches to the study of anxiety-like
behavior in mice Neuroscience and Biobehavioral Reviews 25 (2001)261±273
9.Hasty P et al; Nature;1991;350;243
10.yunfengli 基因敲除技术的最新进展 丁香园电子期刊2003-12
11.陈其军,肖玉梅等 植物功能组研究中的基因敲除技术 植物生理学通讯 2004-1,40:121~126
12.U.Klinner *,B.Sch€afer Genetic aspects of targeted insertion mutagenesis in yeasts FEMS Microbiology Reviews 28 (2004)201–223
13.Joel S.Bedford∗,Howard L.Liber Applications of RNA interference for studies in DNA damage processing,genome stability,mutagenesis,and cancer Seminars in Cancer Biology 13 (2003)301–308
14.Georg Mellitzera,b,Marc Halloneta,Lan Chena,Siew-Lan Anga,Spatial and temporal ‘knock down’ of gene expression by electroporation of double-stranded RNA and morpholinos into early postimplantation mouse embryos Mechanisms of Development 118 (2002)57–63
15.H_akan Thonberg~undefined Camilla C.Sch_eele,Cecilia Dahlgren,and Claes Wahlestedt Characterization of RNA interference in rat PC12 cells: requirement of GERp95 Biochemical and Biophysical Research Communications 318 (2004)927–934
16.Alokes Majumdar1,Alexander Khorlin2,Natalia Dyatkina2Targeted gene knockout mediated by triple helix forming oligonucleotides nature 1998
17.Roberto P.Stock1,Alejandro Olvera1,Ricardo Sánchez1,Inhibition of gene expression in Entamoeba histolytica with antisense peptide
nucleic acid oligomers Nature Publishing Group 2001