北大伊成器团队 Nature Method 发文,揭示单碱基编辑脱靶风险!
丁香学术
270
2018 年,南方科技大学贺建奎团队使用 CRISPR 基因编辑技术进行人体基因编辑,露露和娜娜两名婴儿因此降生。
这次冒进的实验,很快在全世界掀起轩然大波,引发了人类对基因编辑技术应用的深思。
贺建奎事件之所以造成如此大的轰动,不仅是因为他逾越了医学伦理的边界,更是因为他让两名婴儿暴露在了基因编辑技术最大的风险之下,那就是「脱靶效应」。
尽管学术界已经对 CRISPR 技术的安全性做了大量改进与研究,我们依旧无法摆脱基因编辑脱靶所带来的严重后果。一次错误的编辑,足以给人体带来不可挽回的严重伤害。
近年来,人类基因编辑的「工具包」不断被丰富,除了不同版本的 CRISPR 之外,以哈佛大学刘如谦教授为首的团队,开发出了更加精准的单碱基编辑「神器」,为基因编辑带了更多的可能。
但新的工具也带来了新的问题,那就是它们足够安全吗?

图片来源:Nature Method
2021 年 6 月 8 号,北京大学合成与功能生物分子中心伊成器带领团队,在 Nature 子刊 Nature Method 上刊登了题为 Detect-seq reveals out-of-protospacer editing and target-strandediting by cytosine base editors 的论文 [1], 报道了胞嘧啶碱基编辑器 (cytosine baseeditor,CBE) 的脱靶风险。
研究内容:
CBE 可以将碱基中脫氧胞苷(dC)转化为脫氧尿苷(dU),并最终转换为脱氧胸苷 (dT),实现 DNA 中 C 到 T 的编辑。

图片来源:Nature Method
也正是因为 CBE 进行的 C 转 T 编辑需要中间体 dU,研究人员设计了一套名为 Detect- Seq 的测序方法,通过尿嘧啶 DNA 糖基酶(Uracil-DNA Glycosylase, UDG)识别 dU 的存在,并以此捕捉被编辑的碱基。

图片来源:Nature Method
研究团队对此前未被报道过脱靶现象的,包括 VEGFA_site_2,HEK293_site_4 与 EMX1 在内的几个著名单碱基编辑靶点进行了 Detect-seq,并分别识别出了数十到数百个推测出来的 sgRNA 结合位点,这些位点都表现出了较强的 Detect-seq 信号。去除 CBE 所依赖的 sgRNA,APO 以及 UCI 后,相应的测序信号都出现了极大的降低。这些结果都表明了 CBE 脱靶效应的存在。
作者对脱靶现象进行了进一步细分,发现这些脱靶现象发生的靶点可以被分为 Cas9 依赖性以及 Cas9 非依赖型。

图片来源:Nature Method
为了验证 Detect-seq 的可靠性,作用将 Detect-seq 与此前被用来研究 Cas9 依赖型脱靶效应的工具进行了对比。与 GUIDE-seq 相比,Detect-seq 识别出了大部分 GUIDE-seq 在 EMX1 与 VEGFA_site_2 中识别到的位点,以及半数 HEK293_site_4 中识别到的位点。除此之外,Detect_seq 还识别出了大量 GUIDE-seq 未曾识别出的脱靶突变位点。
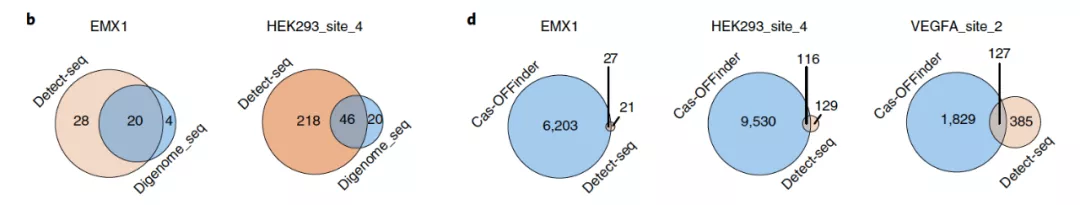
图片来源:Nature Method
与基于全基因组测序的脱靶效应研究工具相比,Detect-seq 也得到了相似的结果。同时,Detect-seq 能捕捉到 30-50% 由计算机预测出来的脱靶位点。这些数据都表明 Detect-seq 是一个特异性与敏感度优异的 CBE 脱靶效应研究工具。

图片来源:Nature Method
除了通过 sgRNA 推测出来的结合位点(putative sgRNA binding sites,sPBS)上发生的脱靶效应,研究团队还发现了更加令人惊讶的现象。那就是 CBE 的脱靶效应,还能发生在 sPBS 之外,这些编辑位点甚至可能离 sPBS 距离数个甚至数百个碱基。同时 CBE 所依赖的 Cas9 进行编辑需要原间隔序列临近基序(protospacer adjacentmotif,PAM)的存在,可该文作者发现,CBE 还造成了大量间隔序列外(out-of-protospacer)的脱靶编辑。
除此之外,CBE 应该只编辑含有 PAM 的 DNA 链,可研究团队还发现了不含 PAM 的链上,也出现了脱靶编辑。这些数据都表明,CBE 中广泛存在多种形式的脱靶效应。

图片来源:Nature Method
随后,作者探究了造成这些脱靶位点的原因。由于脱靶编辑更多的发生在 PAM 远端序列,研究团队推测 DNA 二级结构与 CBE 脱靶编辑有关。岭线性回归结果显示,PAM 远端的间隔序列外编辑与 pRBS 前 5 到 8 个碱基的错配数呈现出了明显的相关性。而 PAM 远近双侧的错配则与不含 PAM 链的脱靶编辑呈现相关。尽管有了这些数据,作者发现对 CBE 脱靶效应的预测依旧非常困难。
最后,研究团队对 YE1 以及 Cpf1 (Cas12a)-BE 等其他 CBE 工具进行了类似的评估。尽管 BE4max-YE1 表现出了更少的脱靶编辑,但依旧有巨大的提升空间。而基于 Cas12a 的碱基编辑工具,在 Detect-seq 中呈现出了更多的脱靶突变。这些数据再次说明了 Detect-seq 是一个有效的 CBE 脱靶效应研究工具,也表明了我们的基因编辑工具还需要进一步完善。
研究意义:
本文的研究结果,无疑给被寄予厚望的 CBE 泼了一盆冷水。
由于此前关于 CBE 脱靶效应的多个研究给出了不同的结果,本研究通过识别 dU 的产生建立了新的评估方法,更加有效的对 CBE 脱靶位点进行了鉴别,充分证明 CBE 能够导致大量的脱靶编辑。除此之外,作者还证明 CBE 相关的脱靶编辑可以发生在间隔序列外与不含 PAM 的 DNA 链,揭示 CBE 的临床使用需要接受更多的检验。
延伸阅读:
贺建奎的前车之鉴,让学术界对于单碱基编辑这种新兴基因编辑技术的安全性保持着警惕。
实际上,近几年关于单碱基编辑,尤其是 CBE 脱靶效应的研究层出不穷。

图片来源:Nature Biotechnology
2017 年,在刘如谦团队的单碱基编辑问世仅仅一年后,来自韩国首尔大学的 Jin-Soo Kim 团队就在 Nature Biotechnology 发表了题为 Genome-wide target specificities of CRISPR RNA-guided programmabledeaminases 的研究 [2], 在基因组层面评估了 CBE 的特异性,并给出了 CBE 高度特异的结论。
但两个来自中国的团队并不这么认为。

图片来源:Science

图片来源:Science
2019 年 3 月,来自中科院神经科学研究所的杨辉团队与来自中科院遗传与发育研究所高彩霞团队在 Science 上「背靠背」分别发表了题为 Cytosine base editorgenerates substantial off-target single nucleotide variants in mouse embryos[3] 与Cytosine, but not adenine,base editors induce genome-wide off-target mutations in rice[4] 的论文, 报道了 CBE 导致的脱靶突变,在对小鼠与水稻的基因编辑中广泛存在。
本次伊成器团队的新研究,再次给单碱基编辑的安全性蒙上了一层阴影,却也给 CBE 技术的进一步改进指明了新的方向。
参考文献: