手把手教你学会基因组本地比对
丁香园
在自己拥有一个或者更多基因组的情况下,如何在这些基因组中找到自己想要的功能基因,这对于基因组分析来说,比较重要。而利用测序公司给我的功能注释结果,由于所用的注释的数据库的老旧等原因,往往在注释结果是很不准确的, 在很多实验室中公司给的功能注释结果都是不会用的。本地比对对于基因组基因功能分析来说显得尤为重要。
对于本地比对,首先是要在 NCBI 下载本队比对软件 blast,下载在这里就不多说,下载好就需要进行安装了,win 安装步骤如下:
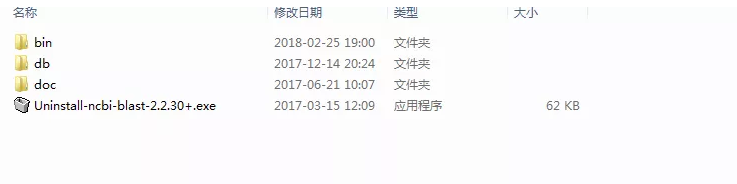
安装流程:双击安装到 C:Blast,生成 bin 和 doc 两个子目录,其中 bin 是程序目录,doc 是文档目录,这样就安装完成,同时需要另建一个 db 文件。
用户环境变量设置:右键点击「我的电脑」- 属性,然后「高级系统设置」选项 -「环境变量」,在用户变量下方点击「新建」- 变量名:BLASTDB,变量值:C:Blastdb(即数据库路径)。在系统变量下方「Path」添加变量值:C:Blastin。
最后在你的电脑 C 盘就有一个 blast 的文件夹,里面包括 bin、db 以及 doc 三个子目录,如下:
安装好后就是对你的自己序列的处理了。自己序列可以是你在公司测的一段或者更多序列,也可以是自己在网上下载的功能基因序列,比如毒力基因、耐药基因序列等,在做本地比对过程中,公司给的序列一般都不需要做处理,而自己下载的需要将序列中的空格和回车键去掉,不然会影响比对,这个我一般是将序列复制到 word 上面,当然 wps 也好操作,然后点击替换,举例如下:
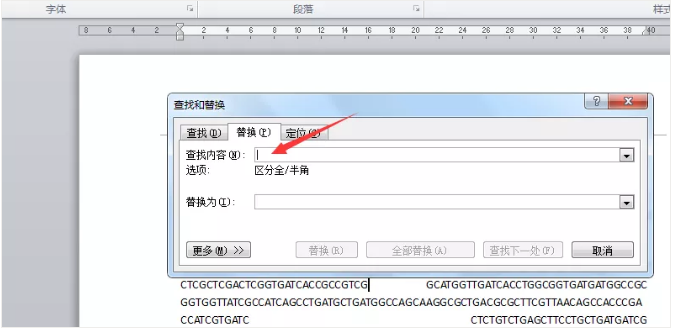
在查找内容处敲一个空格键,替换处不管,点全部替换,会显示有多少个空格键。
接下来是去回车键:

在查找内容出输入 ^p,就是按住 shift,然后按 6 键,最后在输入小写的 p,填好后点全部替换,所有的回车键就去掉了。
处理好自己的序列后,自己的序列需要保存为 txt 格式。切记多个基因可以放在一个文件里面,基因和基因之间是用回车键隔开的,如下:

最后需要将自己的序列文件放在 bin 文件里面,注意记住自己的命名,我举例命名为 zhongguo(中国)。
然后是建库,建库所用的序列往往是某些功能基因数据库下载的功能基因序列,比如细菌的耐药基因库,毒力基因库等,也可以是自己的基因组序列。这些都是直接把他们以 fasta 的格式放入 db 文件中就是了。也要记住名字,我举例的为 shijie(世界)。
最后就是比对了,首先是在电脑搜索处输入 cmd,然后打开 cmd 程序,如下:

输入命令,比对命令如下:
cd c:Blastdb
cd c:Blastdb>makeblastdb -in 建库名称.fasta -dbtype nucl -out 建库名称.fastadb
cd c:Blastin(将需要比对的序列复制粘贴进去)
cd c:Blastin>blastn -query 自己序列文件名称.txt -db 建库名称.fastadb –out 比对输出结果名称.txt
每一步复制上去后都要按回车键,命令输入号后,比对时间一般就是几秒钟,举例比对命令输入如下:

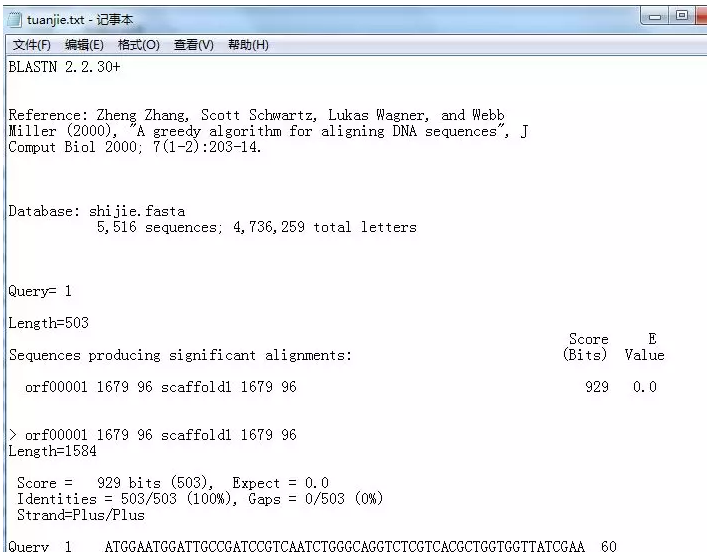
我举例中自己序列文件名称为 zhongguo,建库名称为 shijie,比对输出结果名称为 tuanjie。
最后在 bin 文件夹中去找自己命名的输出文件,我这里是 tuanjie,她是 txt 格式的,双击就可以打开了。我的比对结果如下:
详细的本地比对就这样了哦!