多序列比对的目的是发现多条序列的共性,对于研究分子结构、功能及进化关系极为有用。目前的方法还在不断地发展中,大多数算法都是基于渐进的比对的思想,在序列两两比对的基础上逐步优化多序列比对结果。目前常用的多序列比对程序有 MUSCLE、MAFFT、Clustal、COBALT、T-Coffee 等,它们都可以用于核算或蛋白序列的比对。
多序列比对的基本原理是发现多条序列的共性,对于研究分子结构、功能及进化关系极为有用。目前的方法还在不断地发展中,大多数算法都是基于渐进的比对的思想,在序列两两比对的基础上逐步优化多序列比对结果。
目前常用的多序列比对程序有 MUSCLE、MAFFT、Clustal、COBALT、T-Coffee 等,它们都可以用于核算或蛋白序列的比对。
(一)MUSCLE
A. 解压程序。建立 MUSCLE 安装目录,如: D : \muscle。将 MUSCLE 安装包解压到该目录。
B. 设置环境变量。将 MUSCLE 安装目录添加到环境目录中。
注意事项:这个步骤可以省略,但是设置后可以为使用带来方便。方法和上面 BLAST+中提到的设置方法一样。
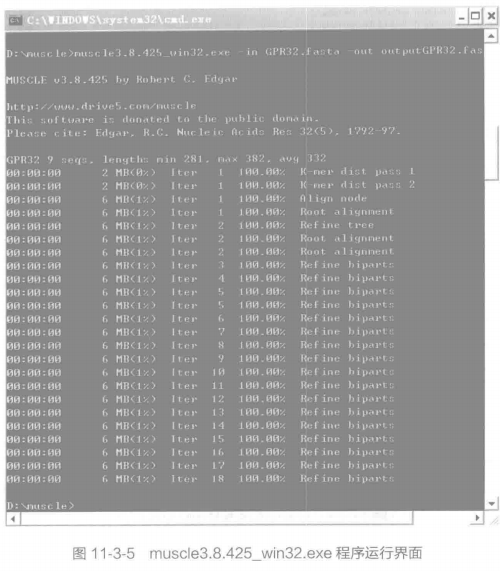
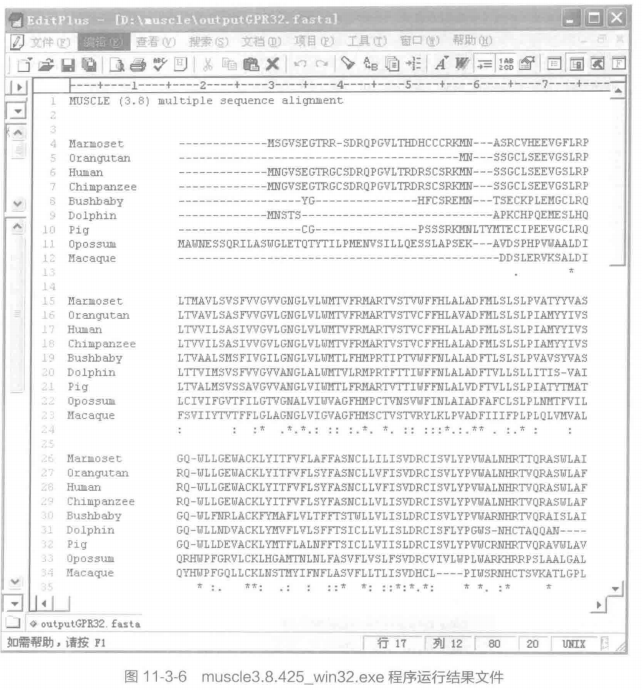
C. 使用方法。MUSCLE 属于 DOS 程序,运行时指定输人和输出文件就可以了。默认的输入、输出格式也是 fasta 格式。下面就以人 (human)、黑猩猩 (chimpanzee) 、 红毛猩猩 (orangutan)、 负鼠 (opossum)、从猴 (bushbaby)、 猪 ( pig)、猕猴 (macaque)、 狨猴 (marmoset) 和海豚 (dolphin) 等 9 个物种的"G 蛋白偶联受体 32 (G Protein-Coupled Receptors GPR32) "为例进行多序列比对,来说明 MUSCLE 的用法。首先可先将这些序列放置于 GPR32.fasta 中。输入比对命令:muscle3.8.425_ win32.exe -in GPR32 fasta-out outputGPR32.fasta-clw, 如图 11-3-5 所示。GPR32 的多列此对结果如下,结果中以特定的符号代表每个位点的保守性,比如: "」号表示保守性高的位点,如图 11-3-6 所示。
(二)ClustalX
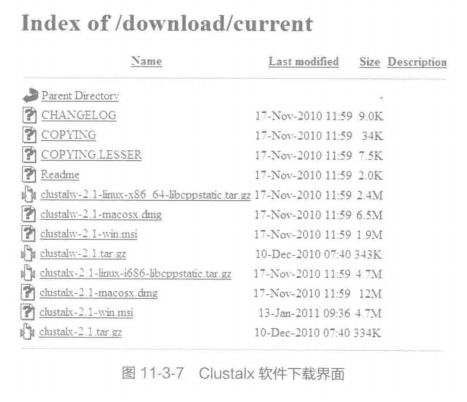
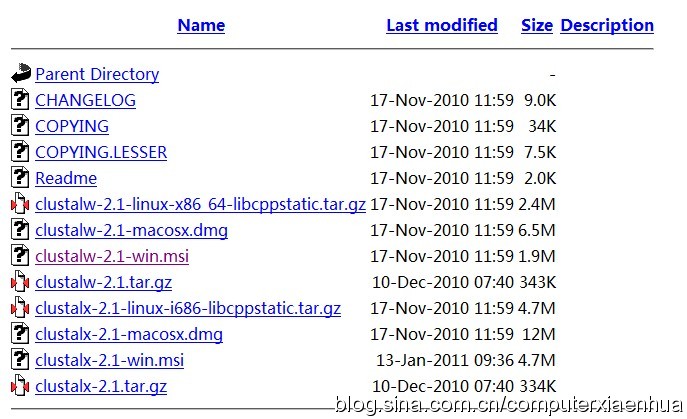
A. 安装。将 clustalx-2.1-win.msi 程序安装在 D : \clustal 下。
B. 使用方法。以人、黑猩猩、红毛猩猩、负鼠、丛猴、猪、猕猴、狨猴和海豚等 9 个物种的 GPR32 序列为例,进行 ClustalX 多序列比对。
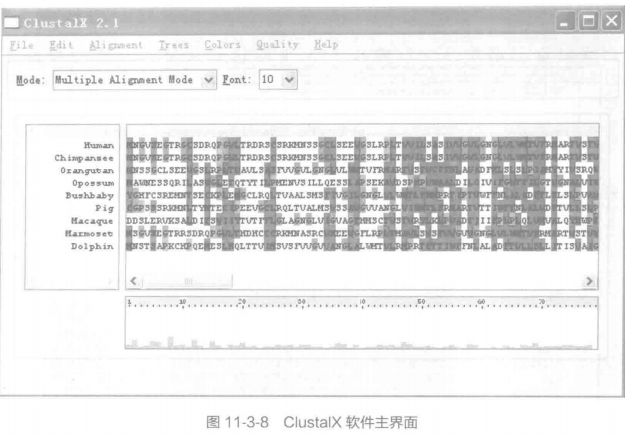
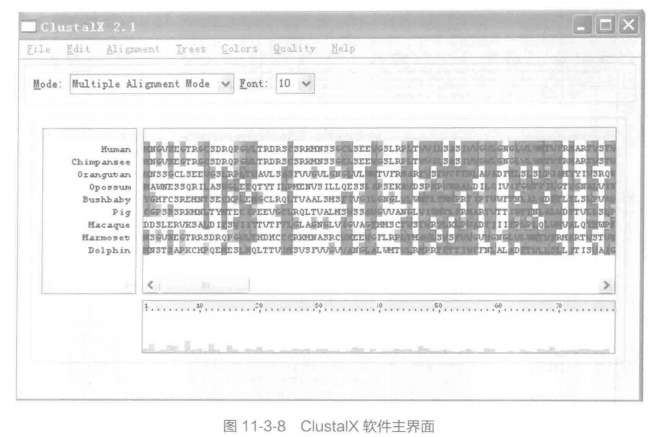
①将这些序列放置于 GPR32.fasta 中。主界面窗口如图 11-3-8 所示。
注意事项:输入序列的格式比较灵活,可以是 FASTA 格式,还可以是 PIR、SWISS-PROT、 GDE、Clustal、 GCG/MSF、RSF 等格式。
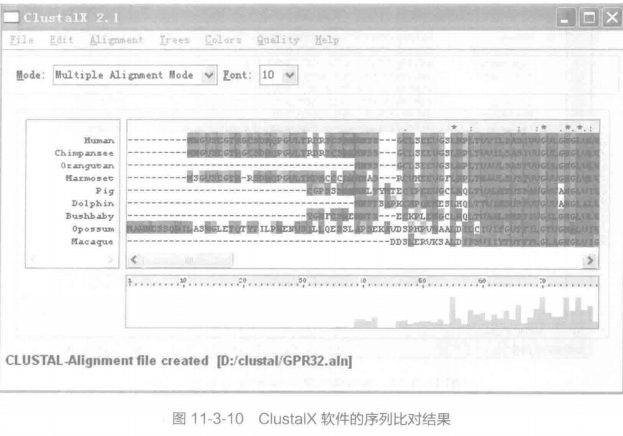
②对标尺上方的序列进行编辑操作。设定比对的一些参数。通常情况下,我们可以使用默认参数。比对参数主要有 6 个,分别是: Reset New Gaps before Alignment, Reset AlI Gaps before Alignment,Pairwise Alignment Parameters, Multiple Alignment Parameters,Protein Gap Parameters 和 Secondary structure Parameters。这里我们选择 Multiple Alignment Parameters 这一项,如图 11-3-9 所示。
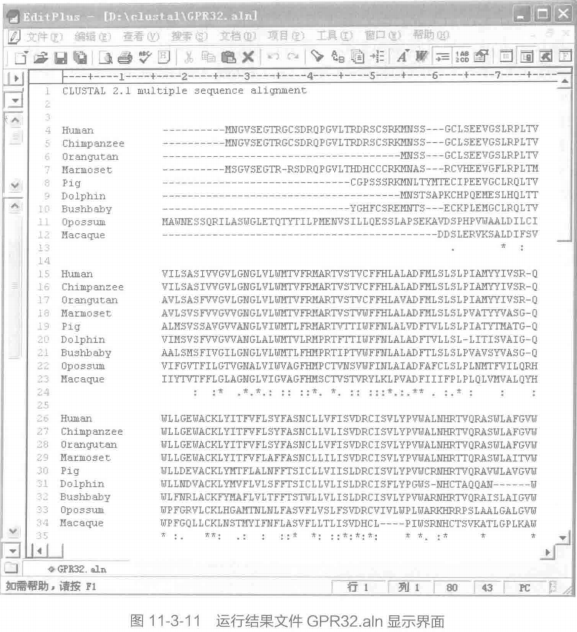
③返回菜单栏选择"DoComplete Alignment"标签,此时会弹出输出文件路径的设置窗口,设置 Output Guide Tree 和 Output Alignment Files 的保存路径,点击"OK" 按钮程序自动开始序列的完全比对, 比对所需时间因序列文件的大小、长度和计算机性能的不同而会有些差别,如图 11-3-10 所示。当左下方状态栏提示"CLUSTAL-Alignment File created [D : /clustal/GPR32.aln] "时说明序列此对已经完毕,这时在文件保存位置的目录下会生成两个文件,即 GPR32.aln 和 GPR32.dnd. 其中 aln 是序列此对的文件,可进-步用于构建系统发育树,而 dnd 是向导树文件。如图 11-3-11 和图 11-3-12 所示。

(三)Linus 环境下 ClustalW 的基本步骤
A. 进入 ClustalW 目录,按屏幕提示选择 1, 输入列文件,如图 11-3-14 所示。
B. 选择比对的形式,设置参数和输出文件的保存格式,按提示选择,然后开始比对。如图 11-3-15 所示。比对时会生成两个文件,即 GPR32.aln 和 GPR32.dnd, 其中 aln 是序列比对的文件,可进一步用于构建系统发育树,而 dnd 是向导树文件。
注意事项:我们也可以在 web 上使用 ClustalW, 比如: EBI 就提供了 ClustalW 的在线服务,网址为: http : //www.ebi.ac.uk/Tools/msa/clustalw2/。在线使用 ClustalW 是很简单的, 只要我们按照提示做相应的操作即
可完成。如图 11-3-16 所示。