基因家族分析之获取全部cDNA碱基序列构建进化树
生信技能树
今天让我们跟着教程,以趋化因子基因家族为背景来获取序列进行多序列比对后绘制系统发育树。
趋化因子背景知识
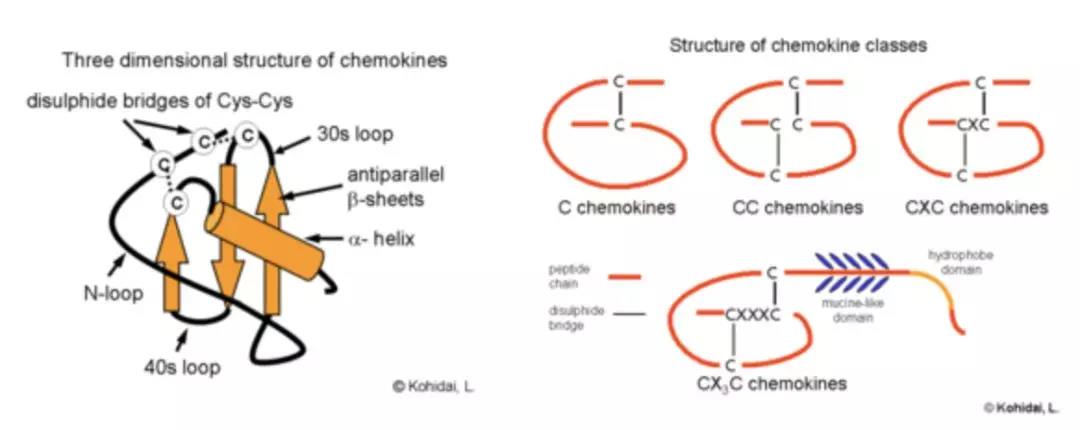
趋化因子被分为四个主要的亚家族:CXC,CC,CX3C 和 XC。所有这些蛋白质都通过与 G 蛋白质连接的跨膜受体(称为趋化因子受体)相互作用而发挥其生物学作用,该受体在其靶细胞表面被选择性地发现。
维基百科关于趋化因子介绍:https://zh.wikipedia.org/wiki/
参考文章:Charo IF, Ransohoff RM. The many roles of chemokines and chemokine receptors in inflammation. N Engl J Med. 2006 Feb 9;354(6):610 - 21.
也称做趋化激素、趋化素或是化学激素。
是一小分子细胞因子家族蛋白。趋化因子蛋白的共同结构特征包括,分子量小(约 8 - 10 千道尔顿),有四个位置保守的半胱氨酸残基以保证其三级结构。这些小蛋白因其有定向细胞趋化作用而得名。
当然,这些蛋白有些趋化因子历史上还有其他的名字,包括已知的 SIS 细胞因子家族、 SIG 细胞因子家族,SYC 细胞因子家族和血小板因子- 4 家族。
有的趋化因子被认为促进炎症反应,而有些趋化因子被认为在正常的修复过程或发育中控制细胞的迁徙。在所有脊椎动物和一些病毒和一些细菌中有趋化因子存在,但不存在于其他无脊椎动物。这些蛋白质结合到趋化因子受体而起作用,趋化因子受体是 G 蛋白偶联受体,选择性地表达在靶细胞表面。
当然,命名往往不是那么的规律,比如 IL- 8 是其中一个成员,在该基因家族中的 ID 是 CXCL8。
典型结构:
数据下载
1. 首先要拿到 CCL 基因家族的基因列表,可以在人类基因组命名委员会数据库下载:
https://www.genenames.org/data/genegroup/#!/group/ 483
2. 去 gencode 拿到数据库下载最新的 gtf 文件
https://www.gencodegenes.org/human/release_30.html
![]()
3. 去 ensemble 数据库下载人类的 cdna 的序列
![]()
家族成员一览表
前面我们介绍过趋化因子被分为四个主要的亚家族:CXC,CC,CX3C 和 XC,全部基因下载如下:
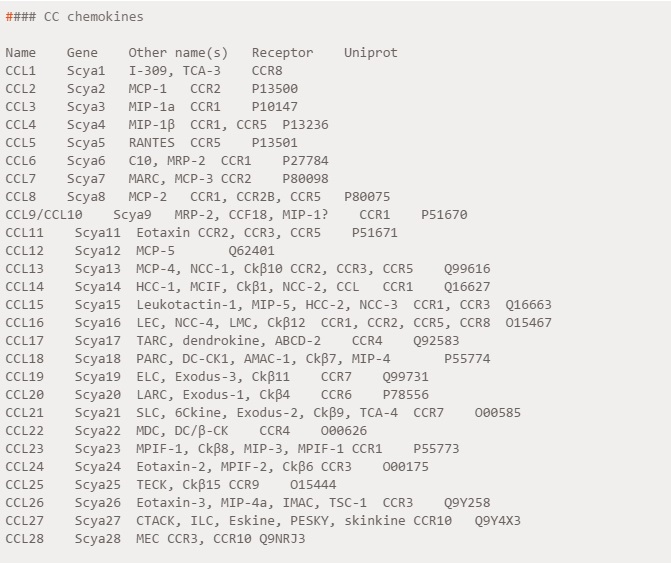

有了如上所述的基因信息,就可以根据我们从 gencode 下载的 gtf 文件进行。
获取 CCL 家族基因的转录本 id
在人类基因组命名委员会数据库下载拿到的是一个 csv 文件,打开文件,第二列 Approved symbol 就是 gene symbol id,总共有 45 个,保存到一个文件中,如:CCL_genes

然后从 gtf 文件中拿到 CCL 基因家族的所有转录本 id,共 124 个,以及对应的蛋白质 id(部分转录本没有蛋白 id)
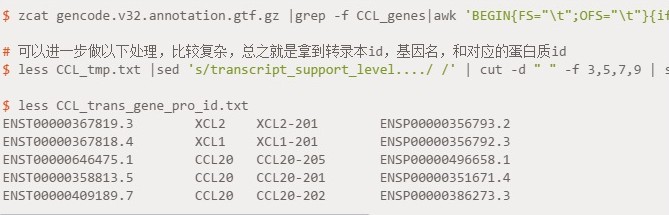
(PS:我觉得上面的 grep 脚本运行肯定会很慢,但是我懒得去修改它了。)
可以把转录本 id 单独保存为一个文件,比如我这里保存为 CCL_trans_id.txt

获取转录本的碱基序列
从人类 cdna 的 fasta 文件中提取提取上面转录本 id 的碱基序列这里需要用到一个 perl 单行命令:
![]()
使用起来其实很简单,需要两个输入文件,第一个是要获取的转录本 id 号,即上面的 CCL_trans_id.txt,第二个是人类的 Homo_sapiens.GRCh38.cdna.all.fa,结果输出就是 fasta 格式,可以保存为 CCL_trans_cdna.fa
转录本 id 转成 CCL
把 id 替换成 CCL,这样子可读性比较强一些,处理起来有点复杂,首先我们先把上面拿到的 CCL_trans_cdna.fa,每一条序列合为一行,然后用 awk 命令将处理后的结果与前面的 CCL_trans_gene_pro_id.txt 按指定行进行合并,再进行一定的处理,最后转成 fasta 格式,比较复杂,需要花较多的时间理解下面的代码:

做这么多操作就是为了一个目的:将 CCL_trans_cdna.fa 中的 id 转换成可读性较强的基因 id

拿到最后的 all_id.fa 文件后,就可以进行进化树分析了
绘制 CCL 基因家族的 cdna 进化树


可以看到,至少趋化因子的四个主要的亚家族:CXC,CC,CX3C 和 XC 是可以分开的。