CRISPR/Cas9 基因敲除实验 | 如何确定目标基因?
春卷00
今天主要的内容是准备工作:如何确定目标基因。
clear objectives 明确目标--敲除抑癌基因
首先,登录 Catalogue Of Somatic Mutations In cancer (COSMIC) Database
https://cancer.sanger.ac.uk/cosmic
其次,根据癌症类型查看肿瘤体细胞突变统计,并下载体细胞突变 list。
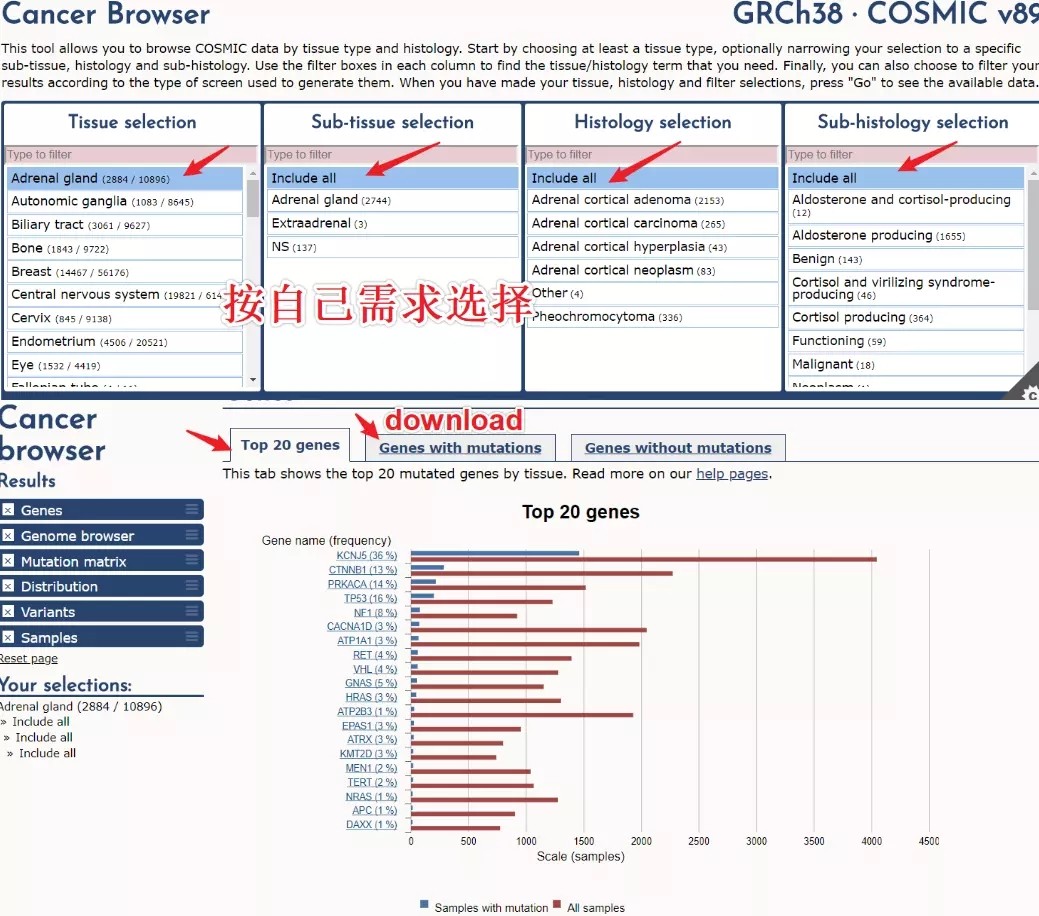
find and download
接着,找到tumor suppressor gene database
https://bioinfo.uth.edu/TSGene/
摸索并下载相应的肿瘤抑制基因数据 list
TSGs
同时,通过网页搜索,尽量查到更多关于 Tumor suppressor gene 相关信息
https://www.cellsignal.com/contents/resources-reference-tables/human-oncogenes-amp-tumor-suppressor-genes/science-tables-oncogene

CST with oncogene and tumor suppressor gene
再者,使用 R 语言将从不同 database 获取得到的 tumor suppressor gene 的 list 进行 overlap,再查阅 overlap 剩余基因在目的细胞中的表达水平--该部分依赖生物信息学技术,本部分结果省略。
接下来需要继续查阅文献,缩小范围得到更为精准的证据,有以下几点需要考虑:
1. 与要研究的疾病相同或者相似;
2. 与所用的材料相同或者相似;
3. 与要进行的方法相同或者相似;
4. 与研究目的相同或者相似;
5. 是否有直接的证据表明该基因相关。
在开始之前仍需要确认的信息
在大数据筛选+文献确认之后,就要开始着手敲除事项,在这之前有一个非常重要的点在很多教程中都没有提及,包括我之前写的 sgRNA 设计推文 CRISPR-Cas9 基因敲除 sgRNA 设计
常规情况下:
敲除所有 isoform 都有的基因片段,以达到全敲的效果;
在保证敲除所有 isoform 的基因片段之后,尽量敲往前一点,以免太过后面,导致后半部分虽然不完整,但仍有功能;
来自我的好友喜糖的提醒,作为大段敲除,最好是之前敲掉是 3 的倍数的外显子,以达到移码的效果,使得该位点之后的基因全部错位,以致表达缺失;
根据以上要点,我们不由的思考起来,我们敲除基因,仅仅是简单的敲除掉它的所有亚型的完整性吗?显然不是,我们需要的是,不仅仅敲除该基因表达,更重要的是摧毁该基因的功能!那么就设计到一个非常重要的知识点:基因功能域,所以我们需要通过敲除基因的功能域,从而得到 loss of function 的效果。
https://www.uniprot.org/uniprot/ 了解一下。
因此,在设计 sgRNA 之前,我需要搞清楚目的基因的结构域。
以 PTEN 为例
1. 无论如何,NCBI 在前面:
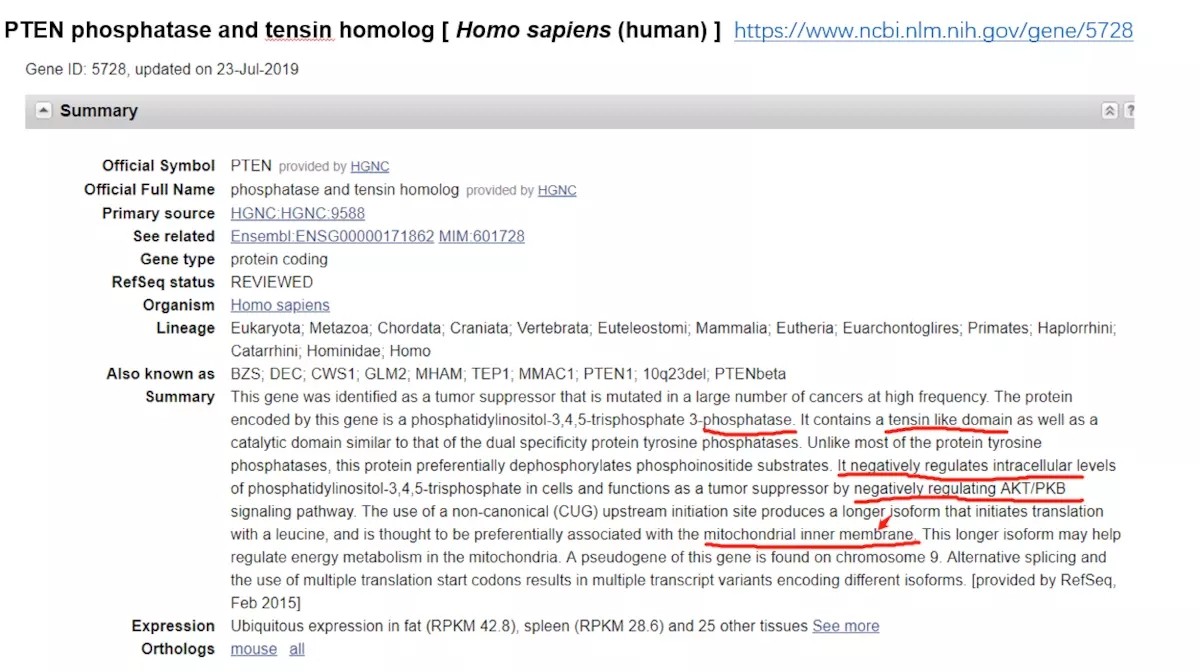
NCBI_PTEN
这边主要表明 PTEN 是一个带有类张力蛋白结构域的双特异性蛋白酪氨酸磷酶。和其他磷酸酶不一样(其他磷酸酶磷酸化蛋白后呈现激活状态),PTEN 磷酸酶催化底物磷酸化,从而负性调控细胞内磷脂酰肌醇- 3、4、5 -三磷酸腺苷(听着像一个信号传导用的什子),并且负性调控 AKT/PKB 信号通路,从而起到肿瘤抑制因子的作用。
其次,由非典型 (CUG) 上游起始位点的使用产生一个较长的同工异构体(PTEN-long 或者 PTENα),该异构体通过亮氨酸启动翻译,并且被认为优先与线粒体内膜相关联,这种较长的亚型可能有助于调节线粒体的能量代谢。该基因的一个假基因在 9 号染色体上被发现。选择性剪接和使用多个翻译起始密码子导致多个转录变体编码不同的亚型。
那么接着来看,PTEN 到底有多少个亚型,一则可以直接下载 PTEN 基因序列在 snapGene 软件查看,也可直接在该基因的 NCBI 页面上查看。值得注意的是,我们常常在谷歌学术上不停的搜索一个基因相关的文献,但很少注意到 NCBI 该基因页面下就已经总结好各种相关文献可以直接查阅。
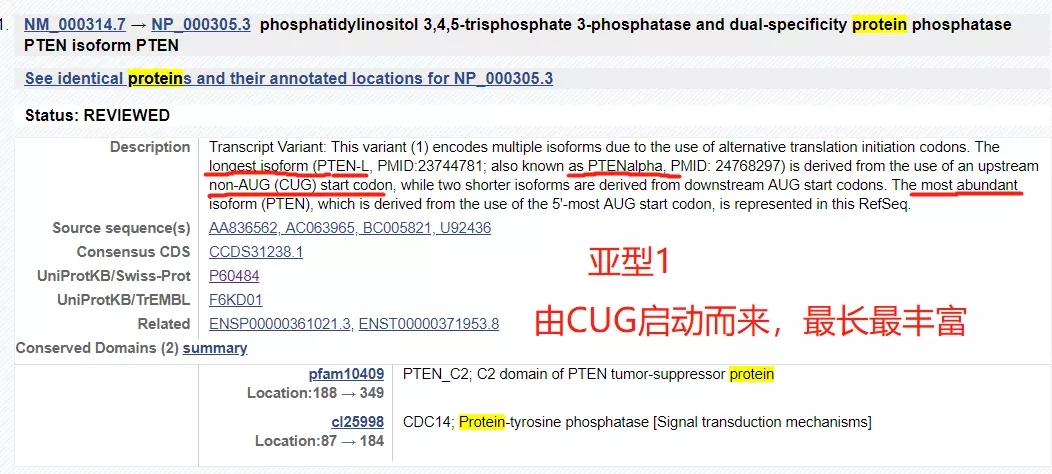
isoform1

2 and 3
2. 参考文献:

文章总结:

PTEN
3. 其他文献总结:
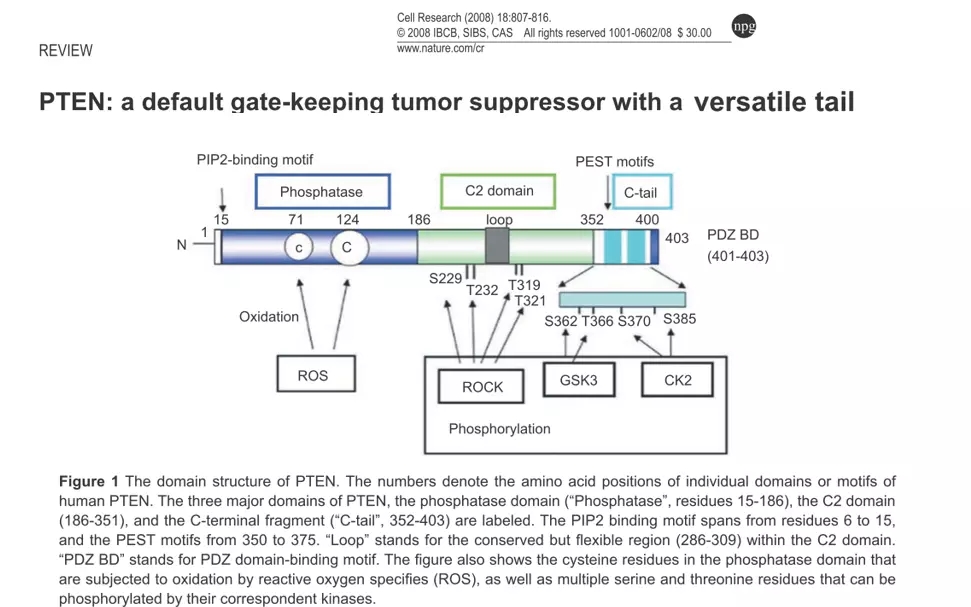
磨刀不误砍柴工
在这里我们简单介绍了如何寻找一个基因的功能域,那么根据各自的目的,去选择敲除不同的位置,从而构建合理的实验模型进行研究。如果一开始不够了解该基因的情况,胡乱选一个位点敲除,不仅结果不严谨,容易被人推翻,更是浪费自己的宝贵时间和课题组经费。以上仅仅是举个例子,具体细节仍需要自行把控。
选定位点之后,就可以进行下一步:sgRNA 和引物设计