相信做 qPCR 的研友经常会碰到以下令人抓狂的时刻:
三个重复的 Cqmax 和 Cqmin 能差到两个循环;
提的样本中总是含有很多 PCR 抑制剂,导致 Cq 值偏大;
极低丰度样本的 Cq 值总是游走在 40 和 N/A 边缘,让人崩溃;
绝对定量时需要的标准品既没有商品化,自己制备起来又很费时费力……
数不胜数的坑,等着有人来跳……
那真的束手无策了吗?
其实归结起来,qPCR 实验中碰到的难题无非是重复性差、扩增易受抑制、灵敏度低和需要标准曲线等,接下来介绍的 Digital PCR(数字 PCR)就可以完美的解决这些问题!
数字 PCR 是什么?
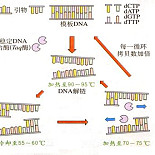
数字 PCR 是一种新型的核酸(DNA、cDNA 或 RNA)定量技术,它采用有限稀释的方法,使用泊松分布分析数据从而得到靶标的绝对拷贝数浓度。
数字 PCR 实验中,一个完整的 qPCR 反应体系(包括模板、DNA 聚合酶、引物、探针等)被分隔为若干个(通常数万个到几百万个)独立的反应区间(纳升级体积),经过 PCR 扩增后,包裹有靶标 DNA 的反应区间会发出荧光信号,标记为阳性;反之为阴性。
最后根据阴性反应区间的比例经过泊松分布计算得到靶标 DNA 的拷贝数浓度。
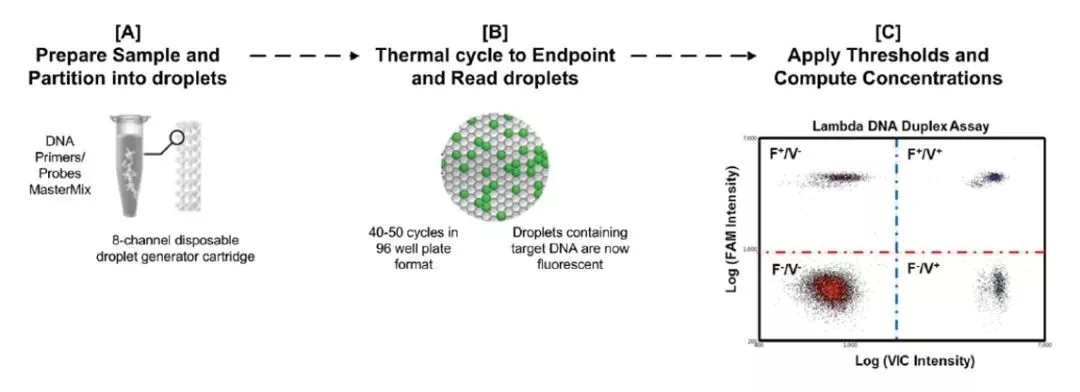
图片来源:Analytical chemistry
其实数字 PCR 不算是新技术了,它的原型可以追溯到 1992 年。Sykes 等报道了基于样品有限稀释和泊松分布数据处理的巢式 PCR 定量技术,并提出了数字 PCR 的构想 [1]。
1999 年,Vogelstein 和 Kinzler 采用 96 孔板系统发展了微升级的 PCR 扩增方法,用于结肠癌的突变基因 ras 的定量分析 [2],使数字 PCR 的应用实践成为了可能。
后来芯片蚀刻技术和微流控技术的发展,使得数字 PCR 真正有了用武之地。
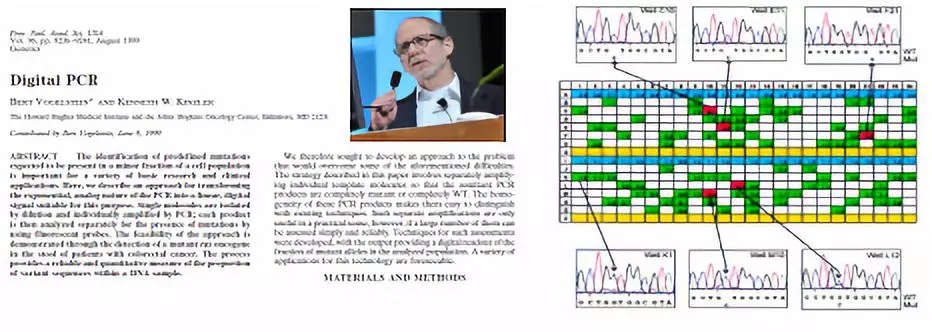
图片来源:文章截图
数字 PCR 的优势
相较于 qPCR,数字 PCR 有以下优势:
真正的绝对定量,无需标准品和标准曲线;
不再像 qPCR 那样严重依赖于扩增效率,对 PCR 抑制耐受程度高,模板适用度广;
更高的分辨率,能够区分 1.1 倍的浓度差异,适合高拷贝数的 CNV(16 copy vs 18 copy)实验;
更高的灵敏度,可以检测到单拷贝的靶标,适合单细胞基因表达等研究。
重复性好,intra-assay 和 inter-assay 的 CV 较 qPCR 低 [3]。
数字 PCR 实验的操作
数字 PCR 的实验无需太过复杂的操作,但需要相关设备的辅助才可以实现。数字 PCR 从 partition 制备方式上分为芯片式和微滴式,操作大同小异,一般分为以下几步:
配置 Reaction Mix。包含引物、荧光探针或者染料、模板和 DNA 聚合酶,不同厂家对体积要求也不一,一般 15 - 25 μl。
Partition 分隔。通过芯片或者微流控设备将配好的体系处理成 10000 - 30000 个 partition,不同的厂家其 partition 的数量也不同。
PCR 扩增。使用普通的 PCR 循环仪或者平板扩增系统将生成的 partition 进行扩增。
读取数据。使用 CCD 拍照方式或者流式细胞术技术对 partition 进行信号读取,最后软件根据阴性的 partition 比例计算得到靶标的浓度,单位是 copy/μl。
数字 PCR 的应用
目前数字 PCR 的应用已经非常成熟,通俗的讲,qPCR 能做的,数字 PCR 轻松加愉快,qPCR 做不到的,数字 PCR 也可以胜任。
SNP 方面,与疾病的分子标志物检测相关的文献多如过江之鲫,尤其是非小细胞肺癌相关的 EGFR 突变。Oxnard 等使用数字 PCR 技术监测血浆中 EGFR 敏感突变和耐药性突变(T790M)[4]。
基因表达方面,已经深入到单细胞的水平。Albayrak 等使用数字 PCR 研究单细胞基因的转录组丰度和蛋白组丰度的相关性 [5],其中用到的邻位连接技术 PLA 很有新意,将低丰度蛋白的检测转化为相对容易的 DNA 检测。
CNV 方面,数字 PCR 可以用于检测皮肤中体细胞的拷贝数嵌合现象 [6]。
当然也有其他方向的各种新奇应用,比如四种不同 RNA 剪接体的同时检测 [7],将 T4 单细胞包进 partition 中进行 HIV 病毒库的检测 [8] 等。
套用句过时了的话:只有你想不到的,没有数字做不到的。
如果小伙伴们对数字 PCR 感兴趣的话,接下来的几期,笔者会 focus 在数字 PCR 的实验设计方面,希望能够帮助大家加深对这一技术的理解。
参考文献:
1.Sykes, P. J., et al. "Quantitation of targets for PCR by use of limiting dilution." Biotechniques 13.3 (1992): 444 - 449.
2.Vogelstein, Bert, and Kenneth W. Kinzler. "Digital pcr." Proceedings of the National Academy of Sciences 96.16 (1999): 9236 - 9241.
3.Bharuthram, Avani, et al. "Comparison of a quantitative Real-Time PCR assay and droplet digital PCR for copy number analysis of the CCL4L genes." Infection, Genetics and Evolution 25 (2014): 28 - 35.
4.Oxnard, Geoffrey R., et al. "Association between plasma genotyping and outcomes of treatment with osimertinib (AZD9291) in advanced non–small-cell lung cancer." Journal of clinical oncology 34.28 (2016): 3375.
5.Albayrak, Cem, et al. "Digital quantification of proteins and mRNA in single mammalian cells." Molecular cell 61.6 (2016): 914 - 924.
6.Abyzov, Alexej, et al. "Somatic copy number mosaicism in human skin revealed by induced pluripotent stem cells." Nature 492.7429 (2012): 438.
7.Sun, Bing, and Yun-Ling Zheng. "Simultaneous quantification of multiple alternatively spliced mRNA transcripts using droplet digital PCR." Digital PCR. Humana Press, New York, NY, 2018. 387 - 400.
8.Yucha, Robert W., et al. "High-throughput characterization of HIV- 1 reservoir reactivation using a single-cell-in-droplet PCR assay." EBioMedicine 20 (2017): 217 - 229.