在线生信分析平台如何进行差异表达分析?—GeneMatrix实战教程
上海吉凯基因医学科技股份有限公司
随着高通量测序和质谱技术的普及,组学实验已成为每个课题组不可或缺的研究手段。然而组学实验产出的数据往往十分庞大,处理这些数据也需要专业的生物信息学和统计学知识,这令很多研究者望而却步。
为了解决这一难点,吉凯基因建设了在线生信分析平台GeneMatrix(点击进入)。GeneMatrix功能强大,包含临床数据分析、组学数据分析和基础统计分析三大模块,共43个应用程序。每个小程序都配备了详尽的使用说明和示例文件,文件上传、下载和参数设置均可通过点击按键实现,界面友好,使用方便,没有生物信息学和编程背景也可使用。运行的输出文件和图片可以存放在私有云盘内,保证数据安全的同时方便反复调用。更重要的是,目前GeneMatrix完全免费开放,只需完成简单的注册步骤,即可享受完整功能。今天,小编就以一个具体实例,为大家讲解如何利用GeneMatrix进行差异表达分析,为您讲解以下技巧:
-
使用GeneMatrix进行差异表达分析
-
使用GeneMatrix绘制火山图
数据准备
我们使用的数据集来自Keith Anderson等人于2011年发表的对于前列腺癌雄激素依赖基因特征的研究(GSE22606)。该实验采用双因素析因设计,分别使用人造雄激素R1881诱导和iRNA介导SRF基因沉默的方式对前列腺细胞系LNCaP进行处理,研究其与空白溶剂对照样本间的基因表达差异,每组设置了三个生物学重复。所采用的实验平台为Affymetrix人类表达谱芯片Human Genome U133 Plus 2.0 Array。为简化分析流程,本教程仅关注R1881诱导组与空白溶剂对照组间的差异,点击下方链接可获取示例数据。
差异基因表达分析
差异表达分析的目的是确定哪些基因在不同条件下的表达水平存在差异。通常我们需要两个输入文件:1. 样本表型数据(samples.tsv);2. 基因表达矩阵,也称为计数数据(counts.tsv)。
在开始分析之前,我们有必要先观察一下数据:
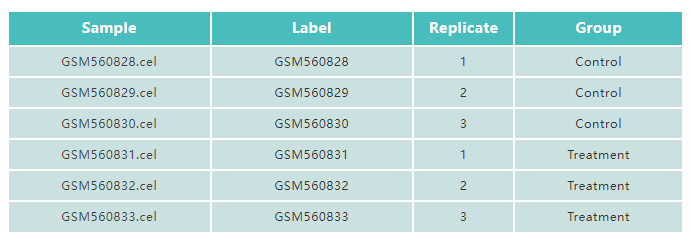
表1
表1显示的是样本表型数据,文件格式是以TAB分隔的文本表格,文件的第一列为样本名;第二列为标签名,用于对样本注释;第三列为样本顺序号,在成对分析中,作为成对样本的标识;第四列为分组名,用于差异比较分组。
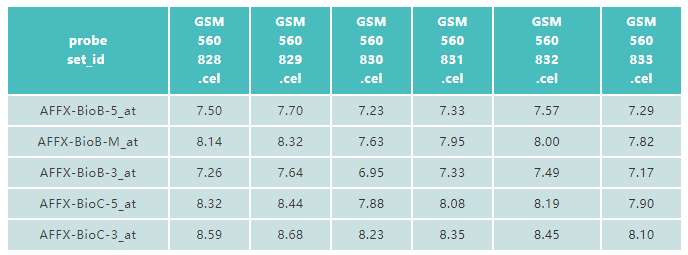
表2
表2显示的是计数数据(前5条记录),文件格式同样是以TAB分隔的数据框,文件的列名称为样本名,必须与样本表型数据中的样本名相对应;行名称为特征名,可以是芯片ID、基因名等标识;数据矩阵为基因表达计数。需要注意的是,这里的基因表达计数是经过归一化(normalization)处理并保存为以2为底的对数形式。归一化的原因和方法这里不展开介绍,为了启发思考,大家可以尝试观察一下每一个样本的基因表达计数的分布形态、总和以及均值,并比较这些度量在各样本中的异同。
在准备好上述文件后,我们便可打开GeneMatrix,在应用市场中搜索差异分析并进入,点击立即应用,输入自定义项目名称后点击创建,在样本文件选项选择上传samples.tsv文件, 原始数据选项选择上传counts.tsv文件。输出文件名称可以自定义,这里设置为changes.txt。在完成输入文件和输出文件的设置后,我们还需要设置额外的参数。在显示更多参数的下拉菜单中,我们设置试验组为Treatment,对照组为Control,注意这里设置的值必须与样本表型数据中的分组名相对应。
比较方法即为差异比较中的方差建模策略,这里推荐选择limma。有研究表明,与t检验相比,limma在功效(power)和假阳性率(false-positive rate)方面有显著提升,特别是处理小样本量的情况[1]。本实验未采用配对设计,因此将成对分析设置为否。为了获得完整的数据输出用于后续绘制火山图,在此步骤中,差异倍数、显著性水平和错误发现率(FDR)的阈值都设置为不筛选,后续我们可以通过Python Pandas表格处理设置更精细的过滤标准。在设置完全部的参数后,我们点击提交按钮。
等待片刻后,即可在我的项目中查看(点击放大镜图表)运行结果。如需将数据保存到本地,请点击下载,但更推荐的做法是将结果存入云空间,以便接下来作为其他应用程序的输入文件。
让我们来查看一下输出结果changes.txt(表3):
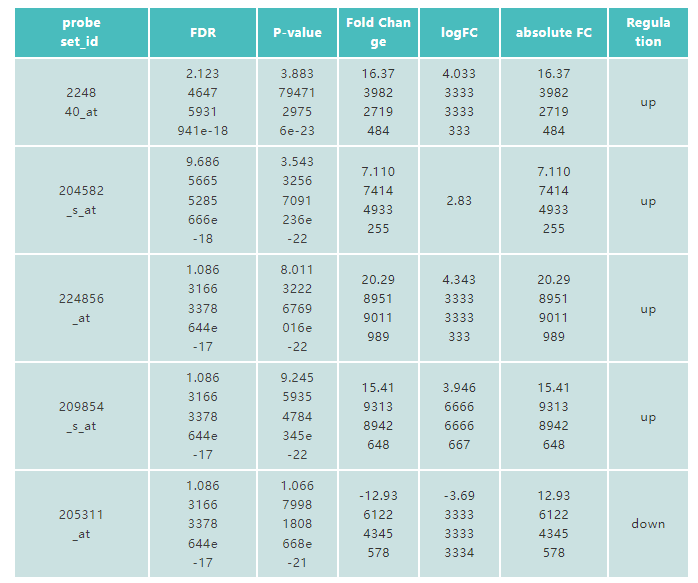
表3
除了芯片ID以外,我们得到了一些新的统计量。通常,我们在P-value上使用0.05的阈值来定义显著的表达量改变。FDR是一个调整后的P-value,也称为q-value,用于减少多重检验(差异表达分析通常涉及对上千甚至上万的基因的检验)引入的假阳性,因此FDR的置信度比P-value更高。Fold Change表示与对照组相比处理组中的基因上调或下调的倍数。而logFC和absolute FC分别为Fold Change对数形式和绝对值形式。Regulation则显示了上调或下调的状态。在差异表达分析中,最受关注的统计量是FDR和logFC,对其设置阈值并过滤,将会得到我们关心的表达量发生显著变化的基因(DEG)。
火山图绘制
在上一步骤中,我们得到了表达量变化数据changes.txt,我们使用它作为绘制火山图的输入文件。在GeneMatrix应用市场中搜索火山图并进入,选择输入文件和定义输出文件名为volcanoPlot.png。在显示更多参数的下拉菜单中,我们设置显著性水平为0.05,差异倍数为2,差异数据类型为差异倍数对数值。同时不要忘记根据实际情况更改显著性数据列和差异倍数列,这里我们选用FDR和logFC的列索引分别为2和5。其他参数保持默认,并点击递交,稍适等待后即可查看结果。结果如图1所示,红色高亮的点即代表显著上调或下调的基因。
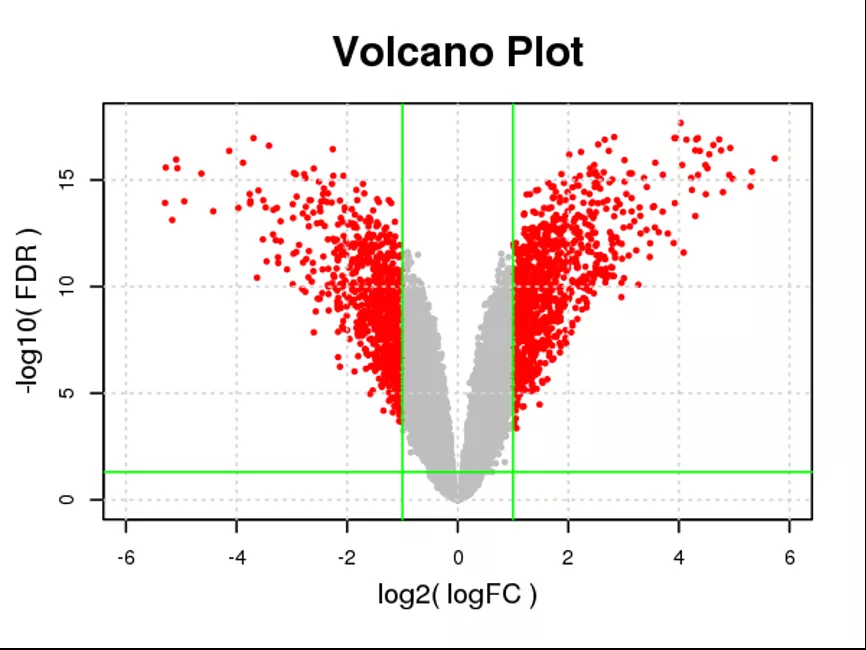
图 1
至此,我们介绍了使用GeneMatrix进行差异表达分析和绘制火山图。除此之外,GeneMatrix还有更多的功能等待您探索,我们也将在后续的教程中对其进行介绍。
【参考文献】
1.Jeanmougin, M., de Reynies, A., Marisa, L., Paccard, C., Nuel, G., & Guedj, M. (2010). Should we abandon the t-Test in the analysis of gene expression microarray data: A comparison of variance modeling strategies. PLoS ONE, 5(9), 1–9. https://doi.org/10.1371/journal.pone.0012336