施一公团队又出新作!进一步解析人类 U2 snRNP 组装过程,提供癌症突变机制新见解
丁香学术
导读
pre-mRNA 的剪接是由一种被称为剪接体的大而动态的 RNA-蛋白质复合物执行的。剪接体由 5 个小的核糖核蛋白颗粒(snRNPs,包括 U1、U2、U4、U5 和 U6)和非 snRNP 因子组装而成。每个 snRNP 由一个单独的小核 RNA (snRNA),7 个常见的 Sm 蛋白和一些特定的蛋白质因子构建。在这 5 个 snRNP 中,U2 snRNP 在内含子的识别和前体折叠的组装过程中起着重要作用。
人类的 U2 snRNP 尤其复杂,它的组装是一个多步骤的过程,并且人们对此知之甚少。17S U2 被认为是直接参与早期剪接体组装的人类 U2 snRNP 的功能形式。17S U2 snRNP 的核心成分包括 SF3b 配合物、SF3a 配合物、12S U2 核心、剪接因子 TAT-SF1 和 DDX46。体外实验也表明,SF3b 与 12S U2 核依次组装,形成一种被称为 15S 粒子的中间产物,最终形成 SF3a 复合物。然而,对组装这一过程的蛋白质因子却知之甚少。
最新的研究表明,具有 RNA 伴侣活性的 DDX42 可能是在 U2 snRNP 组装完成后释放的。而与 DDX42 相反,DDX46 是 17S U2 的一个组成部分,在剪接前体的组装和分支位点的校对过程中发挥着重要作用。但 DDX46 是否在 U2 snRNP 组装中有额外的作用尚不清楚。此外,虽然 DHX15 在内含子-套索剪接体 (ILS) 复合体分解中的机制已被广泛了解,但其在 U2 snRNP 和早期剪接复合体中的作用仍不清晰。
要解决这些关键问题,就必须阐明这些 RNA 解旋酶的结构和功能。最近的研究既揭示了 17S U2 snRNP 的整体结构,也揭示了 SF3B1 与 DDX46 之间的详细相互作用,但 DDX42 和 DHX15 在早期剪接复合体中的结构信息仍然缺乏。
2023 年 2 月 17 日,西湖大学施一公及张晓峰团队在 Nature Communications 杂志发表了题为 Mechanisms of the RNA helicases DDX42 and DDX46 in human U2 snRNP assembly 的文章,报道了 DDX42-SF3b 配合物的高分辨率结构和含有 DDX42 的 17S U2 snRNP 的假定组装中间体 (以下简称 DDX42-U2 配合物)。此外,他们还分离出一种含有 DHX15 的 U2 snRNP。
他们的研究结果揭示了 SF3B1 与 DDX42、DDX46 和 pre-mRNA 的多嘧啶束 (polypyrimidine tract, PPT) 相互作用的共同模式。结合生化分析,他们还揭示了 DDX42 和 DDX46 在 U2 snRNP 组装中的作用,并为 SF3B1 突变导致的癌症提供了新的见解。

图片来源:Nature Communications
主要研究结果
样品制备及结构测定
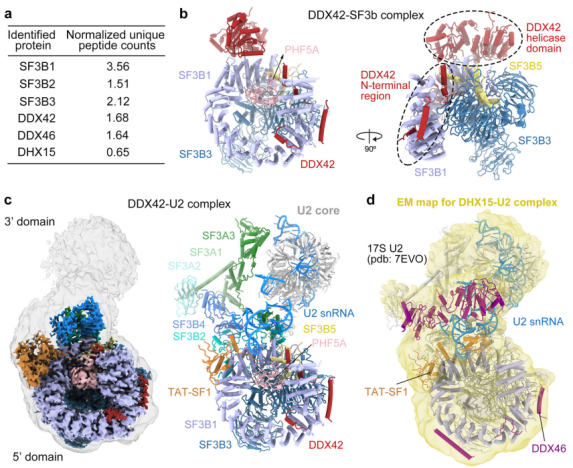
首先,研究人员纯化了 DDX42-SF3b 复合物以及含有 DDX42 的潜在剪接体复合体。在 DDX42 的 N 端融合了一个 Flag 标记,并利用该标记对内源性复合体进行亲和选择。纯化后的样品在交联条件下进行梯度离心。最终样本只包含 5 种 snRNA 中的 U2。经过 2D 和 3D 分析,人类 DDX42-U2 复合体最终被重建,平均分辨率为 2.7 Å。
使用类似的策略,他们在 DHX15 上使用 Flag 标签拉下内生剪接复合物。经过亲和选择和梯度离心,他们分离出一个包含 DHX15、U2 snRNA 和 17S U2 snRNP 的大部分蛋白质成分的大分子复合物 (称为 DHX15-U2 复合物),该复合物显示了与 17S U2 snRNP 相似的结构特征。
图片来源:Nature Communications
DDX42 锚定在 SF3b 复合物上
在 DDX42-SF3b 复合物中,SF3B1、SF3B3、SF3B5 和 PHF5A 的构象与游离 SF3b 配合物的构象几乎相同。DDX42 结合在 SF3b 核心的外围,并主要通过其 N 端扩展序列锚定在核心上。与这些序列基序相反,DDX42 的解旋酶结构域由两个 RecA 结构域组成,松散地连接在 SF3B1HEAT 的 C 端。
DDX42- SF3b 接口最显著的特点是将 DDX42 N-plug 放置在 SF3B1 的表面凹槽区域中,来自 N-plug 的带负电荷的氨基酸与来自 HR4-HR9 的带正电荷的残基形成了氢键 (H 键) 网络。与 N-plug 相反,DDX42 的 a−3 螺旋通过 SF3B1 的 HR5 与 HR3 的外部结合。
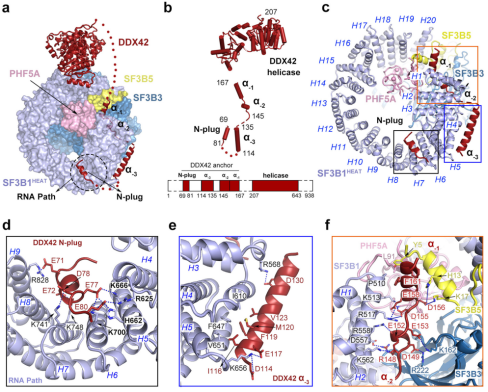
图片来源:Nature Communications
DDX42-U2 复合物的整体结构
DDX42-U2 复合物的整体结构与 17S U2 snRNP 相似,它们都表现出二级结构,3' 结合域灵活地连接到 5' 端。与 17S U2 相比,DDX42-U2 复合体中 SF3b、SF3a 和 U2 snRNA 的构象几乎相同。此外,TAT-SF1 的 UHM 结构域与 U2 snRNA 的 Linker 结构域在 DDX42-U2 复合物的结构中缺失。
与 DDX42-SF3b 配合物相比,DDX42 的 N 端序列,包括 N-plug 和螺旋结构,仍然与 SF3B1 结合,但解旋酶结构域已从 SF3B1HEAT 的 c 端移位。因此 DDX42 解旋酶结构域在 DDX42- U2 复合物中的确切位置尚不清楚。
DDX42-SF3b 与 DDX42-U2 的结构叠加显示了 DDX42 的解旋酶结构域与 U2 snRNA 之间的重叠位置。此外,DDX42 的解旋酶结构域也与 TAT-SF1 的 Linker 结构域重叠,这有助于维持稳定构象。TAT-SF1 的连接子结构域主要与 SF3B1HEAT 的 HR16 和 HR17 残基相互作用,这些相互作用可能在 DDX42 的解旋酶结构域从 SF3B1 取代的过程中起着至关重要的作用。
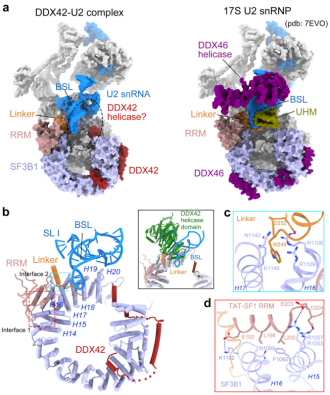
图片来源:Nature Communications
DDX42 和 DDX46 竞争性结合 SF3b
将 DDX42- SF3b 和 DDX42-U2 与先前报道的 17S U223 和组装剪接体进行结构比较,发现 SF3B1 的 RNA 路径依次被三个结构基序占据:DDX42- SF3b 和 DDX42-U2 中 DDX42 的 N-端、17S U2 中 DDX46 的酸性环以及组装剪接体中 pre-mRNA 的聚嘧啶束。此外,DDX42 和 DDX46 的 N 端序列在 SF3B1 的凸面上共享额外的锚定位点。因此,SF3B1 释放 DDX42 是 17S U2 snRNP 成熟期间 DDX46 募集的先决条件。从这个意义上说,DDX42 和 DDX46 都充当 RNA 路径的伴侣,并保留其参与下一个相互作用伴侣的功能。
为了促进对 DDX42 和 DDX46 的机制理解,他们使用生物层干涉术定量测量了它们与 SF3b 复合物的相互作用。DDX42 和 DDX46 对 SF3b 配合物分别表现出 23 nM 和 66 nM 的明显结合亲和力。由于 DDX42 和 DDX46 都占据了 SF3B1 的 RNA 通路,因此预测它们在与 SF3b 的结合方面具有竞争力。当大量过量供应 DDX46 时,DDX42 从预组装的 DDX46-SF3b 复合物中取代 DDX46,形成 DDX42-SF3b 复合物。类似地,大量过量的 DDX46 也能够从 DDX42-SF3b 复合物中取代 DDX42,这种竞争性结合可能最终促进 17S U2 snRNP 的成熟。
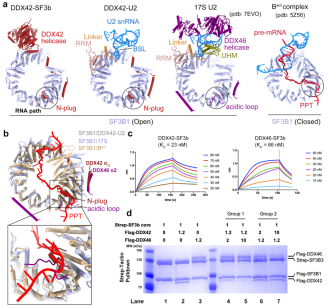
图片来源:Nature Communications
结语
综上所述,在这项研究中,他们发现 DDX42 与 SF3b 自发结合,形成 DDX42-SF3b 复合物,然后招募 12S U2 核心粒子和 SF3a 组装 DDX42-U2 配合物。虽然 DDX42 活性的靶点尚不清楚,但他们得到的结构数据表明,DDX42 可能在 U2 snRNA 在 SF3B1 上的调节过程中发挥重要作用。
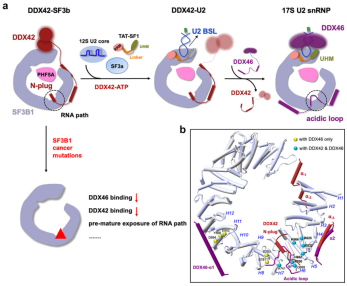
图片来源:Nature Communications
SF3B1 的体细胞突变导致调控异常的 RNA 剪接,并在多种癌症中观察到。与 DDX42 或 DDX46 相互作用的 SF3B1 残基被分为三类:仅与 DDX42 相互作用,仅与 DDX46 相互作用,以及与两者同时作用。有趣的是,仅与 DDX42 相互作用的 SF3B1 残基不会受到癌症衍生突变的影响;与 DDX42 相反,仅与 DDX46 相互作用的三个 SF3B1 残基 (Asp894, Tyr898 和 Glu902) 在癌症中发生突变。此外,与 DDX46 和 DDX42 相互作用的 7 个 SF3B1 残基是癌症突变的靶点,包括最常见的突变残基 Lys700。因此,本研究的结果为 SF3B1 突变所致癌症的机制提供了新的见解。
题图来源:站酷海洛
参考文献:
1. Yang, F., Bian, T., Zhan, X. et al. Mechanisms of the RNA helicases DDX42 and DDX46 in human U2 snRNP assembly. Nat Commun 14, 897 (2023).
2. Tholen, J., Razew, M., Weis, F. & Galej, W. P. Structural basis of branch site recognition by the human spliceosome. Science 375, 50–57 (2022).
3. Zhang, Z. et al. Structural insights into how Prp5 proofreads the pre-mRNA branch site. Nature 596, 296–300 (2021).