【操作步骤】如何从零开始拆解一篇肿瘤生信文章,并化为己用
科研论文时间
今天,笔者就以一篇 SCI 文章为例,带各位小可爱看一看怎么才能更快 GET 一篇近期发表的生信分析文章中主要讲了些什么,以及我们该如何学习大佬的思路并化为己用。
今天,用来举例的这篇文章是发表在 International Journal of Molecular Sciences 的A Comprehensive Bioinformatics Analysis of UBE2C in Cancers(DOI:10.3390/ijms20092228),影响因子在 4.5,是纯生信文章中还不错的分数辣。
至于为什么又是肿瘤?别问,问就是肿瘤撑起了生信分析文章的四分之三壁江山。
当然,在读文章和读图之前我们还是要对文章进行一个概念化的认识。通过大致阅读摘要,我们可以获知:
UBE2C 是一种泛素化的共轭酶,作者通过两个数据库——TCGA 和 GTEx,发现这个酶的表达和肿瘤的不良预后相关,并且筛选出了一些之前不太有研究的、和 UBE2C 相关的一些基因靶点。
在这里笔者提出一个小建议,各位小可爱不妨在读完摘要后,代入自身视角先思考一下:
如果是你们进行这项研究,会通过哪几部分结果,用什么样的分析方法来验证并且得出摘要中所提出的结论。然后,再带着思路去阅读文章,对比作者是怎么样证明的。
这个小技巧非常有助于科研思维和逻辑的培养,对以后做实验和写文章都很有帮助。
比如这篇文章,我们至少可以提出以下三个推断:
1)UBE2C 在肿瘤中存在差异性表达(表达量统计学分析);
2)UBE2C 的差异性表达会影响肿瘤的预后(生存分析);
3)与 UBE2C 表达变化的上下游相关基因和他们的生物过程(蛋白互作分析,富集分析);
那么,接下来,我们就带着自己的思路,速读一下这篇文章。(文章原文在「科研论文时间」公众号后台回复「1019」即可获取全文)
首先,我们先过一遍文章的结果部分的题目,然后再重点解读一下文章结果中的五张组图和几张表格。
结果一:
果不其然,是 UBE2C 在肿瘤的表达水平
这一部分结果包含了 2 张图和一张表格。
(1)图 1:乍一看上去就非常的漂亮,不同线条显示的是 27 组不同肿瘤(红)和正常对照(绿)组织内 UBE2C 的基因表达水平,黑色横线代表均值。
本质来讲仍然是一张绘制上比较简单的散点图,比较容易理解。
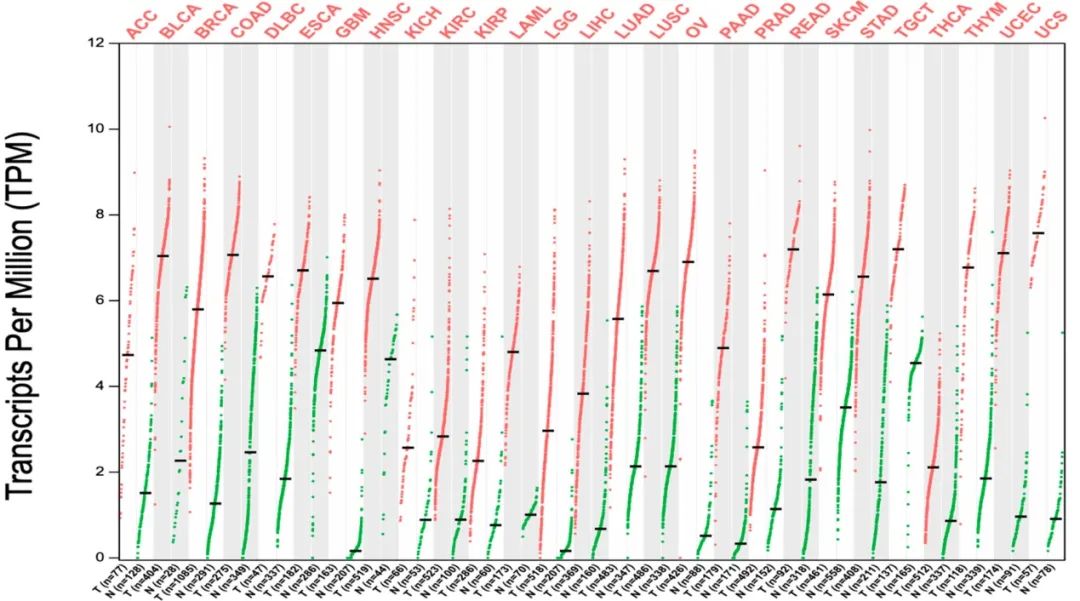
图 1(来源:文献图 1)
(2)表 1:这里作者进一步选择了 16 种肿瘤类型,在「组织学分型」,「分子分型」、「肿瘤分级」和「其他患者状态」这四个层面。
对比「正常」和「患者」的 UBE2C 表达,然后将所有具有统计学显著性的 P 值结果列在了表 1 中。
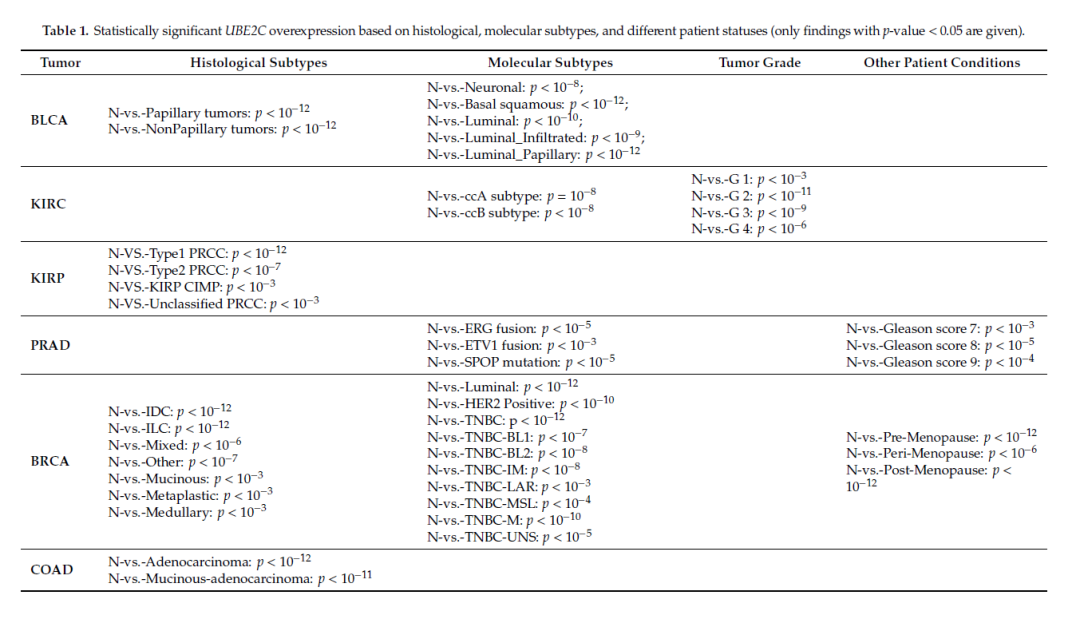
图 2(来源:文献表 1 的部分截图)
(3)图 2:图 2 是比较好理解的箱形图,这一部分作者在肿瘤的病理分期层面更细致的对比了正常和不同肿瘤病理期,以及各期中 UBE2C 的表达差异。
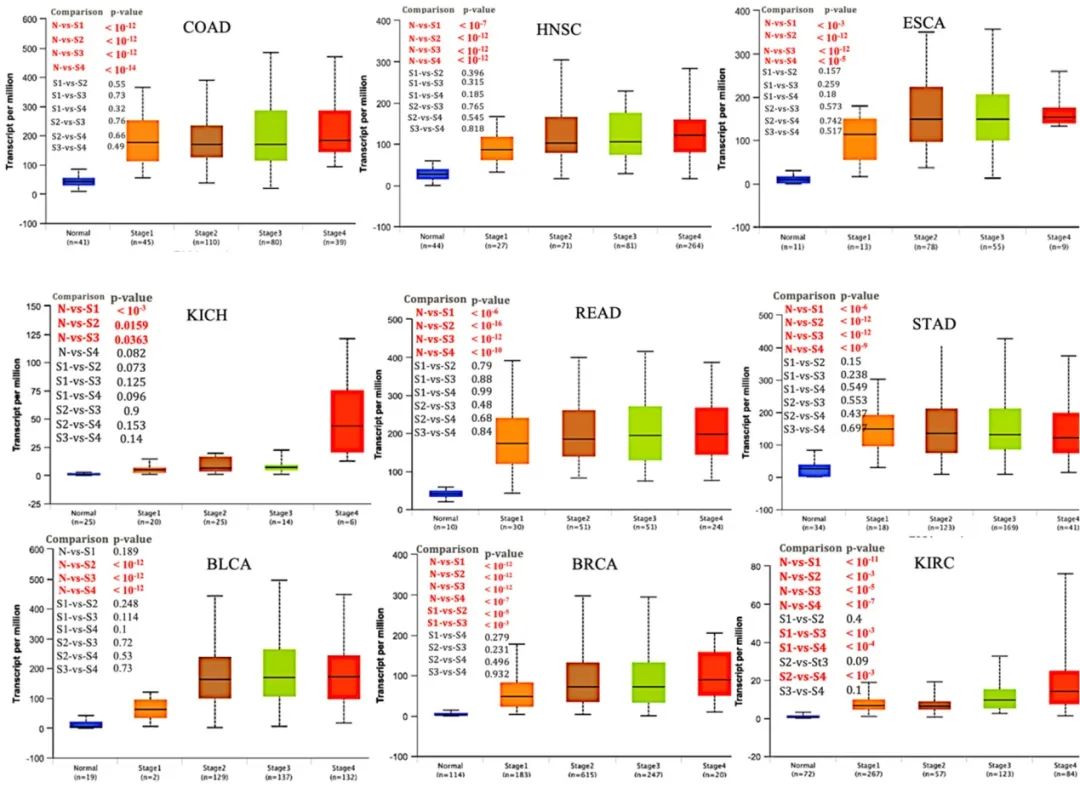
图 3(来源:文献图 2 的部分截图)
结果二:UBE2C 过表达在肿瘤预后中的作用
这一部分结果包含了 2 张图。
图 3,图 4:还是熟悉的曲线,还是熟悉的味道,是生存曲线没错了。
这两张图完全可以放在一起阅读。只需要读最上面的 x 轴标题即可,图 3 是总生存期,图 4 是无病生存期。
蓝色曲线是 UBE2C 表达较低的组,红色曲线是 UBE2C 表达较高的组。同样是曲线越陡峭,代表较低的生存率或较短生存期,从下面两张图中不难读出红色曲线明显陡峭一些。
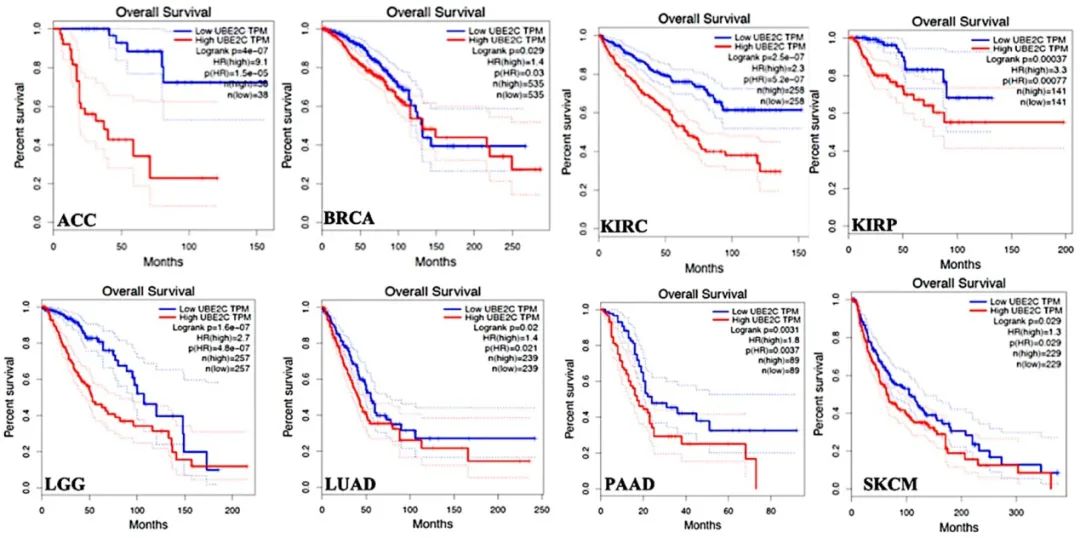
图 4(来源:文献图 3)
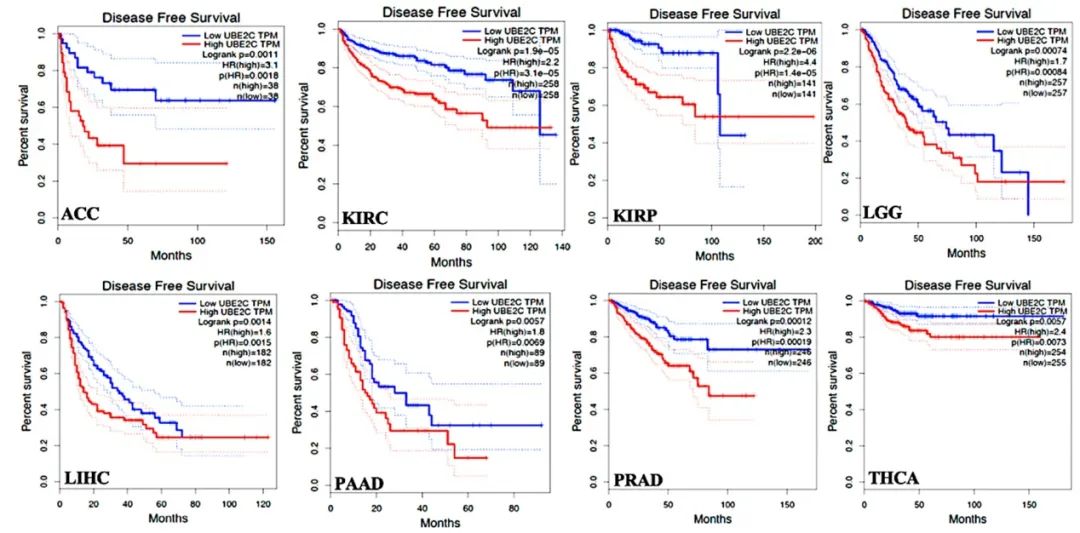
图 5(来源:文献图 4)
结果三:UBE2C与其他肿瘤基因表达的相关性
这一部分结果包含了 2 张表格。
(1)表 2:这里作者罗列了一堆与 UBE2C 表达正相关和负相关的基因,以及他们的结合位点。
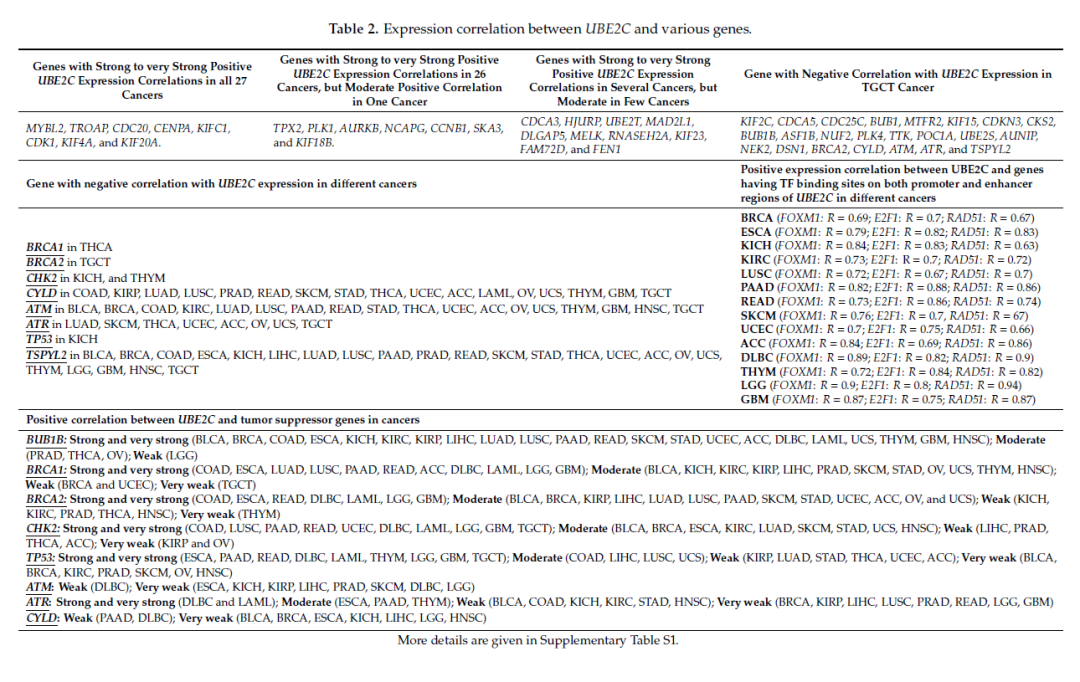
图 6(来源:文献表 2)
(2)表 3:这里作者选择了几个 UBE2C 相关重点基因,详细分析了一下。

图 7(来源:文献表 3)
结果四:UBE2C 蛋白互作网络
这一部分结果包含了图表各一张。
(1)图 5:熟悉的蛋白互作图,蓝色代表驱动家族成员,红色代表参与比较重要的细胞过程的分子,绿色是抑癌基因。
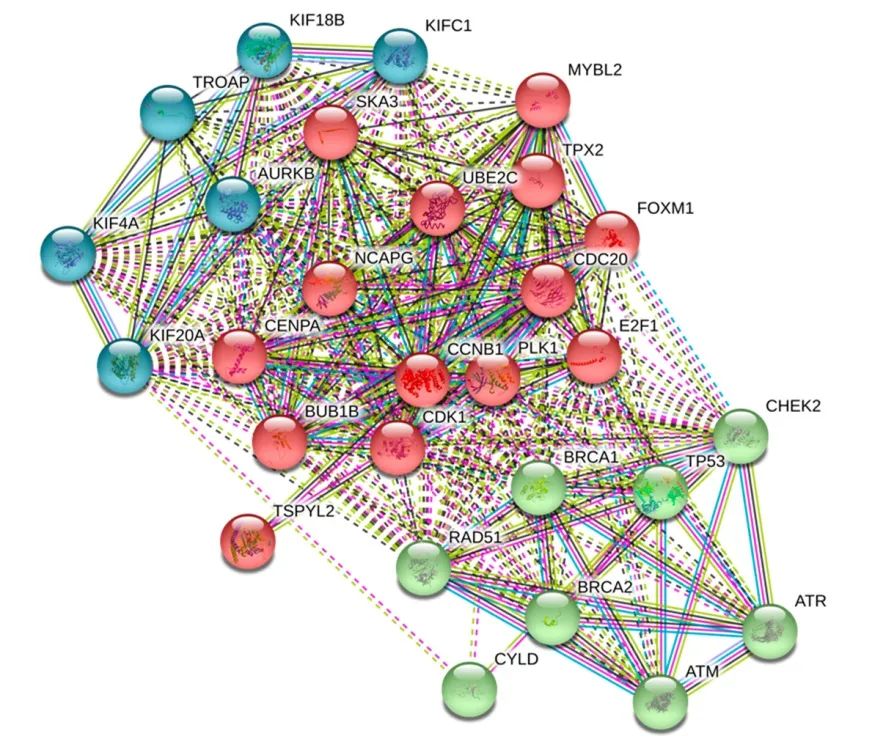
图 8(来源:文献图 5)
(1)表 4:KEGG 通路分析,包含了这些关键基因参与的细胞过程。
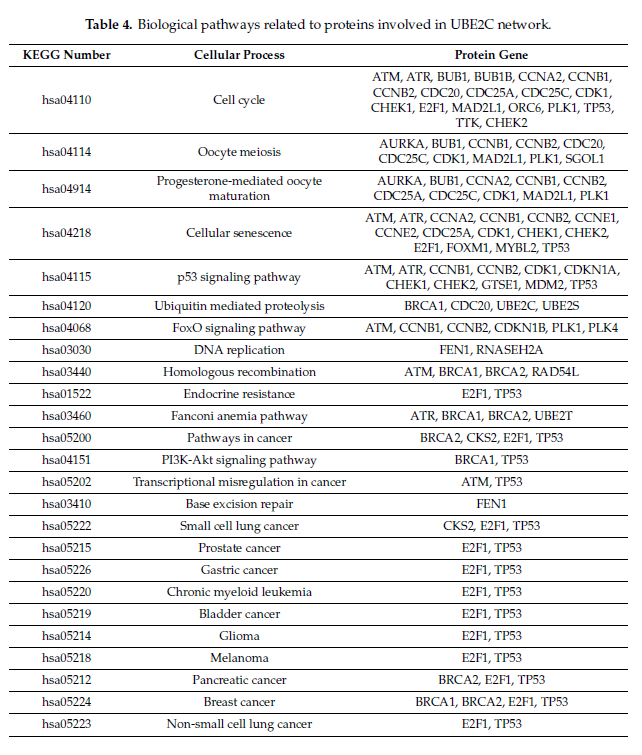
图 9(来源:表 4)
好的,图和表读完了,其实我们这篇文章也已经读到了尾声。
下面,我们再来回忆一下这篇文章结果部分的主要内容:

图 10(来源:自己做的)
总结起来,这篇文章主要的逻辑和我们推测的基本一致:
(1)选取一个基因;
(2)检测它在疾病中是否存在差异性表达;
(3)差异性表达的意义;
(4)与这一表达相关的生物学过程和靶点;
说到这里,是不是快速读懂一篇文章也没有那么难了呢?那今天我们就先读到这里,祝小可爱们读文愉快,撒花 ~
小彩蛋:笔者再补充以下有关这两个数据库的内容。
TCGA 是肿瘤生信分析的老朋友了,它的全称是 The Cancer Genome Atlas(肿瘤基因组图谱),目前共计包含 36 个癌种的转录本,甲基化,突变等数据信息。
而 GTEx 数据库是 TCGA 数据库的好伙伴,由于 TCGA 纳入的大部分是肿瘤组织转录本结果,就会存在对照不足的问题。
所以这篇文章的作者也采用了 GTEx 数据库提供的正常组织测序结果,来补充增加对照组的样本数目。











