【GEO 应用】一篇时髦的生信文章是如何炼成的——检索篇
科研论文时间
随着公共网络数据的增多,测序数据分析技术的发展,我们可以利用的数据越来越多。很多时候,我们可以直接对公共数据库的数据进行分析,进而可以进行数据挖掘,得到的结果用来发表论文。
生信信息学文献复现
这篇《Identification of candidate biomarkers and analysis of prognostic values in ovarian caner by integrated bioinfomatics analysis》(PMID: 27757782. IF: 2.92 )就是利用网络公共芯片数据对卵巢癌的发生进行的数据挖掘。
这种芯片数据分析的论文现在越来越多了。

下面,我们就根据这篇文献,帮大家分析一下这类文献是如何炼成的。
一、文章的基本脉络
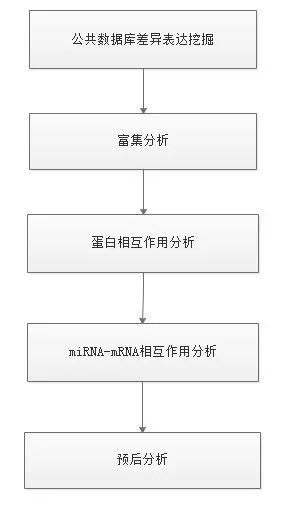
通过阅读文献的材料方法我们可以发现,这篇文献的使用了 3 组数据,都是来自于 GEO 数据库。

之前没有接触过芯片数据的同学可能不知道 GEO 数据库是什么。简单来说,GEO 就是一个可以检索芯片数据的数据库。至于其中的 GSE36668 这样的编号,就类似于文献的 PMID 一样,为自身数据库给每个数据集自己的编码。
二、我们来试一下
1. 我们可以进入 GEO 数据库。(https://www.ncbi.nlm.nih.gov/gds/)
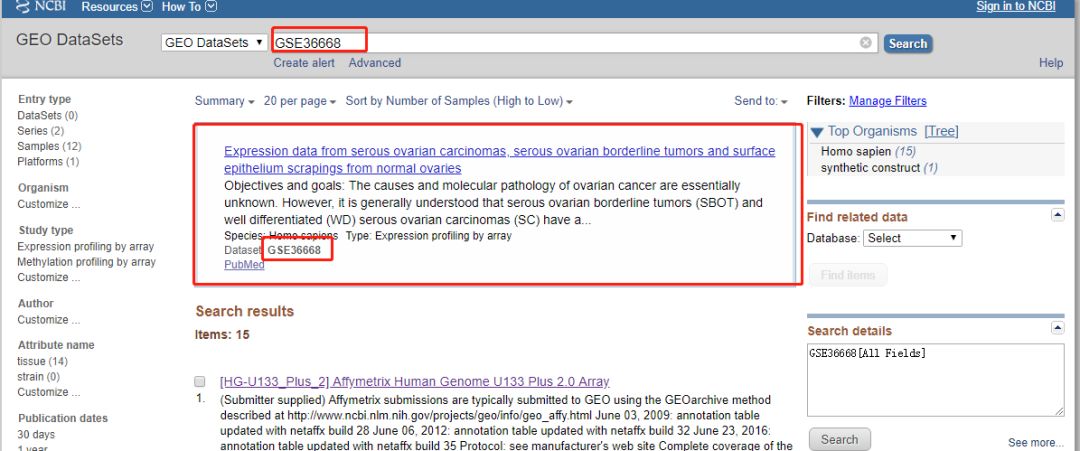
2. 检索关键词,可以是疾病(breast cancer),可以是芯片物种(human),也可以是定向检索 GSE 编码。我们检索 GSE36668,即可得到这个芯片的相关信息。
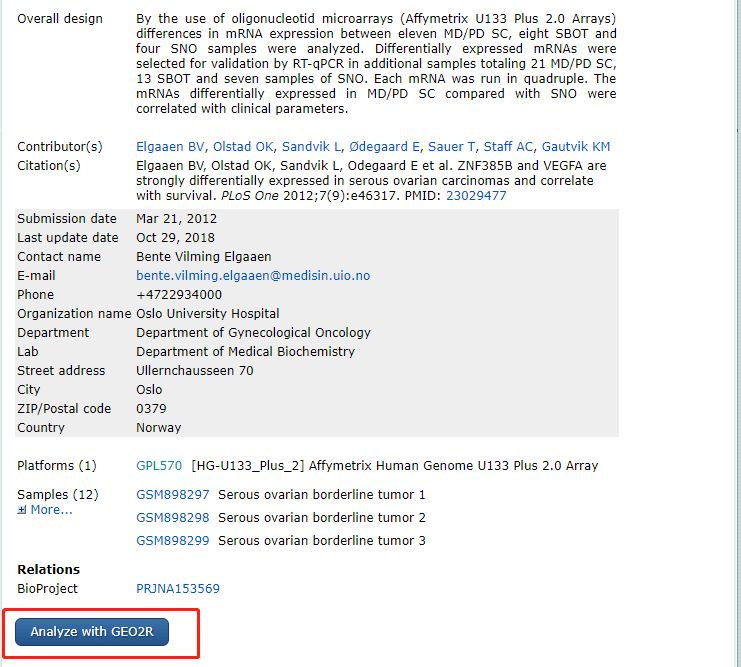
3. 点击进去之后我们可以看到关于这个芯片的详细信息,这样,这个芯片的信息就检索出来了。

这是第一步,那么对于这个芯片我们要怎么分析呢?
这篇文章里面用到的是「GEO2R」。这个工具是 GEO 数据库自己开发的专门用来分析芯片数据的在线的工具。
三、我们要怎么用呢?

1. 我们在刚才检索的到的界面往下拉即可发现有一个「GEO2R」的标志。
2. 点击「GEO2R」看到这个数据集里具体的数据信息。
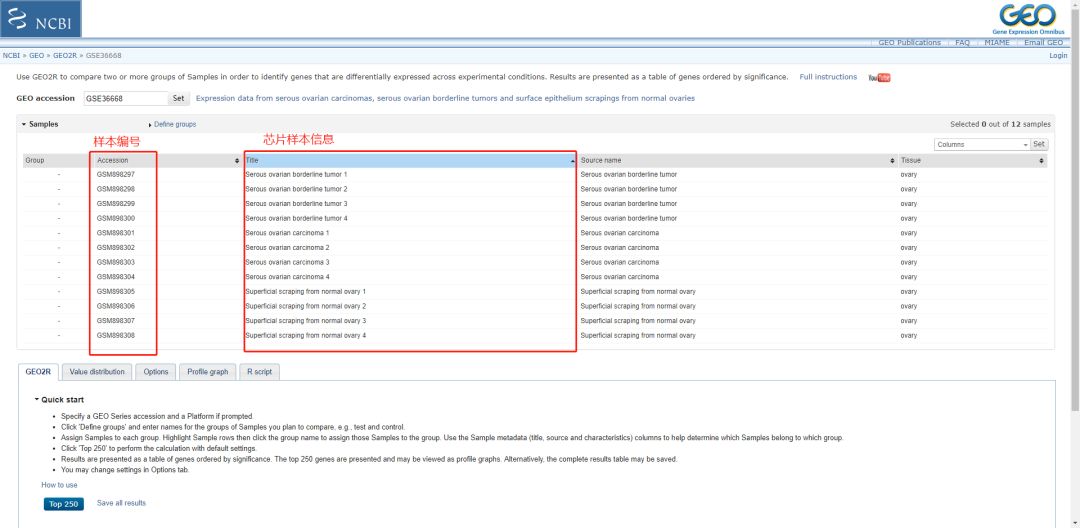
3. 然后对分析的数据进行分组。按照文章的题目我们要分成肿瘤组和正常组。
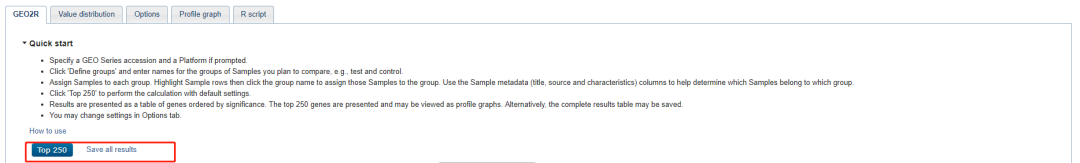
4. 分好组后,点击 top250 即可看到这组数据集里面前 250 个最有差异的基因。点击「top250」。
5. 如下图显示为 top250 的结果。结果中我们可以看到所有探针的差异的 p 值及矫正 p 值,也可以看到基因名,还有 logFC。
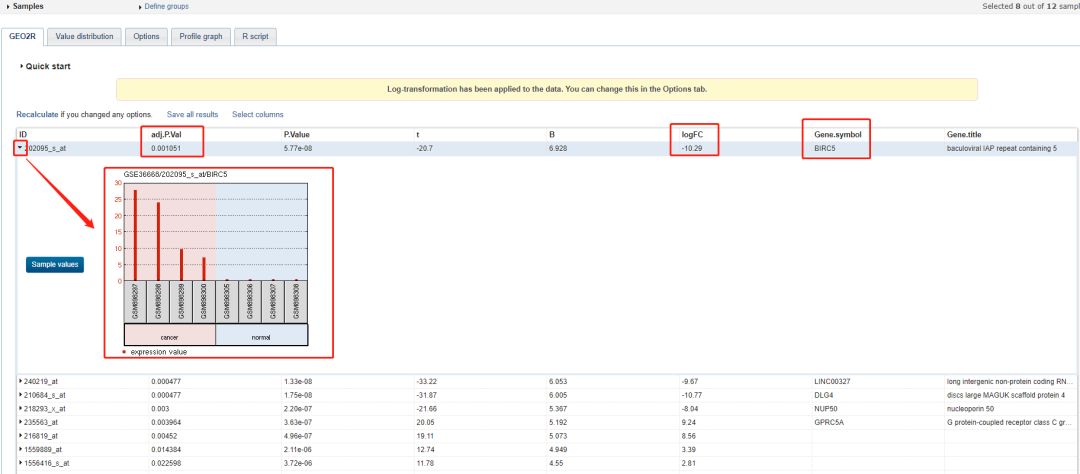
6. 同样,我们可以点击「save all results」即可得到这个数据集中所有的结果。
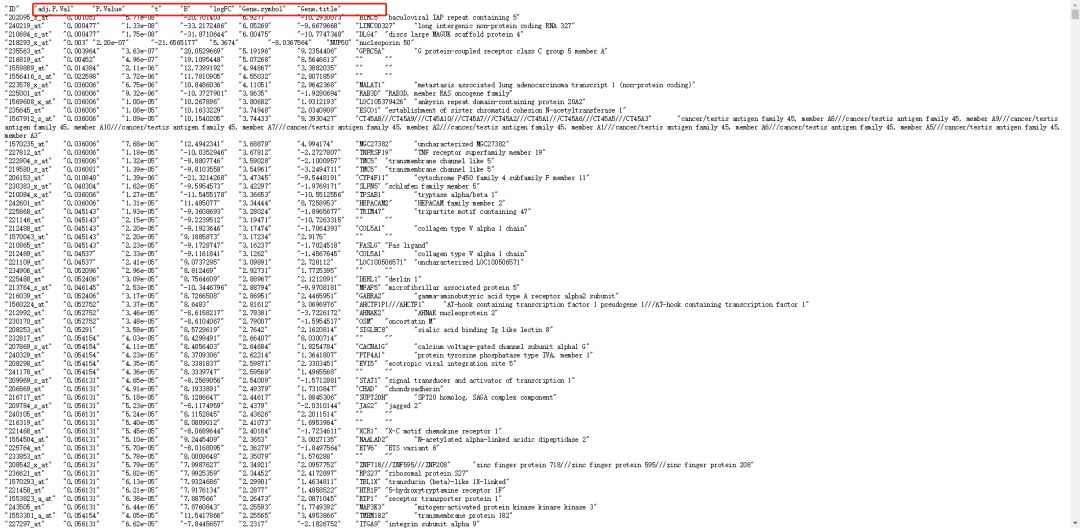
7. 这样的话,我们只需要全选—复制—粘贴到 excel 里面就行了。
按照矫正的 P 值(adj. P < 0.05)及 logFC 的绝对值 >1 即代表有差异。
通过 GEO2R 我们就能得到了差异的基因。按照这个文献的数据,选了三个数据集取交集。











