Blastn之引物序列验证篇
互联网
先找到需要设计引物的目标基因的在有引物序列的 mRNA 序列,可以通过全文(如最先发现该基因的文章),全文中有时可以找到大鼠 c-jun mRNA 序列的 ACCESSION NUMBER ,在 PubMed 中的“ Nucleotide ”中用此 ACCESSION NUMBER 就能找到序列。可以利用这个 ACCESSION NUMBER 在 PubMed 中的“ Nucleotide ”中搜索到序列后,点击右边的“ Links ”,再点击里面的“ Related Sequences ”就能找到所有的大鼠 c-jun mRNA 序列及其他物种的一些 c-jun mRNA 序列。
文献中的引物最好先验证其序列正确,并用软件分析引物比较好后再采用。通过 BLAST 验证,既可知道序列是否正确,也可同时了解引物的特异性、引物的位置及 PCR 产物的大小等。
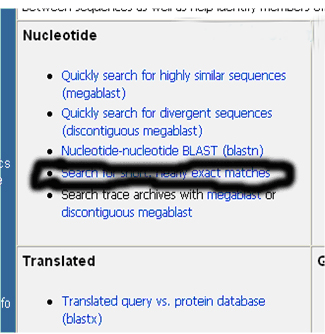
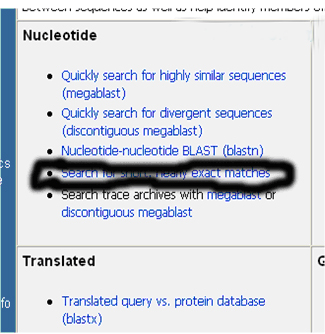
如果仅仅是为了验证引物序列是否正确(仅适合于基于完全匹配原则设计的引物),也可以用 Blast 的“ Nucleotide-nucleotide BLAST (blastn) ”或“ Search for short, nearly exact matches ”搜索,具体方法为:
1. 打开 Blast ,点击“ Nucleotide-nucleotide BLAST (blastn) ”或“ Search for short, nearly exact matches”
2. 等新页面完全显示后,将引物序列直接 copy 到“ Search ”框(没有必要将下游引物序列转换成其互补链),下面 3 种方法都可以(我觉得这 3 种方法的搜索结果没有什么区别,没仔细比较过哦!)
(1)直接拷贝上游引物序列,然后直接将下游引物序列拷贝在上游引物后面
(2)直接拷贝上游引物序列,在上游引物序列后加一空格,然后直接将下游引物序列拷贝在空格后面
(3)直接拷贝上游引物序列,换行,然后直接拷贝下游引物序列
注意:记住上、下游引物的长度,如上游引物为 19bp 、下游引物为 18bp ,这在查看 Blast 结果时有用。
3. 点击“ BLAST !”,新页面完全出来以后,点击“ Format !”。
4. 结果完全出来后,查找有没有和 1-19 、 20-37 同时完全匹配的基因,其他的就不用考虑了。当然,如果下游引物序列所对应的基因片段的 3' 端正好和上游引物序列的 3' 端的 1 或 2 个碱基互补,那就可能是 19-37 或 18-37 。
如果有,那你的引物序列就是正确的。从文献上查的引物,除了要用 Blast 验证其序列是否正确外,还是应该用软件分析分析到底引物好不好。