【转帖】染色质免疫沉淀(ChIP)技术的难点及应用
丁香园论坛
3051
【转帖】染色质免疫沉淀(ChIP)技术的难点及应用
本文转自密理博中国博客,转载请注明出处
作者:柴晶晶
序言
随着人类基因组测序工作的基本完成,功能基因组学的研究逐渐成为研究的热点。而基因表达的调控又是功能基因组学的一个重要研究领域。研究某个蛋白因子的调控功能,可以通过对蛋白活性(激活或抑制其活性),蛋白数量(过表达Overexpression或基因缺陷型Knockout), 以及蛋白功能(功能缺陷型蛋白Dominant-negative mutation)的控制,影响下游基因的表达,而下游基因的变化又可以通过基因芯片(cDNA Microarray),抑制消减杂交(Suppression Subtractive Hybridization),差异显示RT-PCR等方法进行研究[1]。然而这些方法都无法提供证据证明这些变化是受某个蛋白因子直接调节的,还是间接的其他变化导致的结果。所以,要想提供蛋白因子直接调控的证据,就要直接检测蛋白质-DNA的相互作用。传统的方法包括转录因子结合实验(Transcription Factor Assay),电泳迁移率变动分析(electrophoretic mobility shift assay),DNase I 足印法(DNase I footprinting),酵母单杂交系统等。但这些方法都有一定的局限性,不能充分反映生理情况下DNA与蛋白相互作用的真实情况,而且很难捕捉到在染色质水平上基因表达调控的动态瞬时事件[2]。
染色质免疫沉淀技术(Chromatin Immunoprecipitation,简称ChIP)是研究体内蛋白质与DNA相互作用的一种技术。它利用抗原抗体反应的特异性,可以真实地反映体内蛋白因子与基因组DNA结合的状况。特别是近年来由于该技术不断的发展和完善,其应用范围已经从研究目的蛋白与已知靶序列间的相互作用,发展到研究目的蛋白与整个基因组的未知序列的相互作用;从研究一个目的蛋白与DNA的相互作用,发展到研究两个蛋白与DNA共同结合的相互作用;从研究启动子区域的组蛋白的修饰,发展到研究结合在DNA序列上的蛋白复合物。随着对基因功能研究的不断深入,这项技术正越来越多的被应用于科研的各个领域。目前已经有成熟的ChIP试剂盒出售,如Millipore公司提供的EZ-ChIP试剂盒,使得越来越多的研究者更容易地采用染色质沉淀技术在许多研究领域取得了成功。
ChIP技术的原理
染色质免疫沉淀技术的原理是:在生理状态下把细胞内的DNA与蛋白质交联在一起,通过超声或酶处理将染色质切为小片段后,利用抗原抗体的特异性识别反应,将与目的蛋白相结合的Dundefined*段沉淀下来。染色质免疫沉淀技术一般包括细胞固定,染色质断裂,染色质免疫沉淀,交联反应的逆转,DNA的纯化,以及DNA的鉴定。因为ChIP实验涉及的步骤多,结果的重复性较低,所以对ChIP实验过程的每一步都应设计相应的对照,而且对结果的分析也需要有一定的经验。对于刚刚开始使用ChIP技术的研究人员来说,使用成熟的商品化试剂盒和相关的技术服务会达到事半功倍的效果,比如Millipore公司的EZ-ChIP试剂盒就是专门为初学者设计的入门产品。下面我们就最基本的实验步骤,实验中的小技巧以及需要注意的问题简单介绍一下。
1. 细胞固定
甲醛能有效的使蛋白质-蛋白质,蛋白质-DNA,蛋白质-RNA交联,形成生物复合体,防止细胞内组分的重新分布。甲醛的交联反应是完全可逆的,便于在后续步骤中对DNA和蛋白质进行分析。交联所用的甲醛终浓度为1%,交联时间通常为5分钟到1个小时,具体时间根据实验而定。值得注意的是,交联时间如果过长,细胞染色质难以用超声波破碎,影响ChIP结果,而且实验材料也容易在离心过程中丢失。交联时间如果过短,则交联不完全,产生假阴性。甲醛的交联反应可被加入的甘氨酸终止。
2. 染色质断裂
交联后的染色质可被超声波或Micrococcal Nuclease切成400~600 bp的片段(用琼脂糖凝胶电泳检测),以便暴露目标蛋白,利于抗体识别。超声波是使用机械力断裂染色质,容易引起升温或产生泡沫,这都会引起蛋白质变性,进而影响ChIP的效率。所以在超声波断裂染色质时,要在冰上进行,且要设计时断时续的超声程序,保证低温。另外,超声探头要尽量深入管中,但不接触管底或侧壁,以免产生泡沫。总超声时间也不要太长,以免蛋白降解。
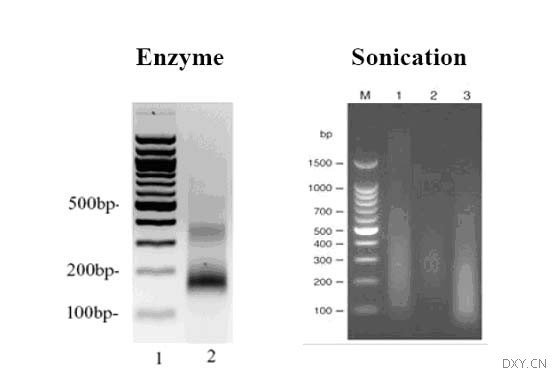
Micrococcal Nuclease可以将染色质切成一到几个核小体,比超声波处理的结果更精致,更均一(图1)。另外,酶反应的条件比较温和,对DNA和DNA-蛋白复合物的损伤较小,而且蛋白不易变性。酶处理染色质适用于新鲜的细胞或组织样品和冰冻样品。在研究组蛋白时,经常采用没经过甲醛固定的Native ChIP(N-ChIP)的研究方法。因为N-ChIP没经过甲醛固定,超声波处理会打断组蛋白和DNA的结合,所以只能选择酶处理染色质的方法。对于甲醛固定的样品,一般选择超声波处理方法。也有研究人员使用酶处理的方法研究甲醛固定较温和的样品。Millipore公司有商品化的Micrococcal Nuclease处理的ChIP试剂盒(EZ-Enzyme)提供。

3. 染色质免疫沉淀
Input对照:
在进行免疫沉淀前,需要取一部分断裂后的染色质做Input对照。Input是断裂后的基因组DNA,需要与沉淀后的样品DNA一起经过逆转交联,DNA纯化,以及最后的PCR或其他方法检测。Input对照不仅可以验证染色质断裂的效果,还可以根据Input中的靶序列的含量以及染色质沉淀中的靶序列的含量,按照取样比例换算出ChIP的效率,所以Input对照是ChIP实验必不可少的步骤。
Beads选择:
接下来,利用目的蛋白质的特异抗体通过抗原-抗体反应形成DNA-蛋白质-抗体复合物,然后使用Agarose beads或Magna beads沉淀此复合物,特异性地富集与目的蛋白结合的Dundefined*段。再经过多次洗涤,除去非特异结合的染色质后,用SDS+NaHCO3洗脱免疫沉淀复合物。Magna beads是近年来出现的一种新型beads,它使用方便,不像Agarose beads那样容易破裂,所以在操作过程中更简单,而且免去了离心的步骤,节省不少时间。Millipore公司最新推出的Magna ChIP试剂盒就是采用这种Magna beads。
抗体选择:
染色质免疫沉淀所选择的目的蛋白的抗体是ChIP实验成功的关键。因为在蛋白质与染色质交联结合时,抗体的抗原表位可能因为与结合位点的距离太近,不能被抗体识别,所以不能有效地在体内形成免疫沉淀复合物,直接影响ChIP的结果。所以不是所有的抗体都能做ChIP实验的,只有经过ChIP实验验证后的抗体才能确保实验结果的可靠性,比如Millipore公司的ChIP Validated抗体等。
阳性与阴性对照:
在做ChIP实验时,一定要做好实验对照,因为没有对照,很难对实验结果的可靠性进行评估。阳性抗体和阴性抗体对照是最基本的实验对照。阳性抗体通常选择与已知序列相结合的比较保守的蛋白的抗体,常用的包括组蛋白抗体或RNA Polymerase II抗体等。阴性抗体通常选择目的蛋白抗体宿主的IgG或血清。目的蛋白抗体的结果与阳性抗体和阴性抗体的结果相比较,才能得出正确结论。另外,还应考虑目的蛋白抗体与DNA的非特异性结合的可能,所以通常还会选择一对阴性引物,即目的蛋白肯定不会结合的DNA序列,作为该抗体的阴性对照。最佳的阴性对照引物是在靶序列上游的一段与目的蛋白肯定不能结合的序列。如果目的蛋白没有商品化的适用于染色质免疫沉淀实验的抗体,只有其他用途的抗体时,可以先做蛋白质免疫沉淀(Immunoprecipitation)检测。如果抗体可以成功的沉淀蛋白,再进行染色质免疫沉淀实验的检测。
4. 交联反应的逆转和DNA的纯化
用不含DNase的RNase和Proteinase K,65oC保温6小时逆转交联,经DNA纯化柱回收DNA或用酚氯仿抽提、乙醇沉淀纯化DNA。DNA纯化柱纯化DNA的质量高,有利于下一步PCR等方法的检测。因为甲醛不仅交联DNA-蛋白质,还交联蛋白质-蛋白质,所以还可以对DNA序列上的蛋白质复合物进行分析。在逆转交联时不使用Proteinase K,然后用丙酮回收有机相中的蛋白质,进行分析。
5. DNA的鉴定
最常用的DNA的鉴定方法是半定量PCR和Real-time PCR。由于启动子区域的序列具有多样性的特点,所以不同的细胞系或不同的动物品系的同一基因的启动子序列有可能不同。而且启动子区域多富含CG的序列,其PCR条件可能需要相应调整。有条件可设计不止一对引物来反复验证ChIP实验的结果(图2)。

ChIP技术的应用
染色质免疫沉淀的DNA适用于多种分析方法。如果目的蛋白的靶序列是已知的或需要验证的,可采用狭缝杂交(Slot blot)的方法,把靶序列特异性探针与染色质免疫沉淀的DNA杂交,来验证目的蛋白与DNA靶序列的特异性结合。还可以根据靶序列设计引物,用半定量PCR的方法进行测定,或采用Real-time PCR方法进行定量分析。如果目的蛋白的靶序列是未知的或高通量的(high-throughput),可采用Southern杂交。但因为免疫沉淀的DNA量较少,所以在研究时通常要用PCR方法扩增DNA探针,再进行整个基因组扫描。还可以把沉淀的DNA克隆到载体中,进行测序,寻找该序列附近的开放阅读框,发现新的基因调节序列。
目前,随着人类基因组测序工作的基本完成,研究目的蛋白和整个基因组的相互作用逐渐成为研究的热点。由于基因组中的信息量非常大,上述常规方法通常无法满足科研的需要。近年来发展起来的ChIP-chip技术将基因组DNA芯片(chip)技术与染色质免疫沉淀技术(ChIP)相结合,为研究目的蛋白与整个基因组相互作用提供了可能。ChIP-chip 技术通过标记染色质免疫沉淀富集的Dundefined*段,和另一个被标记不同探针的对照组样品一起,与DNA芯片杂交,再利用各种生物信息学方法对收集到的信号进行分析,具体的实验步骤请参考Dr. Richard Young在Nature Protocols上的文章[3]。ChIP-chip技术已经被广泛应用于研究转录因子在整个基因组中的信号网络[4, 5, 6],染色质修饰机制在基因组中的调控[7, 8],DNA的复制,修复以及修饰[9, 10],基因的转录与核运输[11, 12, 13]等诸多方面。
染色质免疫沉淀技术还可用于分析两种蛋白共同结合的DNA序列,即ChIP reChIP方法。ChIP reChIP是在第一次ChIP的基础上不解交联,而继续进行另一个目的蛋白的免疫沉淀,从而得到与两种目的蛋白都结合的DNA序列。值得注意的是,因为通过两次免疫沉淀富集的DNA量比较少,所以在分析时通常要把多次免疫沉淀的DNA浓缩后再进行操作。
随着染色质免疫沉淀技术受到广泛的关注,运用该技术发表的文章也逐渐增多,大家越来越多的开始关注如何改进ChIP的方法。Millipore最新推出的Magna ChIP试剂盒,利用磁珠分离DNA-蛋白-抗体复合物,提高了ChIP的效率,简化了操作的过程,缩短了实验的时间,还为同时进行多个目的蛋白的研究提供了可能,是经常使用ChIP技术的研究人员的理想选择。
近年来ChIP技术也被用于研究RNA-蛋白的相互作用,其原理与DNA类似,也包括甲醛固定,超声波破细胞,免疫沉淀,交联逆转,RNA纯化和RNA鉴定等步骤。所不同的是,交联逆转只用Proteinase K,要进行RNA纯化和不含RNase的DNase处理,分析时用RT-PCR,芯片杂交要用cDNA芯片等[13, 14]。
参考文献
1. 王春雨,石建党,朱彦,张琚。染色质免疫沉淀技术在研究DNA与蛋白质相互作用中的应用。遗传HEREDITAS (Beijing), 27(5): 801, 2005.
2. Im H, Grass JA, Johnson KD, et al. Measurement of protein-DNA interaction in vivo by chromatin immunoprecipitation. Methods Mol. Biol., 284: 129, 2004.
3. Lee T. I., Johnstone S.E., Young R.A., Chromatin immunoprecipitation and microarray-based analysis of protein location. Nat. Protocols, 1, 729-747, 2006.
4. Nal B., Mohr E., Ferrier P.. Location analysis of DNA-bound proteins at the whole-genome level: untangling transcriptional regulatory networks. Bioessays 23, 473-476, 2001.
5. Hanlon S.E., Lieb J.D.. Progress and challenges in profiling the dynamics of chromatin and transcription factor binding with DNA microarrays. Curr. Opin. Genet. Dev. 14, 697-705, 2004.
6. Banerjee N., Zhang M.Q.. Identifying cooperativity among transcription factors controlling the cell cycle in yeast. Nucleic Acids Res., 31; 7024-7031, 2003.
7. Carter N.P., Vetrie D.. Applications of genomic microarrays to explore human chromosome structure and function. Hum. Mol. Genet. 13 (Spec No 2), R297-R302, 2004.
8. Van Steensel B., Henikoff S.. Epigenetic profiling using microarrays. Biotechniques, 35, 346-357, 2003.
9. MacAlpine D.M., Bell S.P.. A genomic view of eukaryotic DNA replication. Chromosome Res., 13, 309-326, 2005.
10. Woodfine K., Carter N.P., Dunham I., Fiegler H.. Investigating chromosome organization with genomic microarrays. Chromosome Res., 13, 249-257, 2005.
11. Lei E.P., Krebber H., Silver P.A.. Messenger RNAs are recruited for nuclear export during transcription. Genes. Dev., 15, 1771-1782, 2001.
12. Ishii K., Arib G., Lin C., Van Houwe G., Laemmi U.K.. Chromatin boundaries in budding yeast: the nuclear pore connection. Cell, 109, 551-562, 2002.
13. Rodriguez-Navarro S. et al. Sus1, a functional component of the SAGA histone acetylase complex and the nuclear pore-associated mRNA export machinery. Cell, 116, 75-86, 2004.
14. Nitranjanakumari S., Lasda E., Brazas R., Garcia-Blanco M.. A. Reversible cross-linking combined with immunoprecipitation to study RNA-protein interactions in vivo. Methods, 26(2); 182-190, 2002.
15. de Silanes I L., Zhan M., Lai A., Yang X., Gorospe M.. Identification of a target RNA motif for RNA-binding protein HuR. PNAS, 101(9); 2987-2992, 2004.
作者简介:柴晶晶博士毕业于北大生物系,在美国北卡罗来纳大学获病理系博士学位,留美期间主要从事肿瘤和表观遗传学的研究,曾任中国科学院上海生命科学院细胞与生化所副研究员。
本文转自密理博中国博客,转载请注明出处
作者:柴晶晶
序言
随着人类基因组测序工作的基本完成,功能基因组学的研究逐渐成为研究的热点。而基因表达的调控又是功能基因组学的一个重要研究领域。研究某个蛋白因子的调控功能,可以通过对蛋白活性(激活或抑制其活性),蛋白数量(过表达Overexpression或基因缺陷型Knockout), 以及蛋白功能(功能缺陷型蛋白Dominant-negative mutation)的控制,影响下游基因的表达,而下游基因的变化又可以通过基因芯片(cDNA Microarray),抑制消减杂交(Suppression Subtractive Hybridization),差异显示RT-PCR等方法进行研究[1]。然而这些方法都无法提供证据证明这些变化是受某个蛋白因子直接调节的,还是间接的其他变化导致的结果。所以,要想提供蛋白因子直接调控的证据,就要直接检测蛋白质-DNA的相互作用。传统的方法包括转录因子结合实验(Transcription Factor Assay),电泳迁移率变动分析(electrophoretic mobility shift assay),DNase I 足印法(DNase I footprinting),酵母单杂交系统等。但这些方法都有一定的局限性,不能充分反映生理情况下DNA与蛋白相互作用的真实情况,而且很难捕捉到在染色质水平上基因表达调控的动态瞬时事件[2]。
染色质免疫沉淀技术(Chromatin Immunoprecipitation,简称ChIP)是研究体内蛋白质与DNA相互作用的一种技术。它利用抗原抗体反应的特异性,可以真实地反映体内蛋白因子与基因组DNA结合的状况。特别是近年来由于该技术不断的发展和完善,其应用范围已经从研究目的蛋白与已知靶序列间的相互作用,发展到研究目的蛋白与整个基因组的未知序列的相互作用;从研究一个目的蛋白与DNA的相互作用,发展到研究两个蛋白与DNA共同结合的相互作用;从研究启动子区域的组蛋白的修饰,发展到研究结合在DNA序列上的蛋白复合物。随着对基因功能研究的不断深入,这项技术正越来越多的被应用于科研的各个领域。目前已经有成熟的ChIP试剂盒出售,如Millipore公司提供的EZ-ChIP试剂盒,使得越来越多的研究者更容易地采用染色质沉淀技术在许多研究领域取得了成功。
ChIP技术的原理
染色质免疫沉淀技术的原理是:在生理状态下把细胞内的DNA与蛋白质交联在一起,通过超声或酶处理将染色质切为小片段后,利用抗原抗体的特异性识别反应,将与目的蛋白相结合的Dundefined*段沉淀下来。染色质免疫沉淀技术一般包括细胞固定,染色质断裂,染色质免疫沉淀,交联反应的逆转,DNA的纯化,以及DNA的鉴定。因为ChIP实验涉及的步骤多,结果的重复性较低,所以对ChIP实验过程的每一步都应设计相应的对照,而且对结果的分析也需要有一定的经验。对于刚刚开始使用ChIP技术的研究人员来说,使用成熟的商品化试剂盒和相关的技术服务会达到事半功倍的效果,比如Millipore公司的EZ-ChIP试剂盒就是专门为初学者设计的入门产品。下面我们就最基本的实验步骤,实验中的小技巧以及需要注意的问题简单介绍一下。
1. 细胞固定
甲醛能有效的使蛋白质-蛋白质,蛋白质-DNA,蛋白质-RNA交联,形成生物复合体,防止细胞内组分的重新分布。甲醛的交联反应是完全可逆的,便于在后续步骤中对DNA和蛋白质进行分析。交联所用的甲醛终浓度为1%,交联时间通常为5分钟到1个小时,具体时间根据实验而定。值得注意的是,交联时间如果过长,细胞染色质难以用超声波破碎,影响ChIP结果,而且实验材料也容易在离心过程中丢失。交联时间如果过短,则交联不完全,产生假阴性。甲醛的交联反应可被加入的甘氨酸终止。
2. 染色质断裂
交联后的染色质可被超声波或Micrococcal Nuclease切成400~600 bp的片段(用琼脂糖凝胶电泳检测),以便暴露目标蛋白,利于抗体识别。超声波是使用机械力断裂染色质,容易引起升温或产生泡沫,这都会引起蛋白质变性,进而影响ChIP的效率。所以在超声波断裂染色质时,要在冰上进行,且要设计时断时续的超声程序,保证低温。另外,超声探头要尽量深入管中,但不接触管底或侧壁,以免产生泡沫。总超声时间也不要太长,以免蛋白降解。
Micrococcal Nuclease可以将染色质切成一到几个核小体,比超声波处理的结果更精致,更均一(图1)。另外,酶反应的条件比较温和,对DNA和DNA-蛋白复合物的损伤较小,而且蛋白不易变性。酶处理染色质适用于新鲜的细胞或组织样品和冰冻样品。在研究组蛋白时,经常采用没经过甲醛固定的Native ChIP(N-ChIP)的研究方法。因为N-ChIP没经过甲醛固定,超声波处理会打断组蛋白和DNA的结合,所以只能选择酶处理染色质的方法。对于甲醛固定的样品,一般选择超声波处理方法。也有研究人员使用酶处理的方法研究甲醛固定较温和的样品。Millipore公司有商品化的Micrococcal Nuclease处理的ChIP试剂盒(EZ-Enzyme)提供。

3. 染色质免疫沉淀
Input对照:
在进行免疫沉淀前,需要取一部分断裂后的染色质做Input对照。Input是断裂后的基因组DNA,需要与沉淀后的样品DNA一起经过逆转交联,DNA纯化,以及最后的PCR或其他方法检测。Input对照不仅可以验证染色质断裂的效果,还可以根据Input中的靶序列的含量以及染色质沉淀中的靶序列的含量,按照取样比例换算出ChIP的效率,所以Input对照是ChIP实验必不可少的步骤。
Beads选择:
接下来,利用目的蛋白质的特异抗体通过抗原-抗体反应形成DNA-蛋白质-抗体复合物,然后使用Agarose beads或Magna beads沉淀此复合物,特异性地富集与目的蛋白结合的Dundefined*段。再经过多次洗涤,除去非特异结合的染色质后,用SDS+NaHCO3洗脱免疫沉淀复合物。Magna beads是近年来出现的一种新型beads,它使用方便,不像Agarose beads那样容易破裂,所以在操作过程中更简单,而且免去了离心的步骤,节省不少时间。Millipore公司最新推出的Magna ChIP试剂盒就是采用这种Magna beads。
抗体选择:
染色质免疫沉淀所选择的目的蛋白的抗体是ChIP实验成功的关键。因为在蛋白质与染色质交联结合时,抗体的抗原表位可能因为与结合位点的距离太近,不能被抗体识别,所以不能有效地在体内形成免疫沉淀复合物,直接影响ChIP的结果。所以不是所有的抗体都能做ChIP实验的,只有经过ChIP实验验证后的抗体才能确保实验结果的可靠性,比如Millipore公司的ChIP Validated抗体等。
阳性与阴性对照:
在做ChIP实验时,一定要做好实验对照,因为没有对照,很难对实验结果的可靠性进行评估。阳性抗体和阴性抗体对照是最基本的实验对照。阳性抗体通常选择与已知序列相结合的比较保守的蛋白的抗体,常用的包括组蛋白抗体或RNA Polymerase II抗体等。阴性抗体通常选择目的蛋白抗体宿主的IgG或血清。目的蛋白抗体的结果与阳性抗体和阴性抗体的结果相比较,才能得出正确结论。另外,还应考虑目的蛋白抗体与DNA的非特异性结合的可能,所以通常还会选择一对阴性引物,即目的蛋白肯定不会结合的DNA序列,作为该抗体的阴性对照。最佳的阴性对照引物是在靶序列上游的一段与目的蛋白肯定不能结合的序列。如果目的蛋白没有商品化的适用于染色质免疫沉淀实验的抗体,只有其他用途的抗体时,可以先做蛋白质免疫沉淀(Immunoprecipitation)检测。如果抗体可以成功的沉淀蛋白,再进行染色质免疫沉淀实验的检测。
4. 交联反应的逆转和DNA的纯化
用不含DNase的RNase和Proteinase K,65oC保温6小时逆转交联,经DNA纯化柱回收DNA或用酚氯仿抽提、乙醇沉淀纯化DNA。DNA纯化柱纯化DNA的质量高,有利于下一步PCR等方法的检测。因为甲醛不仅交联DNA-蛋白质,还交联蛋白质-蛋白质,所以还可以对DNA序列上的蛋白质复合物进行分析。在逆转交联时不使用Proteinase K,然后用丙酮回收有机相中的蛋白质,进行分析。
5. DNA的鉴定
最常用的DNA的鉴定方法是半定量PCR和Real-time PCR。由于启动子区域的序列具有多样性的特点,所以不同的细胞系或不同的动物品系的同一基因的启动子序列有可能不同。而且启动子区域多富含CG的序列,其PCR条件可能需要相应调整。有条件可设计不止一对引物来反复验证ChIP实验的结果(图2)。

ChIP技术的应用
染色质免疫沉淀的DNA适用于多种分析方法。如果目的蛋白的靶序列是已知的或需要验证的,可采用狭缝杂交(Slot blot)的方法,把靶序列特异性探针与染色质免疫沉淀的DNA杂交,来验证目的蛋白与DNA靶序列的特异性结合。还可以根据靶序列设计引物,用半定量PCR的方法进行测定,或采用Real-time PCR方法进行定量分析。如果目的蛋白的靶序列是未知的或高通量的(high-throughput),可采用Southern杂交。但因为免疫沉淀的DNA量较少,所以在研究时通常要用PCR方法扩增DNA探针,再进行整个基因组扫描。还可以把沉淀的DNA克隆到载体中,进行测序,寻找该序列附近的开放阅读框,发现新的基因调节序列。
目前,随着人类基因组测序工作的基本完成,研究目的蛋白和整个基因组的相互作用逐渐成为研究的热点。由于基因组中的信息量非常大,上述常规方法通常无法满足科研的需要。近年来发展起来的ChIP-chip技术将基因组DNA芯片(chip)技术与染色质免疫沉淀技术(ChIP)相结合,为研究目的蛋白与整个基因组相互作用提供了可能。ChIP-chip 技术通过标记染色质免疫沉淀富集的Dundefined*段,和另一个被标记不同探针的对照组样品一起,与DNA芯片杂交,再利用各种生物信息学方法对收集到的信号进行分析,具体的实验步骤请参考Dr. Richard Young在Nature Protocols上的文章[3]。ChIP-chip技术已经被广泛应用于研究转录因子在整个基因组中的信号网络[4, 5, 6],染色质修饰机制在基因组中的调控[7, 8],DNA的复制,修复以及修饰[9, 10],基因的转录与核运输[11, 12, 13]等诸多方面。
染色质免疫沉淀技术还可用于分析两种蛋白共同结合的DNA序列,即ChIP reChIP方法。ChIP reChIP是在第一次ChIP的基础上不解交联,而继续进行另一个目的蛋白的免疫沉淀,从而得到与两种目的蛋白都结合的DNA序列。值得注意的是,因为通过两次免疫沉淀富集的DNA量比较少,所以在分析时通常要把多次免疫沉淀的DNA浓缩后再进行操作。
随着染色质免疫沉淀技术受到广泛的关注,运用该技术发表的文章也逐渐增多,大家越来越多的开始关注如何改进ChIP的方法。Millipore最新推出的Magna ChIP试剂盒,利用磁珠分离DNA-蛋白-抗体复合物,提高了ChIP的效率,简化了操作的过程,缩短了实验的时间,还为同时进行多个目的蛋白的研究提供了可能,是经常使用ChIP技术的研究人员的理想选择。
近年来ChIP技术也被用于研究RNA-蛋白的相互作用,其原理与DNA类似,也包括甲醛固定,超声波破细胞,免疫沉淀,交联逆转,RNA纯化和RNA鉴定等步骤。所不同的是,交联逆转只用Proteinase K,要进行RNA纯化和不含RNase的DNase处理,分析时用RT-PCR,芯片杂交要用cDNA芯片等[13, 14]。
参考文献
1. 王春雨,石建党,朱彦,张琚。染色质免疫沉淀技术在研究DNA与蛋白质相互作用中的应用。遗传HEREDITAS (Beijing), 27(5): 801, 2005.
2. Im H, Grass JA, Johnson KD, et al. Measurement of protein-DNA interaction in vivo by chromatin immunoprecipitation. Methods Mol. Biol., 284: 129, 2004.
3. Lee T. I., Johnstone S.E., Young R.A., Chromatin immunoprecipitation and microarray-based analysis of protein location. Nat. Protocols, 1, 729-747, 2006.
4. Nal B., Mohr E., Ferrier P.. Location analysis of DNA-bound proteins at the whole-genome level: untangling transcriptional regulatory networks. Bioessays 23, 473-476, 2001.
5. Hanlon S.E., Lieb J.D.. Progress and challenges in profiling the dynamics of chromatin and transcription factor binding with DNA microarrays. Curr. Opin. Genet. Dev. 14, 697-705, 2004.
6. Banerjee N., Zhang M.Q.. Identifying cooperativity among transcription factors controlling the cell cycle in yeast. Nucleic Acids Res., 31; 7024-7031, 2003.
7. Carter N.P., Vetrie D.. Applications of genomic microarrays to explore human chromosome structure and function. Hum. Mol. Genet. 13 (Spec No 2), R297-R302, 2004.
8. Van Steensel B., Henikoff S.. Epigenetic profiling using microarrays. Biotechniques, 35, 346-357, 2003.
9. MacAlpine D.M., Bell S.P.. A genomic view of eukaryotic DNA replication. Chromosome Res., 13, 309-326, 2005.
10. Woodfine K., Carter N.P., Dunham I., Fiegler H.. Investigating chromosome organization with genomic microarrays. Chromosome Res., 13, 249-257, 2005.
11. Lei E.P., Krebber H., Silver P.A.. Messenger RNAs are recruited for nuclear export during transcription. Genes. Dev., 15, 1771-1782, 2001.
12. Ishii K., Arib G., Lin C., Van Houwe G., Laemmi U.K.. Chromatin boundaries in budding yeast: the nuclear pore connection. Cell, 109, 551-562, 2002.
13. Rodriguez-Navarro S. et al. Sus1, a functional component of the SAGA histone acetylase complex and the nuclear pore-associated mRNA export machinery. Cell, 116, 75-86, 2004.
14. Nitranjanakumari S., Lasda E., Brazas R., Garcia-Blanco M.. A. Reversible cross-linking combined with immunoprecipitation to study RNA-protein interactions in vivo. Methods, 26(2); 182-190, 2002.
15. de Silanes I L., Zhan M., Lai A., Yang X., Gorospe M.. Identification of a target RNA motif for RNA-binding protein HuR. PNAS, 101(9); 2987-2992, 2004.
作者简介:柴晶晶博士毕业于北大生物系,在美国北卡罗来纳大学获病理系博士学位,留美期间主要从事肿瘤和表观遗传学的研究,曾任中国科学院上海生命科学院细胞与生化所副研究员。

![2,4-二氯-1-萘酚[用于照相技术],2050-76-2,≥97%(GC)(T),阿拉丁](https://img1.dxycdn.com/p/s14/2024/0619/923/2131019441498633081.jpg!wh200)