基因组编辑技术应用于作物遗传改良的进展(上篇)
上海迈其
基因组编辑技术是指可以在基因组水平上对 DNA 序列进行定点改造的遗传操作技术,其在基因功能研究和改造、生物医学和植物遗传改良等方面都具有重大的应用价值。科学家自 20 世纪 90 年代末就开始探索基因组定点编辑技术,但直到 2002 年,也仅在小鼠和果蝇等少数模式生物中实现了同源重组介导的基因组定点编辑,且因同源重组的效率很低,限制了其应用前景。进入 21 世纪后,随着蛋白质结构与功能研究的新突破和人工核酸内切酶 (engineeredendonuclease,EEN) 技术的出现,将特异识别并结合 DNA 的蛋白结构域和 EEN 融合,创造出能够特异切割 DNA 序列的核酸酶 (sequence-specificnucleases,SSNs),从而可以对基因组特定位点进行高效和精确的靶向编辑。
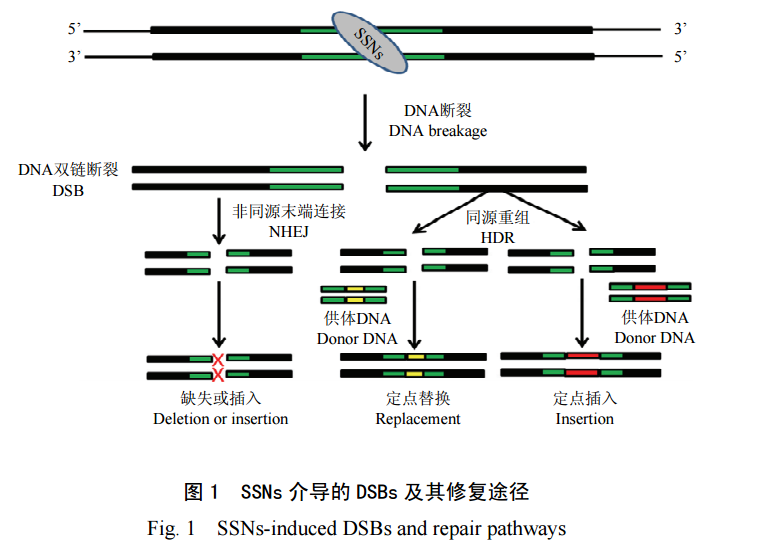
目前,SSNs 主要包括锌指核酸酶 (Zincfingernucleases,ZFNs)、类转录激活因子效应物核酸酶 (transcriptionactivator-likeeffectornucleases,TALENs)、成簇的规律间隔的短回文重复序列及其相关系统 (clusteredregularlyinterspacedshortpalindromicrepeats/CRISPR-associatedCas9,CRISPR/Cas9system) 和 CRISPR/Cpf1 系统。这些 SSNs 的共同特点是都能在基因组特定部位精确切割 DNA 双链,造成 DNA 双链断裂 (DNAdouble-strandbreaks,DSBs);而 DSBs 能够极大地提高染色体重组事件发生的概率。DSBs 的修复机制在真核生物细胞中高度保守,主要包括同源重组 (homology-directedrepair,HDR) 和非同源末端连接 (non-homologousendjoining,NHEJ)2 种修复途径 (图 1)。当存在同源序列供体 DNA 时,以 HDR 方式的修复能够产生精确的定点替换或插入;而没有供体 DNA 时,细胞则通过 NHEJ 途径修复。因 NHEJ 方式的修复往往不够精确,在 DNA 链断裂位置常会产生少量核酸碱基的插入或缺失 (insertion-deletion,InDel),从而导致基因突变。
SSNs 自 2002 年出现以后,就掀起了一股席卷全球的基因组定点编辑研究热潮。尤其是 2010 年出现 TALENs 和 2013 年出现 CRISPR/Cas9 技术后,因它们相对简单、精确、高效,很快被广泛应用于医学、农业、基础研究等领域。因 TALENs 和 CRISPR/Cas9 潜在的巨大应用价值,它们相继被《科学》杂志评为 2012 年和 2013 年的十大科学突破之一。
1 基因组编辑技术在作物遗传改良上的应用进展
目前,在作物遗传改良上应用的基因组编辑技术主要包括 ZFNs、TALENs 和 CRISPR/Cas 系统 3 种类型。其中 CRISPR/Cas 系统包括 CRISPR/Cas9、CRISPR/Cpf1、CRISPR/C2c1 和 CRISPR/C2c2 等亚类型,但应用最多的是 CRISPR/Cas9,而 CRISPR/C2c1 和 CRISPR/C2c2 尚无在作物改良上应用的报道。
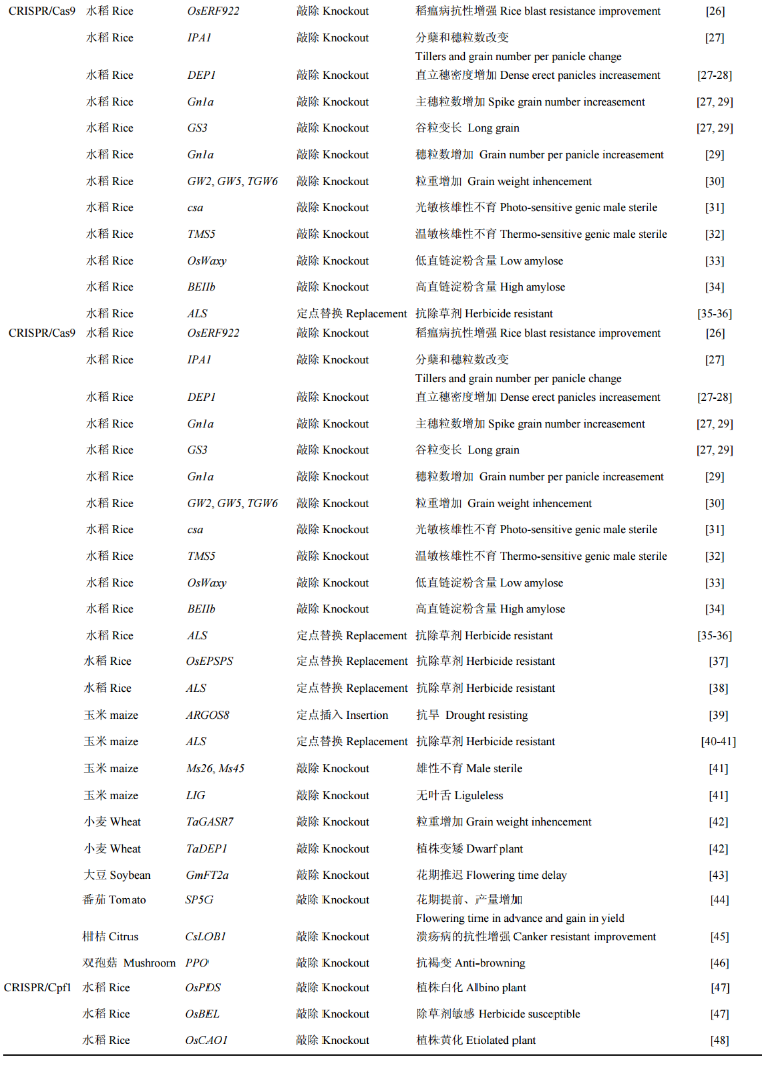
上述 3 类基因组编辑技术均能对植物基因组进行精准的定点敲除、插入和替换,因此,其对于控制作物重要农艺性状基因的功能鉴定、作物重要性状的遗传改良具有巨大的应用潜力。相比传统的常规育种,基因组编辑技术能直接对目标性状基因进行修饰,有可能大幅度提高目标性状聚合的精准度和加快聚合育种进程。而相比传统的转基因育种,基因组编辑在靶向修饰特定基因后,能通过自交或杂交剔除外源基因以消除转基因安全顾虑。因此,自基因组编辑技术成功应用于植物后,对该技术的优化及在作物遗传改良上的应用已成为世界各国和国际农业生物技术公司投资与研发的重点。目前,基因组编辑技术主要集中应用于作物产量、品质、抗性、育性等 4 个方面的遗传改良 (表 1)。现就技术类型和目标性状分述如下。
1.1 ZFNs 技术及其应用
ZFNs 技术被称为第一代基因组编辑技术。ZFN 是一种人工改造而成的核酸内切酶,由锌指蛋白的 DNA 结合域和核酸内切酶 FokI 的切割结构域组成。其中,DNA 结合域通常由 3—6 个锌指结构域 (Zincfingerdomain,ZFD) 串联而成。一个 ZFD 能特异识别 DNA 链上 3 个连续的核苷酸碱基,多个 ZFD 串联后就能特异识别较长的核苷酸序列。FokI 是一种非特异性核酸内切酶,在二聚体状态时才有内切酶活性。因此,实际应用 ZFNs 打靶时,需要在靶点的两侧各设计一个 ZFN。待 2 个 ZFN 结合到结合位点后,2 个 FokI 相互作用形成二聚体,从而在靶点处切割 DNA,产生 DSBs。2002 年,ZFNs 首次成功应用于果蝇内源基因的定向突变。迄今为止,ZFNs 技术已成功应用于人类干细胞、大鼠、果蝇、斑马鱼、拟南芥、烟草和玉米等生物体的基因组编辑。
在作物改良方面,ZFNs 技术迄今仅在玉米性状改良上有一例报道,即 2009 年《Science》报道 SHUKLA 等利用 ZFNs 技术靶向突变玉米 IPK1,获得低植酸含量的玉米突变体。植酸盐是谷物饲料的抗营养组分,低植酸盐含量的谷物饲料能降低来自动物饲养作业所产生废物流中的磷酸盐污染。由于 ZFNs 的构建难度较大,普通分子实验室难以操作,且成本高,预测其很难广泛应用于植物基因组编辑。
1.2 TALENs 技术及其应用
TALENs 是继 ZFNs 之后的第二代基因组编辑技术。TALEN 结构与 ZFN 类似,也由 DNA 结合域和 FokI 的切割结构域融合而成。TALEN 的 DNA 结合域为天然的或经改造的 TAL 效应子 (TALeffector,TALE) 蛋白结构域。天然的 TALEs 都具有高度统一的结构:即高度保守的 N 端和 C 端、中间部分的重复区。不同 TALEs 之间的差异主要在中间的重复区,其由 33—35 个氨基酸的重复单元组成,各重复单元除了第 12 和第 13 位氨基酸高度可变外,其他都高度保守,而这两个高度可变的氨基酸组合被称为重复可变区 (repeat-variablediresidue,RVD)。2009 年,BOCH 等和 MOSCOU 等相继破解 RVDs 与核苷酸碱基之间的识别密码 (即 NI 或 NS—A,NG—T,NN 或 NK—G 和 HD—C),由此打开了人工构建靶向基因组任意位点 TALENs 的大门。TALEN 的工作原理也与 ZFN 相似,TALE 重复区的每个重复单元能特异识别一个特定的核甘酸碱基,多个重复单元串联后就能特异识别较长的核苷酸序列。实际利用 TALENs 打靶时,同样需要在靶点两侧设计一对 TALENs。与 ZFN 相比,TALEN 载体的构建相对简单、成本也更低、普通的分子生物实验室都能操作;此外,TALEN 技术基于重复单元与核苷酸碱基一对一的识别模式,特异性更强,靶向编辑的效率更高。因此,自 2010 年 TALENs 技术出现后,便迅速取代 ZFNs 成为新一代的基因组编辑技术。
MLO(MILDEW-RESISTANCELOCUS) 是大麦、小麦等作物中白毛病菌成功侵染所必需的基因,该基因突变后,大麦表现出对白毛病菌的持久、广谱抗性。2014 年,中国科学院遗传与发育生物学研究所高彩霞团队利用 TALENs 技术同时靶向突变小麦 MLO 的 3 个拷贝,获得了 3 个拷贝都突变的纯合突变体,该突变体小麦对白毛病菌表现极为显著的抗性,证明 TALENs 技术能对小麦这样的多倍体植物进行定向遗传改良。同年,美国 Cellectis 公司研究人员通过 TALENs 技术靶向突变大豆脂肪酸脱氢酶基因 FAD2-1A 和 FAD2-1B,获得的双基因突变体中油酸含量由 20% 提高到 80%、亚油酸含量由 50% 降至 4%,极大地改善了大豆油的品质;随后,该公司又通过 TALENs 技术靶向修饰马铃薯的 VInv,改良了马铃薯的耐冷藏和加工性。此外,SHAN 等利用 TALENs 技术敲除水稻品种日本睛的 OsBADH2,使无香味的稻米产生了香味;MA 等利用该技术敲除水稻脂肪氧化酶基因 Lox3,改良了水稻种子的耐贮藏性;BLANVILLAIN-BAUFUME 等利用该技术对水稻白叶枯病感病基因 SWEET14 启动子中效应蛋白 AvrXa7 和 Tal5、TalC 的 EBEs 进行定点编辑,使感病水稻品种的白叶枯病抗性提高到中抗以上水平。
1.3 CRISPR/Cas9 技术及其应用
CRISPR/Cas 是细菌和古细菌抵御病毒和外源 DNA 入侵的适应性免疫系统。目前,CRISPR/Cas 系统可分为两大类。第一大类由多个 Cas 蛋白组成的复合体行使生物学功能,第二大类由单个 Cas 蛋白 (如 Cas9、Cpf1、C2c1 和 C2c2) 行使生物学功能。CRISPR/Cas9 属于 II 类,仅需要成熟的 crRNA(CRISPR-derivedRNA)、tracrRNA(trans-activatingRNA) 和 Cas9 蛋白就能实现对外源 DNA 的切割。
经人工改造的 CRISPR/Cas9 系统主要由引导 RNA(singleguideRNA,sgRNA) 和 Cas9 蛋白结构域组成。其中,sgRNA 起精确定位作用,其 5′端前 20 个核苷酸碱基决定 Cas9 蛋白特异性切割基因组 DNA 的部位;Cas9 蛋白包含类 HNH 核酸酶结构域和类 RuvC 核酸酶结构域,分别负责切割靶 DNA 互补链和非互补链 DNA。CRISPR/Cas9 系统在工作时,sgRNA 和 Cas9 形成一个嵌合蛋白,该嵌合蛋白切割与 sgRNA5′端前 20-nt 互补的基因组 DNA 双链,产生 DSBs,最后通过 HDR 或 NHEJ 修复途径产生基因突变。2013 年,CPISPR/Cas9 系统成功实现人类细胞的基因组定向编辑,随后被迅速应用于人类、动物和植物等的基因组编辑,并超越 TALENs 成为第三代基因组编辑技术。
CRISPR/Cas9 系统具有构建简单、编辑效率高、容易实现多基因编辑等优势,现已成为应用最广泛的基因组编辑技术,在作物遗传改良和品种培育上具有重大应用潜力。目前,CRISPR/Cas9 技术已成功应用于多种作物如水稻、玉米、小麦、大豆、番茄、柑桔和蘑菇的重要农艺性状遗传改良。
1.4 CRISPR/Cpf1 技术及其应用
CRISPR/Cpf1 隶属于 II 类 typeV-ACRISPR 系统,是迄今发现最简单、有效的 II 类 CRISPR/Cas 系统。CRISPR/Cpf1 特异切割 DNA 双链的原理与 CRISPR/Cas9 相似,但其作用机制不同。Cpf1 是 Cas 蛋白的一种,其功能与 Cas9 类似,目前 AsCpf1(Acidaminococcussp.Cpf1)、LbCpf1(LachnospiraceaebacteriumCpf1) 和 FnCpf1(FrancisellanovicidaCpf1) 已被证实具有 DNA 切割及执行 RNA 加工的活性。Cpf1 蛋白由具有识别 sgRNA 功能的 a-REC 区域和具有核酸酶功能的 NUC 区域构成。a-REC 包含 REC1 和 REC22 个识别结构域;NUC 主要包含 RuvC 结构域、WED(wedge) 结构域、一种推定的核酸酶 (Nuc) 结构域和 PI(PAM-interacting) 结构域,其中,Ruv-C 和 Nuc 核酸酶分别负责切割靶 DNA 的非互补链及互补链的不同位点,由此产生具粘性末端的 DSBs。不同于 SpCas9(StreptococcuspyogenesCas9),Cpf1 仅需一个 42-nt 的 crRNA(3′端有 23-nt 与靶 DNA 序列互补),就能对靶 DNA 双链进行切割。另 AsCpf1 和 LbCpf1 的 PAM 识别区碱基为 5′-TTTN-3′,FnCpf1 的 PAM 识别区碱基为 5′-TTN-3′,不同于 SpCas9 的 5′-NGG-3′。此外,Cpf1 与 SpCas9 切割机制也不一样,SpCas9 切割靶 DNA 互补链的 4 位和非互补链的 16 位,形成一个平末端;而 Cpf1 切割靶 DNA 互补链的 23 位和非互补链的 18 位,形成一个 5-nt 的粘性末端。但最新的研究结果表明,Cpf1 并非只切割靶 DNA 非互补链的 18 位,而是在非互补链的 14—18 位间多个位点切割。当 spacer 序列长度≥20-nt 时,Cpf1 倾向于切割互补链的 18 位;当 spacer 序列长度 <20-nt 时,Cpf1 倾向于切割互补链的 14 位。此外,研究人员还证明 Cpf1 核酸酶在人类细胞中与 SpCas9 具有相当的切割效率,也证实 Cpf1 核酸酶在人类细胞中具有高度的特异性。
目前,CRISPR/Cpf1 技术已成功应用于烟草、大豆和水稻的基因组编辑。2016 年,ENDO 等首次将 CRISPR/Cpf1 系统成功移植于烟草和水稻的基因组编辑,他们将 FnCpf1 和 crRNA 融合开发了 Cpf1-crRNA 系统,在烟草和水稻中的编辑效率最高可达 90%。随后,XU 等将 LpCpf1 分别与 pre-crRNA 和加长 pre-crRNA 融合构建了 crRNA-Cpf1 系统,在水稻中的编辑效率最高可达 41.2%;HU 等也将 LpCpf1 和 crRNA 整合开发了 CRISPR-Cpf1 系统,并利用该系统成功实现水稻内源基因的定点编辑;KIM 等开发了 Cpf1–RNP(ribonucleoprotein) 系统,并成功利用该系统对大豆和烟草的内源基因进行定点突变。此外,BEGEMANN 等将 Cpf1、crRNA 和供体片段构成融合表达载体,对水稻叶绿素 a 加氧酶基因 OsCAO1 进行定点编辑,获得了在靶点产生的预期 InDel 突变和片段插入,此外纯合突变植株整株都产生黄化现象。TANG 等针对 Cpf1 蛋白及 crRNA 的表达特性,构建了 PolII 型启动子融合核酶 (ribozyme) 驱动的 Cpf1 和 crRNA 植物表达单元,并成功对水稻内源基因 OsPDS、OsDEP1 和 OsROC5 进行定点突变。WANG 等将 4 个 DR(directrepeats)-guide 单元组成的 crRNA 分别与 FnCpf1 和 LbCpf1 融合构建 CRISPR/Cpf1 系统,并利用该系统简单、高效地实现了水稻多基因定点编辑。
CRISPR/Cpf1 与 CRISPR/Cas9 一样,都是目前最为简捷、高效和最易实现多基因编辑的基因组编辑技术。两者表达载体的构建都相当简单,且都只需要串联多个 crRNA 即可实现多位点编辑,而编辑效率和特异性也都相当,另因都受 PAM 识别位点的限制,编辑范围也都有一定的局限性。因此,CRISPR/Cpf1 与 CRISPR/Cas9 可以说是不相伯仲的基因组编辑技术,相当于 CRISPR/Cas 技术 “厨房” 两把效果一样但又能互补的 “剪刀”,作为 “厨师” 的我们,只要能达到我们的目标,选择任一 “剪刀” 都可。
1.5 利用 CRISPR/Cas 系统在基因组编辑方式上的技术创新
1.5.1 植物基因组单碱基编辑系统创新和应用
单碱基突变可引起作物许多农艺性状的改变,因此,实施单碱基编辑对作物遗传改良具有十分重要的意义。2016 年 5 月,《Nature》报道了 KOMOR 等将大鼠胞嘧啶脱氨酶 (APOBEC1,一种碱基修饰酶能改变 DNA 序列)、Cas9 变体 (D10A) 和尿嘧啶糖基化酶抑制剂 (UGI) 融合构建了大鼠基因组单碱基编辑系统,并用该系统成功纠正了小鼠细胞中阿尔茨海默病相关的突变,单碱基替换效率高达 75%。随后,有 4 个研究团队将该系统成功改造并用于粮食和蔬菜作物 (水稻、小麦、玉米、番茄) 的基因组单碱基编辑。
2016 年 12 月,中国科学院上海生命科学研究院朱健康团队,以及夏兰琴和赵云德合作团队同时在《MolecularPlant》上发表了其在植物基因组单碱基编辑上取得的最新成果。朱健康团队将 APOBEC1 通过非结构化的 16 残基肽 XTEN 作为接头,融合到 Cas9(D10A) 的 N 末端,并将核定位信号 (NLS) 肽添加到 Cas9(D10A) 的 C 末端,构建了 APOBEC1-XTEN-Cas9(D10A) 植物基因组单碱基编辑系统。利用该系统对水稻 NRT1.1B 和 SLR1 进行编辑,结果表明该系统能在靶位点产生预期的 C→T(G→A) 碱基替换,NRT1.1B 和 SLR1 在靶位点产生预期突变的效率分别为 2.7% 和 13.3%。夏兰琴和赵云德合作团队也利用 APOBEC1、Cas9 变体 (nCas9-D10A) 和 UGI 的融合基因 BE3 构建了植物基因组单碱基编辑系统 pCXUN-BE3。利用 pCXUN-BE3 系统编辑水稻八氢番茄红素脱氢酶基因 OsPDS 和淀粉分支酶 IIb 基因 OsSBEIIb,获得在 3 个靶位点产生预期突变 (靶序列 5′端 4—8 位置有 G→A 或 C→T) 的水稻植株,效率最高可达 20%。
2017 年 2 月,《NatureBiotechnology》杂志报道了高彩霞实验室在水稻、小麦和玉米基因组中实现高效、精确的单碱基编辑的成果。他们同样利用 APOBEC1、nCas9-D10A 和 UGI,构建了植物基因组单碱基编辑系统 nCas9-PBE。利用该系统对 3 种作物 5 个内源基因 7 个靶位点的定点突变结果显示,在靶序列 5′端 3—9 位置能产生预期的 C→T 的替换,其中,单个 C 的替换效率为 0.39%—7.07%,多个 C 的替换效率高达 12.48%;而遗传转化的结果表明,该系统在小麦、水稻和玉米中获得靶区域单碱基替换的效率最高可达 43.48%。同年 3 月,SHIMATANI 等在《NatureBiotechnology》杂志发表了其在水稻和番茄中实现高效的单碱基编辑的最新成果。将 nCas9-D10A(nCas9)、PmCDA1(Petromyzonmarinuscytidinedeaminase) 和 sgRNA 融合构建了植物基因组单碱基编辑系统 nCas9-PmCDA1,并利用该系统对水稻 ALS 和 FTIP1e 以及番茄 DELLA 和 ETR1 进行定点突变。结果表明,在水稻中该系统在靶区域诱导单个或多个预期单碱基替换 (C→T) 的效率最高可达 50%,在番茄中最高可达 80%。
1.5.2 DNA-free 植物基因组编辑系统创新和应用
生物安全问题是限制转基因作物商业化的主要因素。虽然 CRISPR/Cas 对基因进行定点修饰后能通过自交后代分离剔除外源 DNA,获得无外源 DNA 基因 (DNA-free) 的编辑植株。但目前只有美国对 CRISPR 编辑作物安全实行监管豁免,其他国家都还处于观望状态。因此,建立全程 DNA-free 的植物基因组编辑系统对于推动基因编辑作物的商业化利用具有重要意义。目前,DNA-free 植物基因组编辑系统主要有瞬时表达 CRISPR/Cas9 编辑系统和 RGEN(RNA-guidedengineerednuclease) 核糖核蛋白 (RGENribonucleoproteins,RGENRNPs) 编辑系统。
瞬时表达 CRISPR/Cas9 编辑系统的操作流程和原理是:将 CRISPR/Cas9 质粒 DNA(TECCDNA) 或其转录的 RNA(TECCRNA) 通过基因枪法直接转入植物愈伤组织,因是环状质粒或 RNA,故不易被整合进植物基因组中;在完成切割使命后,质粒 DNA 或 RNA 会被细胞内源核酸酶分解,从而实现全程 DNA-free 的基因组编辑。研究人员对该系统在小麦基因组中的定向修饰特性进行了详细的研究。通过对 4 个小麦品种的 7 个内源基因 (共 9 个靶位点) 靶向修饰发现,TECCDNA 在小麦 T0 转基因植株中诱导突变的效率为 1.0%—9.5%,DNA-free 突变植株的比例为 43.8%—86.8%。对比分析显示,TECCDNA 和经典 CRISPR/Cas9 系统的定向编辑效率和脱靶效应上没有明显差别,但都显著高于 TECCRNA。
RGENRNPs 技术是将 CRISPR-Cas 蛋白和 gRNA 在体外组装成核糖核蛋白复合体,该复合体进入细胞后能迅速行使 DNA 切割功能,然后被细胞内源蛋白酶快速分解,从而实现全程无外源 DNA 整合的基因组编辑。RGENRNPs 技术最先应用于低等动物和人类细胞,随后被应用于拟南芥、烟草、莴苣、水稻、矮牵牛花、玉米、小麦和大豆的基因组编辑。2015 年,WOO 等首先将 RGENRNPs 技术成功用于植物的基因组编辑。研究表明,RGENRNPs 在拟南芥、烟草和水稻的原生质体中定向编辑的效率分别为 16%—19%、17%—44% 和 8.4%—23%,在转化莴苣的再生植株中定向编辑的效率为 45.7%,且没有检测到脱靶突变。此外,SUBBURAJ 等通过 RGENRNPs 技术对矮牵牛花内源基因 PhNR(nitratereductase) 的 6 个位点 (NR1-6) 进行定向修饰,发现在原生质体中 RGENRNPs 诱导定点突变的平均效率为 (11.5±2)%。显然,这两项研究只聚焦在植物原生质体层面的基因组定向修饰,还未真正涉及作物的遗传改良。
2016 年 11 月,《NatureCommunications》报道了美国杜邦先锋公司利用 RGENRNPs 技术获得了无叶舌、雄性不育和抗氯磺隆的玉米改良系,标志着 RGENRNPs 技术在主要作物遗传改良上的真正应用。研究人员通过基因枪法将组装好的 RGENRNPs,及 RGENRNPs 和同源供体片段转入玉米愈伤组织,通过 NHEJ 途径实现对玉米无叶舌基因 (LIG)、雄性育性基因 (Ms26 和 Ms45) 进行定点突变,通过 HDR 途径实现对 ALS2 基因定点替换 (编码区的第 165 位脯氨酸突变为丝氨酸),最终获得了全程无外源 DNA 作为载体的 DNA-free 基因编辑玉米,表现为无叶舌、雄性不育和抗氯磺隆;对比分析发现,RGENRNPs 和 Cas9-gRNA 质粒 (即 TECCDNA) 定向编辑效率基本一致,但 RGENRNPs 诱导的脱靶效应显著低于 Cas9-gRNA 质粒诱导的。类似地,LIANG 等利用 RGENRNPs 技术对小麦 TaGW2 进行定向编辑,深度测序结果表明 CRISPR/Cas9RNP 定向编辑的效率较 CRISPR/Cas9DNA 要低,脱靶的机率也更低,且在突变植株中未检测到脱靶现象 .
上述报道都是基于 CRISPR/Cas9RNP 的 DNA-free 植物基因组编辑。2017 年,KIM 等首次利用 CRISPR/Cpf1RNP 成功对大豆 FAD2 和烟草 AOC 实现定点编辑,效率最高达 11.7%,且在大豆基因组潜在脱靶位点未检测到显著突变。
来源:《中国农业科学》 2018,51(1):1-16