SLAF-seq技术:大规模基因分型高通量策略
互联网
17792
大规模基因分型在遗传相关性研究中起着重要作用,尤其是和高通量测序技术结合后,它为基因挖掘带来了新的机遇。大规模基因分型的有效解决方法:SLAF-seq(specific-locus amplified fragment sequencing),该技术前期利用生物信息学方法,对目标物种的参考基因组(或已知BAC序列)进行系统分析,设计标记开发方案,后期根据前期的方案,构建SLAF-seq文库,筛选特异性长度片段进行高通量测序,将获得测序深度和质量满足要求的SLAF片段来代表目标物种的全基因组信息。它具有以下优势:
1. 高深度测序保证分型准确;
2. 简化的策略降低测序成本;
3. 前期简化方案预测保障标记数量最优;
4. 采用double index技术能适用于大群体。
2. 简化的策略降低测序成本;
3. 前期简化方案预测保障标记数量最优;
4. 采用double index技术能适用于大群体。
在文章中,我们用水稻和大豆数据验证了SLAF-seq技术的有效性,2个结果都显示预测的和实际的结果一致性非常高,同样基因分型也非常准确。我们还利用这个技术构建了鲤鱼的高密度遗传图谱(物种高杂合且无参考基因组),对2个亲本和211个子代进行简化测序,得到50,530个SLAF标签,构建了50条连锁群,上图标记5885,标记平均距离0.68 cM。将得到的连锁群与鲤鱼的近缘物种斑马鱼进行比较基因组学研究,结果显示鲤鱼的2条连锁群对应斑马鱼的1条染色体,二者的共线性良好,说明高质量的SLAF分型结果。
SLAF-seq技术为大规模基因分型提供了一个高通量的策略,并且能应用于不同物种和群体,适用性非常广。SLAF标记的质量评估部分的基本原理,采用基因分型质量评分对标记质量进行评估。质量评分采用PHRED 公式的算法,错误率数据用新的方法得到。文章中,对于SLAF简化效率的评估方式和结论,通过仿真研究发现4 以上的测序深度能够保证SLAF的准确性。对鲤鱼4.28×的测序进一步验证,SLAF的质量能将错误率控制在3%(5/190)以内。
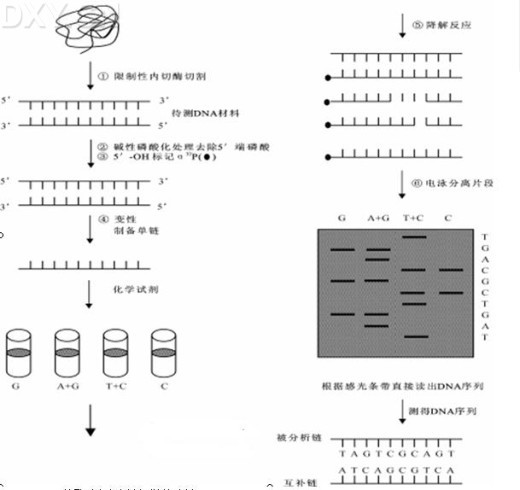
SLAF的技术流程:
(1)预实验:用rice 和soybeans 等training data确定酶和酶切片段大小。
(2)构建文库:包括酶切,连接,PCR反应,凝胶电泳过程。
(3)基因分型:采用配对双末端测序获得短片段DNA序列信息,用软件定义和评估基因型。

下图所反映的分析流程展示如何用质量评分选择高质量遗传标记和个体的动态优化过程。首先对每个SLAF和个体计算低质量的标记。删除标记质量最差的SLAF和个体。重复以上过程,直到所有SLAF标记的平均质量分达到13。通过计算标记或者个体的低质量基因型数量,反复删除标记或者个体,最终使标记平均质量值达到阈值,文章中最终得到7559个标记和160个体。

SLAF标记的质量评估部分的基本原理是贝叶斯方法验证分型准确性,通过基因型覆盖度和多态性计算个体在某个标记位点拥有某个基因型的概率,综合考虑测序深度和测序错误率将每个基因型进行打分。以分值大小评估标记质量。文章中,对于SLAF简化效率的评估方式和结论,实际标记数和预测值尽量接近;SLAF均匀分布于基因组上;、避免重复序列。遗传图谱评估的方案和结果,研究重组断点,检查重组断点的准确性;比对同源物种,检查同源性。只有1.51%分型可疑,25.6%的标记唯一比对到斑马鱼基因组,且有良好的共线性。
SLAF技术结合了位点特异性扩增和高通量测序,可以用于全基因组范围的De Novo SNP开发和大范围的基因分型。为了提高片段选择的高效性,生物信息统计模型被开发出来,用于进行特定位点选择的扩增,这种是基于training 数据的。它包含稀有序列BAC序列和基因组草图序列。这种training 数据事从靶标组织或者其他与进化相关的组织,甚至是GC含量相似的组织中提取的。SLAF高效选择模式能被发展为training 数据,我们分析了几个training 数据组,基因组内重复序列可以被高效的避免。