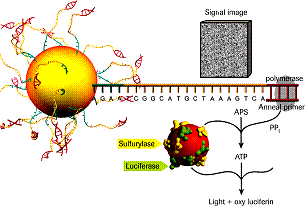
454高通量测序-研究土壤微生物的新手段
互联网
在陆地生态系统中,在土壤中生活有数量庞大的微生物种群,包括原核微生物如细菌、蓝细菌、放线菌及超显微结构微生物, 以及真核生物如真菌、藻类( 蓝藻除外) 、地衣等。它们与植物和动物有着明确的分工,主要扮演“分解者”的角色,几乎参与土壤中一切生物和生物化学反应,担负着地球C、N、P、S 等物质循环的“调节器”[1 ]、土壤养分植物有效性的“转化器”和污染环境的“净化器”等多方面生态功能[2 ]。土壤微生物是气候和土壤环境条件变化的敏感指标,土壤微生物群落结构和多样性及其变化在一定程度上反映了土壤的质量。土壤微生物多样性一般包括微生物分类群的多样性、遗传(基因) 多样性、生态特征多样性和功能多样性[3 ]。由于土壤微生物的复杂性、土壤本身的多变性和研究方法不完善等原因的限制, 以往人们对土壤微生物多样性的研究与动、植物相比远远落后。随着多聚酶链反应(PCR)、核酸测序等现代生物学分子生物学技术的迅速发展, 人们对土壤微生物多样性有了更多的了解;高通量测序技术的发展则为研究土壤宏基因组提供了大量数据,为直接探究土壤中的微生物群落结构提供了客观而全面的信息。
一、对土壤微生物进行研究的方法
1、微生物平板培养法
传统的土壤生态系统中微生物群落多样性及结构分析大多是将微生物进行分离培养,然后通过一般的生物化学性状,或者特定的表现型来分析,局限于从固体培养基上分离微生物。
这种方法只限于极少量(0.1%-1%)可以培养的微生物类群,无法对绝大多数微生物的分类地位和系统发育关系的深入研究。
2、分子生物学技术结合测序的方法
在过去的20多年里,分子生物学技术,尤其16SrDNA技术已经广泛应用于鉴定未知菌的研究中。20世纪80年代以来, 逐步建立起了以分子系统发育分析为基础的现代微生物分子生态学的研究方法, 如PCR-RFLP、PCR-RAPD、PCR-SSCP、荧光原位杂交技术(FISH)、基因芯片(Microarry) 、磷脂脂肪酸图谱分析( Phospholipid fatty acid, PLFA) 、稳定同位素探针( Stable Isotope Probing, SIP) 、PCR-DGGE/TGGE (Denaturing gradient gel electrophoresis, DGGE/Temperature gradient gel electrophoresis, TGGE) 等, 使得研究者能够在分子水平上对土壤微生物多样性进行研究。
其中运用比较广泛并被普遍认可的是PCR-DGGE/TGGE法。DGGE/TGGE 的局限性在于,检测的DNA片段长度范围以200bp ~900bp为佳,超出此范围的片段难以检测[4],PCR 扩增所需G/C 碱基对含量至少达40 % ,通常只能检测到环境中优势菌群的存在,只有占整个群落细菌数量约1%或以上的类群能够通过DGGE检测到[5]。TGGE 仪器所允许的温度范围为15 ℃~80 ℃,较大片段DNA 的Tm 值因为接近80 ℃而使检测难度提高;若电泳条件不适宜,不能完全保证将有序列差异的DNA片段分开,从而出现序列不同的DNA 迁移在同一位置的现象。
3、高通量测序方法
近年来,16S rRNA/DNA为基础的分子生物学技术已成为普遍接受的方法[6,7 ]。研究表明,400~600碱基的序列,足以对环境中微生物的多样性和种群分类进行初步的估计[8],因此454高通量测序的方法因其读长(400~500bp)长和准确性高的特点大量用于微生物多样性的研究。
有了微生物16S rDNA序列,不论是全长还是部分,都可以提交到GenBank采用BLAST程序与已知序列进行相似性分析。Gen Bank将按照与测得序列的相似性高低列出已知序列名单、相似性程度以及这些序列相对应的微生物种类,但更为精确的微生物分类还取决于系统发育分析(phylogenetic analysis)。
高通量测序的优越性体现在:测序序列长,可以覆盖16S /18S rDNA、ITS等高变区域; 测序通量高,可以检测到环境样品中的痕量微生物;实验操作简单、结果稳定,可重复性强;无需进行复杂的文库构建,微生物DNA扩增产物可以直接进行测序,实验周期短;测序数据便于进行生物信息分析。
该方法得到顶级期刊的认可(Nature等),已成为土壤微生物多样性检测的重要手段。上海美吉生物公司已有若干成功案例,为客户提供的土壤样本进行高通量测序并进行生物信息学分析。高通量测序因其数据量大,操作过程简单,尽可能避免了繁琐的实验操作过程所造成的样本损失,能够相对客观的反应出土壤样本的真实情况。
二、高通量测序在土壤微生物研究中的应用
1、 研究土壤微生物的物种多样性
微生物物种多样性主要从对微生物类群即细菌、真菌和放线菌这三大类群的数量及其比例组成来描述微生物多样性,或者按照微生物在生态系统中的作用将其划分成不同的功能群(function group),通过某一功能群中物种的分类及其数量来研究土壤微生物多样性,如对土壤中的产甲烷细菌、固氮菌、根瘤菌等的多样性进行研究。以下是几篇具有代表性的文章。
Roesch等[9]采用454测序法对西半球的一个大的横断面的4类土壤进行检测和统计学评价。结果表明,这4种土壤中,最丰富的微生物类群拟杆菌纲、β-变形菌纲和α-变形菌纲。与农业土壤相比,森林土壤的微生物多样性更为丰富,然而检测结果表明森林土壤中的古生菌多样性较少,仅为该位点所有序列的0.009%,而农业土壤的比例则为4% -12%。
土壤中细菌的多样性差距非常大,因土壤结构的不同土壤中的细菌群体也呈现出多样化。Triplett等[10]基于焦磷酸测序法来对16S rRNA的V2-V3区域进行测序,估测9个草地土壤中的菌群的整体和垂直特性。对所有752,838条数据序列进行聚类分析,探索菌群在丰度、多样性和组成成分等方面的特异性。作者发现在不同的土壤层中,细菌系统发生的种群或者亚群的不同分配是与土壤的性质有关的,包括有机碳含量、总含氮水平或者微生物生物量。
2、研究土壤微生物功能多样性
土壤微生物功能多样性指包括微生物活性、底物代谢能力及与N、P、S 等营养元素在土壤中转化相关的功能等, 通过分析测定土壤中的一些转化过程, 如有机碳、硝化作用以及土壤中酶的活性等来了解土壤微生物功能。
氨氧化反应是硝化作用的第一步,也是全球氮循环中由微生物活动形成硝化盐的重要进程。Leininger[11]检测了3个气候区域12块原始和农业用地的土壤里编码氨单加氧酶(amoA)的一个亚基的基因丰度。采用反向转录定量PCR研究及无需克隆的焦磷酸测序技术对互补DNA测序,证实古细菌的氨氧化活性要远高于细菌,证实Crenarchaeota可能是土壤生态系统中最富有氨氧化活性的微生物。
Urich等[12]采用基于RNA的环境转录组学方法同时获得土壤微生物种群结构和功能信息。结果认为,通过该方法可以同时研究微生物生种群结构与功能从而避免其他方法所造成的偏差。群落基因组学分析可以通过研究微生物基因组序列与某些表达特征之间的关系,获得一些微生物功能方面的信息。但同时也需要运用其他方法将特定功能与具有这种特定功能的微生物群落结构对应起来。对rRNA表达基因和与环境因素相关的主要酶类的基因进行定量化和比较分析,将能了解微生物结构与特定功能之间的关系,如硝化、反硝化和污染物降解。
反硝化作用是参与到氮流失和温室气体排放等氮循环过程的重要流程之一。通过宏基因组测序的方法,结合分子检测和焦磷酸测序,Ryan等分离鉴定了操纵编码反硝化过程的酶类。通过筛选77,000个土壤宏基因组文库中得到的克隆,最终分离并鉴定了9个参与反硝化作用的酶簇[13]。
3、 研究环境的突然变化对土壤微生物菌群的影响
环境的突然改变会导致微生物群落的结构和功能发生变化。Zachary等[14]以重水稳定同位素探测技术(H218O-SIP)鉴定与土壤增湿相关的细菌。通过液相色谱/质谱(LC-MS),作者确定H218O中的氧原子结合到了所有的DNA结构成分中。尽管这种结合不是均匀的,还是可以明显的将标记了18O和未标记的DNA区分开来。作者发现DNA和细胞外的H2O中的氧原子在体外没有发生交换,表明掺入DNA的18O是相对稳定的,并且掺入到细菌DNA中的18O的比例较高(48-72h)。土壤增湿后,对土壤中16S rRNA进行高通量测序,发现Alphaproteobacteria、Betaproteobacteria和Gammaproteobacteria的相对比例升高,而Chloroflexi和Deltaproteobacteria的比例则降低。作者通过控制土壤湿度的动态变化,对微生物菌群的结构发生变化进行研究和生态类群的划分。
注:已用454焦磷酸测序法发表的文章总结
1 Archaea predominate among ammonia-oxidizing prokaryotes in soils -- (2006) Nature 442: 806-809--if 36.101
2 454 Pyrosequencing analyses of forest soils reveal an unexpectedly high fungal diversity. --(2009) New Phytologist 184(2): 449-56.--if 6.516
3 Vertical distribution of fungal communities in tallgrass prairie soil.--(2010) Mycologia 102(5): 1027-41.--if 6.2
4 A comprehensive survey of soil acidobacterial diversity using pyrosequencing and clone library analyses. --(2009) ISME Journal 3(4): 442-53--if 6.153
5 Pyrosequencing enumerates and contrasts soil microbial diversity --(2008) The ISME Journal 1: 283-290--if 6.153
6 Bacterial diversity in rhizosphere soil from Antarctic vascular plants of Admiralty Bay, maritime Antarctica. --(2010) ISME Journal 4(8): 989-1001.--if 6.152
7 Effect of storage conditions on the assessment of bacterial community structure in soil and human-associated samples. --(2010) FEMS Microbiology Letters 307(1): 80-6.--if 6.152
8 Soil bacterial and fungal communities across a pH gradient in an arable soil. --(2010) ISME Journal ePub:--if 4.42
9 Examining the global distribution of dominant archaeal populations in soil. -- (2010) ISME Journal ePub: --if 4.42
10 Simultaneous assessment of soil microbial community structure and function through analysis of the meta-transcriptome -- (2008) PloS One 3:--if 4.411
11 Characterization of trapped lignin-degrading microbes in tropical forest soil. --(2011) PLoS One 6(4): e19306.--if 4.411
12 Impact of biochar application to soil on the root-associated bacterial community structure of fully developed greenhouse pepper plants. --(2011) Appl Environ Microbiol ePub:--if 3.8
13 Validation of heavy-water stable isotope probing for the characterization of rapidly responding soil bacteria. --(2011) Appl Eviron Microbiol ePub:--if 3.8
14 Identification of Nitrogen-Incorporating Bacteria in Petroleum-Contaminated Arctic Soils Using 15N DNA-SIP and Pyrosequencing. --(2011) Appl Environ Microbiol ePub: --if 3.8
15 Pyrosequencing-based assessment of bacterial community structure along different management types in german forest and grassland soils. --(2011) PLoS One 6(2): e17000.--3.8
16 Assessment of soil fungal communities using pyrosequencing. --(2010) Journal of Microbiology 48(3): 284-9.--if 3.8
17 Disclosing arbuscular mycorrhizal fungal biodiversity in soil through a land-use gradient using a pyrosequencing approach. --(2009) Environmental Microbiology ePub:--if 3.778
18 Assessment of bias associated with incomplete extraction of microbial DNA from soil. Feinstein LM, Sul WJ, Blackwood CB. (2009) Appl Environ Microbiol 75(16): 5428-33.--if 3.778
19 Characterization of denitrification gene clusters of soil bacteria via a metagenomic approach. --(2009) Applied Environmental Microbiology 75(2): 534-7--if 3.778
20 Tag-encoded pyrosequencing analysis of bacterial diversity in a single soil type as affected by management and land use. --(2008) Soil Biology and Biochemistry 40: 2762-2770--if 3.242
21 Shifts in microbial community structure along an ecological gradient of hypersaline soils and sediments. --(2010) ISME Journal 4(6): 829-38.--if 2.039
22 Metagenomic comparison of direct and indirect soil DNA extraction approaches. --(2011)Microbiol Methods ePub: --if 2.018
23 Horizon-specific bacterial community composition of German grassland soils as revealed by pyrosequencing-based analysis of 16S rRNA genes. -- (2010) Appl Environ Microbiol 76(20): 6751-9.
参考文献
[1] Li F D. Prosperous Areas of Current Soil Microbiology. Acta Pedologica Sinica , 1993 , 30(3) : 229~236.
[2] Xu G H , Li Z G. Microbial Ecology. Nanjing : Southeast University Press , 1991.
[3] Luo S M. Agroecology. Beijing : Chinese Agriculture Press , 2001. 72~96.
[4] Fromin N , Hamelin J , Tarnawski S , et al . Statistical analysis ofdenaturing gel electrophoresis ( DGE) finger printing patterns [ J ] .Environ Microbiol , 2002 , 4 (11) :634 - 643
[5] Ferris M J , Muyzer G, Ward D M. Denaturing gradient gel electrophoresis profiles of 16S rRNA defined populations inhabiting a hotspring microbial mat community[J ] . Appl Environ Microbiol , 1996 ,62 :340 - 346
[6] Zhang T, Fang H H P. Biotechnol Lett, 2000, 22: 399-405.
[7] Wagner M, Arnann R, Lemmer H, et al. Appl Environ Microbiol, 1993, 59: 1520~1525.
[8] Andreas F, Arthur W, Robert L, et al. FEMS Microbiol Ecol, 1999,30:137~145.
[9] Pyrosequencing enumerates and contrasts soil microbial diversity. Luiz FW Roesch, Roberta R Fulthorpe, Alberto Riva, George Casella, Alison KM Hadwin, Angela D Kent, Samira H Daroub, Flavio AO Camargo, William G Farmerie and Eric W Triplett (2008) The ISME Journal 1: 283-290
[10] Assessment of soil fungal communities using pyrosequencing. Lim YW, Kim BK, Kim C, Jung HS, Kim BS, Lee JH, Chun J. (2010) Journal of Microbiology 48(3): 284-9.
[11] Archaea predominate among ammonia-oxidizing prokaryotes in soils S. Leininger, T. Urich, M. Schloter, L. Schwark, J. Qi, G. W. Nicol, J. I. Prosser, S. C. Schuster, C. Schleper (2006) Nature 442: 806-809
[12] Simultaneous assessment of soil microbial community structure and function through analysis of the meta-transcriptome Urich T, Lanzén A, Qi J, Huson DH, Schleper C, Schuster SC. (2008) PloS One 3:
[13] A comprehensive survey of soil acidobacterial diversity using pyrosequencing and clone library analyses. Jones RT, Robeson MS, Lauber CL, Hamady M, Knight R, Fierer N. (2009) ISME Journal 3(4): 442-53
[14] Validation of heavy-water stable isotope probing for the characterization of rapidly responding soil bacteria. Aanderud ZT, Lennon JT. (2011) Appl Eviron Microbiol


![十二烷基二甲基(3-磺丙基)氢氧化铵内盐 [用于生化研究],14933-08-5,≥98%,阿拉丁](https://img1.dxycdn.com/p/s14/2024/0619/475/6370229598169633081.jpg!wh200)