如何使用 NCBI 查找基因序列、mRNA、Promoter
丁香园
有不少人询问如何查询基因序列、如何进行引物设计、如何使用 BLAST 进行序列比对...... 其实这些问题在 NCBI 上都可以方便的找到答案。
我将结合我自己使用 NCBI 的一些经历跟大家交流一下 NCBI 的使用。主要有以下四部分内容:
如何查找基因序列、mRNA、Promoter
如何查找连续的 mRNA、cDNA、蛋白序列
运用 STS 查找已经公布的引物序列
如何运用 BLAST 进行序列比对、检验引物特异性
今天我们就先讲第一部分:如何查找基因序列、mRNA、Promoter。这里主要用到的是 Map viewer,我们以人的 IL6(白细胞介素 6)为例讲述一下具体的操作步骤。
1. 打开 Map viewer 页面,网址为:http://www.ncbi.nlm.nih.gov/mapview/index.html,在 search 的下拉菜单里选择物种,for 后面填写你的目的基因。操作完毕如下图所示。

2. 点击 GO 出现如下界面。


3. 在步骤 2 图示的右下角有一个 Quick Filter,在 Gene 前面的小方框里打勾,然后点击 Filter。出现下图:

染色体上的红色区域即为你的目的基因所处位置。下面参考序列给出了三个,是不同的部门做出来的。经我验证,序列有微小的差异,但总体来说基本相同。尽管你分别点击后,序列代码等有所差异,但碱基基本一致,不影响大家研究分析序列。
现在普遍采用的是最上面的那个序列,这一条是世界范围的生物科学家用计算机合成的 一个序列。我也推荐大家使用这个序列。
4. 点击上述三条序列第一条序列,即 reference 对应的 Genes seq,出现新的页面,页面如下图所示。

5. 点击上图出现的 Download/View Sequence/Evidence ,即下载查看序列等功能,结果如下图所示。
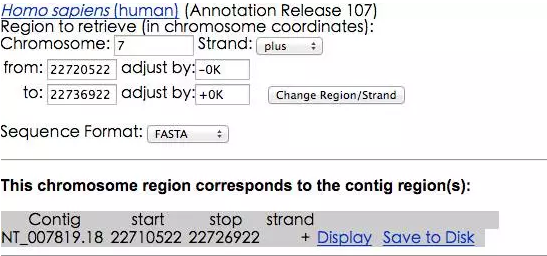
在 Sequence Format(序列输出格式)后面是一个下拉式选择菜单,默认的为 FASTA 格式,还有一个是 GenBank 格式。我推荐大家选择 GenBnak 格式,因为这个格式提供了很多该基因的信息,而 FASTA 格式只有基因序列。
6. 在 Sequence Format 后选择 GenBank,然后点击下面的 Display,目的基因的相关信息和序列就出现在眼前了。点击后如图所示,由于网页较大,只截取一小部分以作示范。
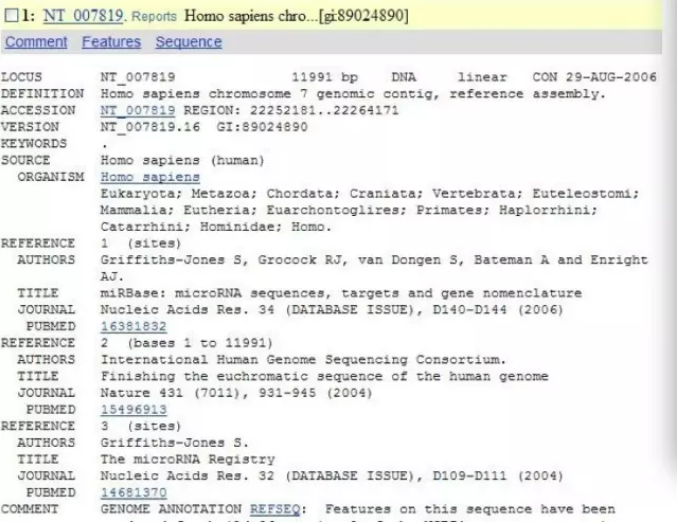
在上述打开的网页中,你可以看到基因长度、基因序列以及这个基因是如何被报道出来的等各种信息。
mRNA join(3598..3678,3841..4031,5090..5203,5911..6057, 7803..8394) ,这代表了从基因的 3598 位开始就是转录区了,即我们常说的 mRNA 片断,由于内含子的存在,所以 mRNA 在 DNA 序列上分成了几段。
CDS join(3660..3678,3841..4031,5090..5203,5911..6057, 7803..7970)
CDS 代表编码序列,即蛋白编码区是从 3660 开始( ATG),由于剪接作用所以 CDS 区也是不连续的。
promoter:转录起始位点前面是基因的调控区,启动子区没有明显的位置定义,大家也只是猜测它的大体位置。如果你要研究 promoter 区的话,建议你选择转录起始位点前的 2000 个碱基进行研究,一般默认的是这样。当然你如果觉得长度太长不好研究的话,也可以只研究 - 1000 到 0 这一千个碱基,因为一般情况下,启动子区的变异都在这个区域内。
怎么样,学会了吗?你不妨试试去找到自己的目的基因序列和启动子。