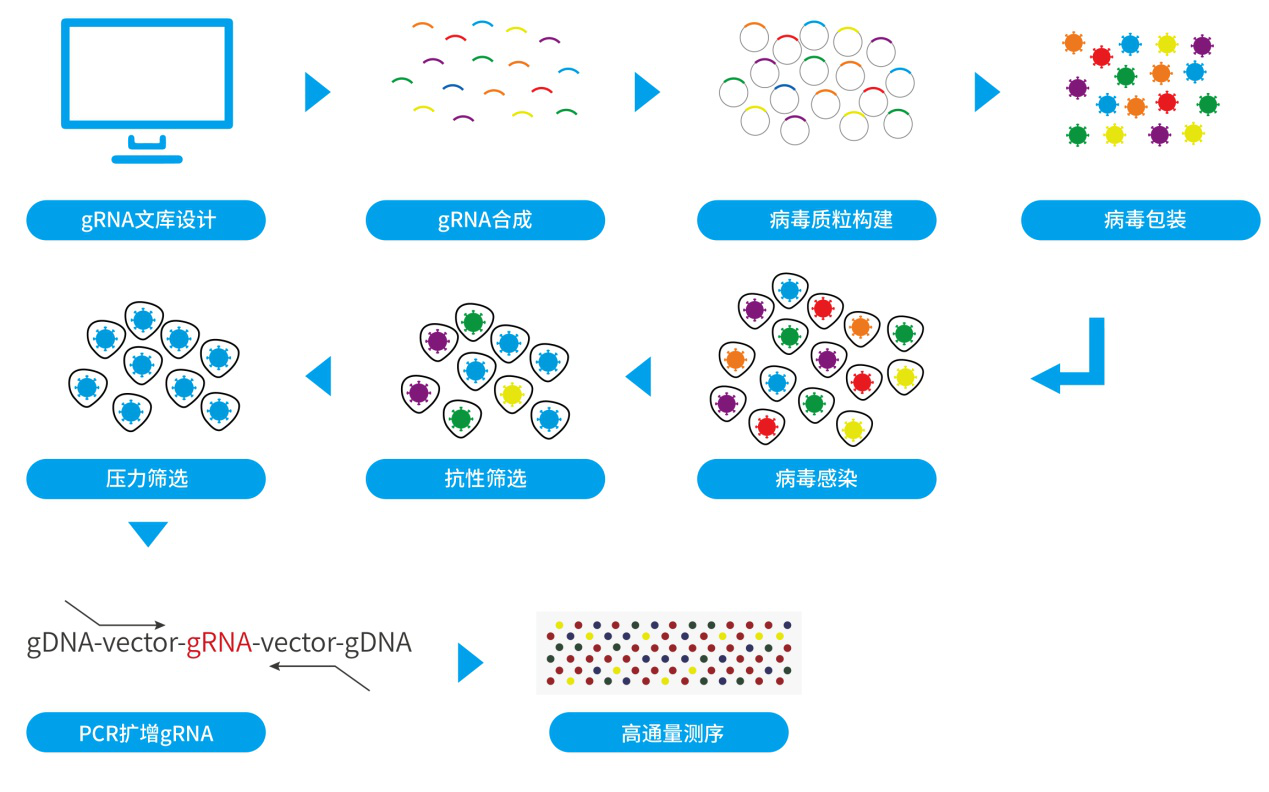
CRISPR-Cas9 全程实操教程:从 gRNA 设计到挑单克隆全程指导
科研小助手
基本原理
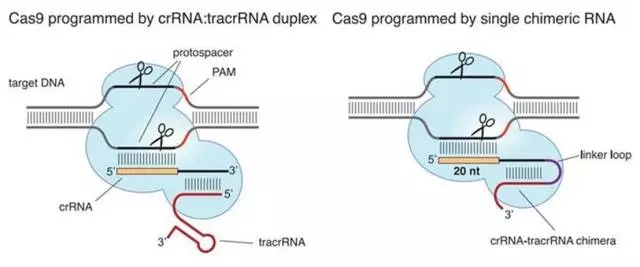
CRISPR-Cas9 是一种细菌获得性免疫,用于降解入侵的外源 DNA。CRISPR RNA (crRNA) 与转录激活 crRNA (Trans-activating crRNA, tracrRNA) 退火形成的复合物能特异性识别与其互补的基因组序列,引导 Cas9 核酸内切酶在目的片段生成 DNA 双链断裂,如下图所示。
通过基因工程手段对 crRNA 和 tracrRNA 进行改造,将其连接在一起得到 sgRNA (single guide RNA)。通过将表达 sgRNA 的原件与表达 Cas9 的原件相连接,得到可以同时表达两者的质粒,将其转染细胞,便能够对目的基因进行基因操作,在 PAM (5'-NGG) 上游~3bp 进行切割,如下图所示。
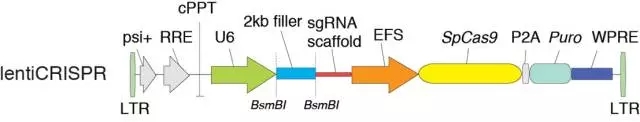
因此本实验的关键步骤是设计一对 20bp(不包括 NGG 在内的)完全互补的 oligo 插入到如上图所示的 filler。另 U6 启动子需要 5' 端的 G 起始转录,因此若设计的 oligo 第一个碱基非 G,需额外增加一个 G。
操作详细流程
1. 设计 sgRNA
关于 sgRNA 的设计我们有专门开过一篇文章《一文掌握 gRNA 的设计,常用方法汇总解析》
确定靶基因的序列,可通过在线网站设计 (http://tools.genome-engineering.org ),或根据靶基因的 CDS 序列自行设计,最方便的是在 human KO Library sgRNA 里选择。无论选择哪种方式,都建议进行一下 blast。
附上在线设计网页截图。输入基因的 name,填写 email address,设计结果稍后会发送到此邮箱,选择序列的类型以及物种。如 sequence type 选择 unique region,一次只能输入 23~500bp 的基因片段,最好一次只输入一个外显子,避免 guide 序列跨越内含子,都选择键入后点击提交,关注邮箱或者在线等。
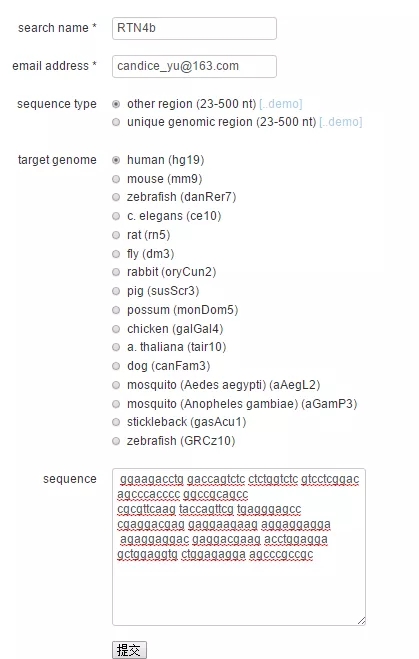
分析中,显示你所键入的基因序列里有 36 对可选的 guide 序列。
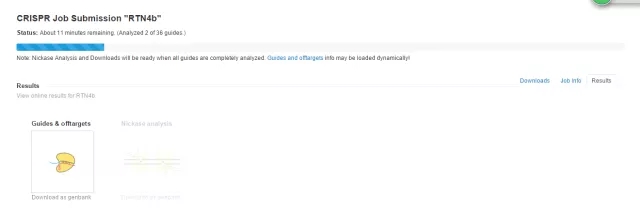
分析结束,点击 Download as genbank 查看结果,此时也会收到邮件。
选择分数较高的 Guide 序列,以 Guide#1 序列为例,2 条单链 oligo 的序列如下:oligo1:5'- caccG CGTTCAAGTACCAGTTCGTG - 3'; oligo2:5'- aaac CACGAACTGGTACTTGAACGc。标红部分与经 BsmBI 酶切后的载体互补配对。BsmBI 又名 BbsI。

oligoDNA 序列的第一个碱基必须是 G,如选取的 Guide 序列的第一个碱基不是 G,需自行添加一个额外的 G。

2. Oligo 退火形成 duplex

PCR 仪 95℃ 5 min,缓慢降温至室温 1 h,1:200 稀释 duplex。
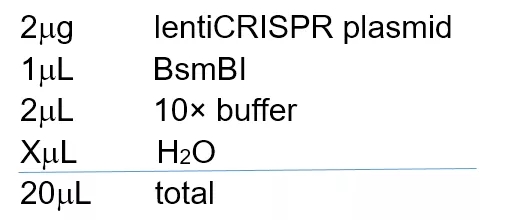
3. 质粒酶切
4. 胶回收
大片段约 11kb 回收;小片段约 2kb 为切下来的 filler,不要。
5. 连接

室温连接 4 - 6 h。
6. 转化(Amp+),挑克隆,摇菌,抽提质粒,测序!
7. 转染
24 孔板,细胞不要过满,同实验室日常转染操作,可选择性设置三个平行。同时转染空载作为阴性对照。每孔 500 ng。
8. puromycin 筛选
转染稳定 48 - 72 h 后加 puromycin 进行筛选,1~3g/mL,直至不再有细胞数量稳定,不再有细胞因不耐药死去。
9. 单细胞分离
由于篇幅原因,有限稀释法具体细节会在后面的文章单独讲解,请关注科研小助手后续文章。100 个细胞铺在一个大盘或 96 孔板中。剩余的细胞冻存。
10. 获得单克隆细胞株
当肉眼可见类似于菌落的细胞团时,将其从大盘或 96 孔板消化下来转移至 12 或 24 孔板中继续扩大培养。注意不要污染到其它细胞团,不要选取过密的细胞团。
11. 扩大培养,提取细胞全基因组 DNA 和细胞总蛋白
12. 测序
Guide 序列上游 100bp 左右和下游 200bp 左右为上下游设计一对引物,以全基因组为模板,以上述引物进行 PCR。PCR 产物送测序,或将产物用 T7E1 进行酶切消化检测突变。
13. Western
与阴性对照相比,靶基因应完全敲除