华人学者开发新型单碱基编辑系统,实现所有碱基的自由转换和增删!
近日,单碱基编辑器的开发者,Broad 研究所 David R. Liu 教授在 nature 杂志发表题为《Search-and-replace genome editing without double-strand breaks or donor DNA》的论文 [1]。
该研究开发出一种新型基因编辑工具——Prime Editor,利用该工具研究者对人类细胞进行了超过 175 次基因编辑,包括靶向插入、删除和所有 12 种类型的点突变,且不需要 DNA 双链断裂(DSB)和供体 DNA 作为模板。并成功修复了导致镰状细胞性贫血症和 Tay-Sachs 病的基因突变。
nature 杂志把该技术称为:「超精确的新型 CRISPR 工具」(图 1), science 杂志也评论它是「超越 CRISPR」的重大突破!

图 1 nature 官网对其评价
图片来源:nature
Prime Editors (PEs) 共三种型号,PE1 系统将逆转录酶(reverse transcriptase, RT)(红色)和 Cas9 核酸剪切酶 (蓝色) 融合,在 pegRNA(prime editing extended guide RNA)(绿色)的引导下,将该复合物带到基因组 DNA(黄、紫色)的靶向位置,并直接将 pegRNA 上的遗传信息延伸复制到目标基因组位点。(图 2)
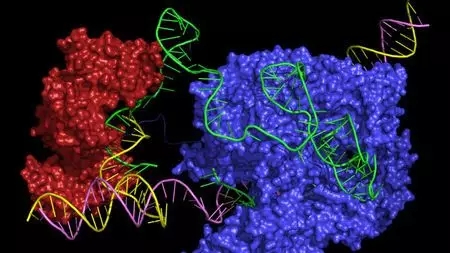
图 2 PE1 系统
图片来源:science
PE2 在 PE1 的基础上,对 M-MLV 逆转录酶氨基酸序列进行修改,增加了热稳定性、加工性以及 DNA:RNA 的底物亲和性,从而提高了编辑效率。
而 PE2 只编辑 DNA 其中一条链,另一条未编辑的链则需进一步靠 DNA 修复来完成编辑。(图 3)
于是,研究者在 pegRNA 下游~50bp 处加上一条 sgRNA,使用 Cas9 剪切酶来切割未编辑的 DNA 链,并以编辑后的 DNA 链为模板,引导 DNA 修复到未编辑的 DNA 链上,建立了 PE3 和 PE3b 系统,进一步对 PE 系统进行了优化。(图 4)
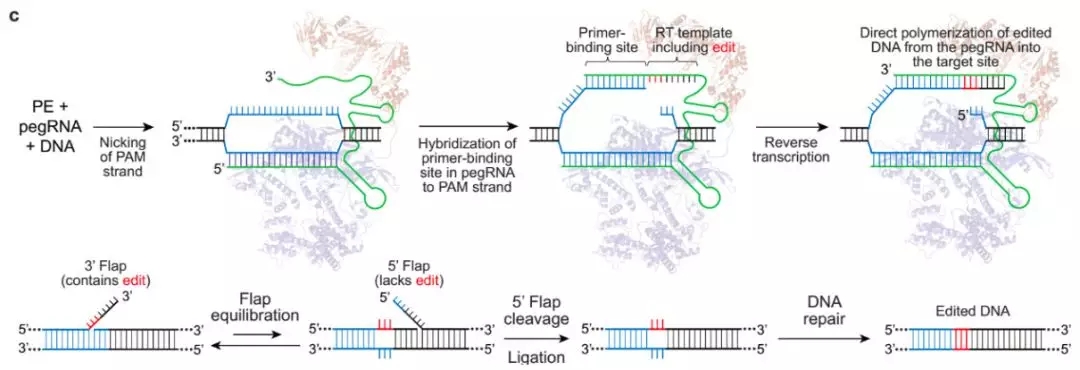
图 3:PE 系统编辑基因原理
图片来源:nature

图 4:PE3 和 PE3 原理
图片来源:nature
如文中所述,Prime Editor 系统相较于同源修复(HDR),提供了效率和产物纯度上的优势;与碱基编辑器(CBE、ABE 见下文)进行了优劣上的互补;在已知的 cas9 脱靶位点上,提供了比 cas9 核酸酶低得多的脱靶效率,原则上可以纠正大约 89% 的已知致病性人类基因变异。
总而言之,Prime Editor 提供了一种单碱基替换和小片段删除、插入的新工具,极大地扩展了基因组编辑的范围并在基因治疗领域具有巨大潜力。
回顾单碱基编辑技术发展历程
2016 年 4 月 David R. Liu 实验室在 nature 发文首次报道了利用 dCas9 蛋白与 APOBEC1(胞苷脱氨酶 1)共表达,开发了胞嘧啶碱基编辑工具 (Cytidine base editors, CBEs)[2]。
CBEs 系统的 dCas9 保留了可被 sgRNA 引导定位到 DNA 靶位点的能力,但不会对 DNA 双链进行剪切,共表达的胞苷脱氨酶直接将目标胞苷转化为尿苷,然后利用 DNA 修复机制将尿嘧啶转化为胸腺嘧啶,可在不对 DNA 双链进行切割的条件下完成将 G/C 碱基转换成 A/T 碱基。
因而一度被认为是比传统的 CRISPR-Cas9 技术更为安全和高效的基因编辑工具。(图 5)
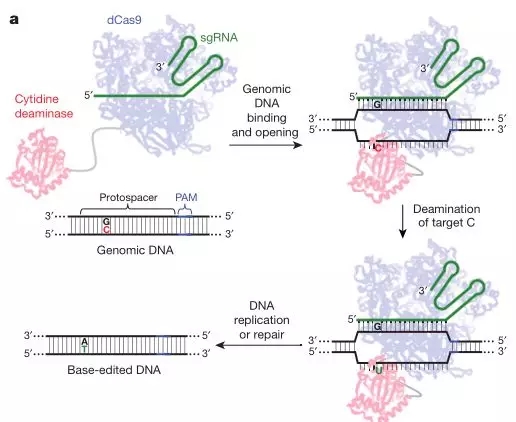
图 5:第一代碱基编辑器(BE1)原理
图片来源:nature
不过,由于第一代 BE 的编辑效率较低,研究者在其基础上融合了嘧啶糖基酶抑制因子 (uracil glycosylase inhibitor, UGI),开发出第二代 BE2。
接着又融合了针对未编辑链的 Cas9 切口酶,以促进 DNA 的修复反应,形成第三代 BE3。
2017 年,Komor AC 等 [3] 在 BE3 的基础上融合了噬菌体 Mu 蛋白 Gam,开发出 BE4。实验证明,BE4 的编辑效率较 BE3 提升了 50%。(图 6)[4]

图 6 :DNA 碱基编辑器及其编辑窗口
图片来源:NATURE REVIEWS GENETICS
随后,在 2017 年,David R. Liu 实验室又一次突破性的开发出了腺嘌呤碱基编辑器 (Adenine base editors, ABEs) [5],该工具打破了 CBEs 仅能编辑嘧啶碱基的限制, 可以将 A/T 碱基转换为 G/C 碱基。
其原理与 CBEs 略有不同,脱氨酶与 Cas9 剪切酶 (Cas9n) 的 N 末端融合,在 gRNA 引导下,非靶向链中的腺苷脱氨,暴露为单链 DNA,然后将腺嘌呤 A 转化为肌苷 I,肌苷在 DNA 水平被当作 G 进行读码复制,然而细胞内对于肌苷的切除修复并不敏感,
因此,ABEs 能够高效地产生 A/T 到 G/C 的突变,且能维持较高的产物纯度。(图 7)
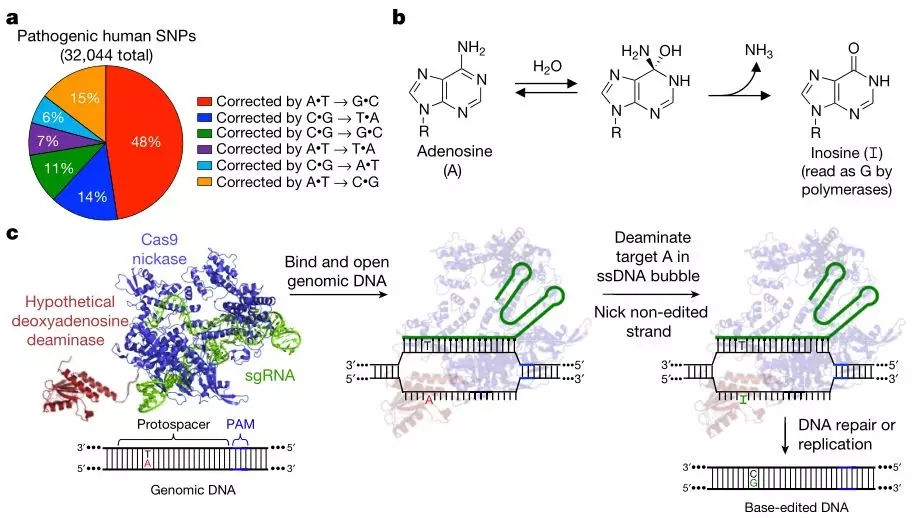
图 7:由 A/T 到 G/C 的碱基编辑范围和原理
图片来源:nature
综上所述,单碱基编辑系统 CBEs 与 ABEs 的开发极大地促进了基因编辑技术的发展,为今后治疗点突变遗传疾病以及生物医学基础研究提供了重要的工具手段。
不过,鉴于以上两者仅能对 C-T, T-C, A-G, G-C 进行转换,对 C-A, C-G, G-C, G-T, A-C, A-T, T-A, T-G 转换无能为力。本次 David R. Liu 团队研发的 Prime Editor 实现了对以上 12 种类型的碱基自由转换,且能对多碱基的小片段实施精准的插入与删除,极大地丰富了单碱基编辑与小片段增删的基因编辑系统。
然而,尽管相较于之前的基因编辑工具而言,Prime Editor 是一次重大的技术突破,但是其最终用于临床点突变遗传疾病的基因治疗还有很长的路要走。
如左二伟研究员所言:「Prime Editing 基因编辑系统与以往基因编辑系统相比避免了 DNA 双链断裂的产生、提高了基因编辑效率,拓展适用范围。然而 Prime editing 组成构件太大也限制了其在体内的临床应用。此外,逆转录酶作为主要构成要件,在细胞中过量表达,其安全性仍然是一个需要考虑的问题。」
参考文献:
[1] Anzalone, A. V. et al. Search-and-replace genome editing without double-strand breaks or donor DNA. Nature, doi:10.1038 /s41586 - 019 - 1711 - 4 (2019).
[2] Komor, A. C., Kim, Y. B., Packer, M. S., Zuris, J. A. & Liu, D. R. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature 533, 420 - 424, doi:10.1038 /nature17946 (2016).
[3] Komor, A. C. et al. Improved base excision repair inhibition and bacteriophage Mu Gam protein yields C_G-to-T_A base editors with higher efficiency and product purity.pdf>. Science Advances (2017).
[4] Rees, H. A. & Liu, D. R. Base editing: precision chemistry on the genome and transcriptome of living cells. Nat Rev Genet 19, 770 - 788, doi:10.1038 /s41576 - 018 - 0059 - 1 (2018).
[5] Gaudelli, N. M. et al. Programmable base editing of undefinedT to undefinedC in genomic DNA without DNA cleavage. Nature 551, 464 - 471, doi:10.1038 /nature24644 (2017).





