有了这款神器,十分钟就能从基因集中获得基因注释信息
当前,高通量测序技术使得我们可以非常轻松就获得海量测序数据,但是如何解释这些数据却并非易事。面对绝大多数的陌生基因,对其进行基因注释显得尤为重要,基因注释信息对我们理解其参与的生物学功能非常重要。
在获得实验组和对照组的差异基因之后,这个时候就需要对这些基因进行注释,在这个过程中,我们需要了解这些差异基因所代表的生物学意义,比如是否富集到代谢、可变剪接通路,是在实验组高表达还是在对照组高表达等等。
目前,可以对基因进行注释的数据库有很多,常用的有生物学过程数据库 GO 和通路数据库 KEGG。
当我们提到基因注释工具时,大多数人第一时间想到的是 DAVID。

实话实说,DAVID 在基因注释方面的功能的确很强大,可信度、公认度也非常高,一切都看似完美,但它有个严重的缺陷,那就是更新速度超级慢,这和日新月异的生物学研究是严重相驳的(最近一次的更新是 2016 年 5 月,而现在 2019 年都快结束了……)
在这里,我们向大家推荐一款神器 -- Metascape。
Metascape 在保留 DAVID 优势的前提下,又完美地弥补了其空缺,下图是 Metascape 最近一次更新的信息,显示是 2018 年 10 月 1 日,并且该网址承诺每月都会有更新,因为他们的宗旨是:「We serve fresh analyses!」

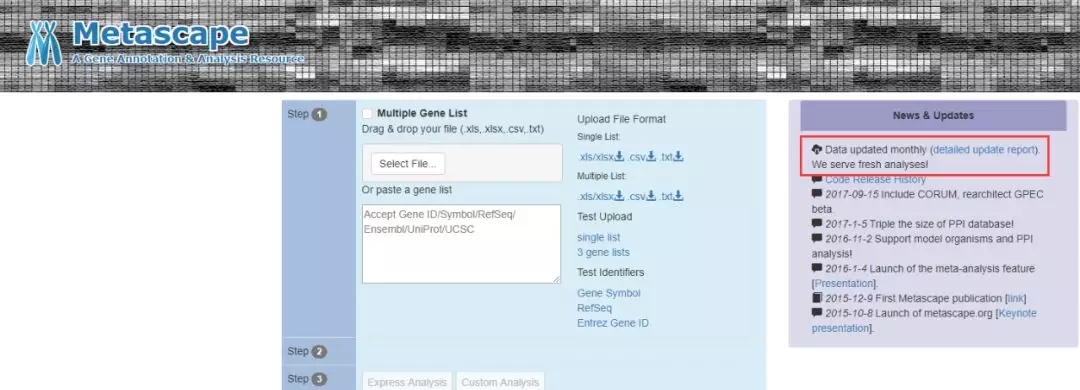
除了高频率的更新速度,Metascape 还是一个近乎「傻瓜式」的基于网页的基因注释工具,几次复制粘贴,然后点击几次「Enter」键,就可以得到基因注释结果了,是不是非常的便捷啊!
接下来,我们就来学习一下如何使用这个工具。
1. 首先,进入 Metascape 官网,我们看到该网站要求的输入文件格式可以是 excel、csv、txt 格式的文件,这基本上符合我们平时保存基因组信息的格式。
除此之外,你也可以直接在下图中的对话框中粘贴自己的基因列表。Metascape 可以识别的基因 ID 包括「Gene Symbol」、「RefSeq」、「Entrez Gene ID」,因此,如果你的基因 ID 不属于这三种,那么在你用该工具的时候,需要先将基因 ID 转换成这三种 ID 之一。
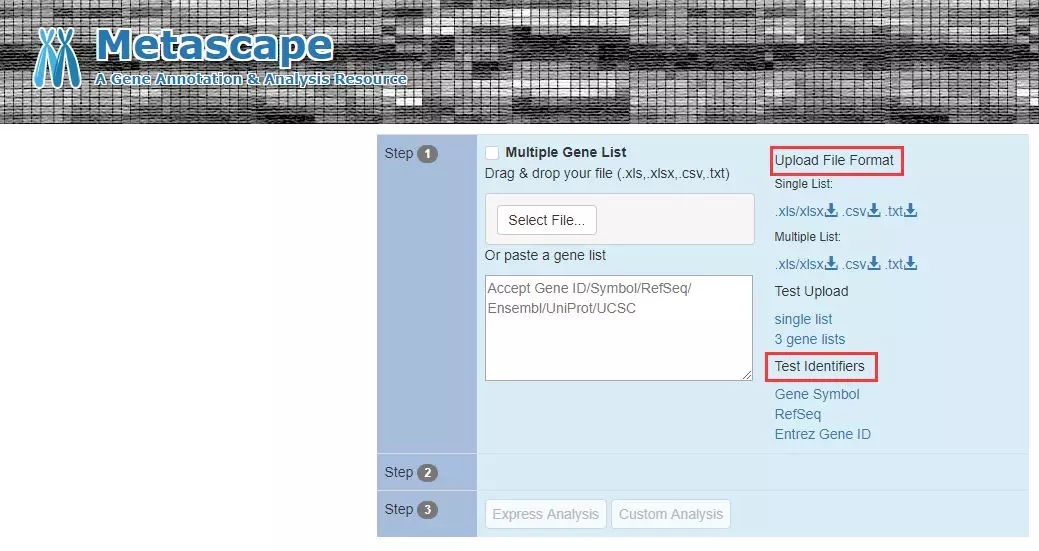
在上图 Test Upload 中有多个 list 及单个 list 上传文件的实例文件,因此大家可以自行下载查看,非常简单。
另外,并不建议同时输入多个基因 list 进行分析,尽管多个基因 list 可能会提高各个 list 之间的关系,但注释结果是非常乱的,不容易解读,当然这可能是个人偏好,如果你就要同时注释多个基因集,那么你需要将上图中「Multiple Gene List」勾选。
在这里,我们通过直接复制、粘贴来完成我们的单个基因 list 的注释,选用的基因集是在 GSEA 官网下载的髓系细胞分化的基因集。
下载链接:
http://software.broadinstitute.org/gsea/msigdb/geneset_page.jsp?geneSetName=GO_MYELOID_CELL_DIFFERENTIATION&keywords=erythrocyte%20development
该基因集包含了 189 个基因。
2. 复制这些基因的名字(Gene Symbol),第一步是将这些基因粘贴的对话框中,点击「Submit」。
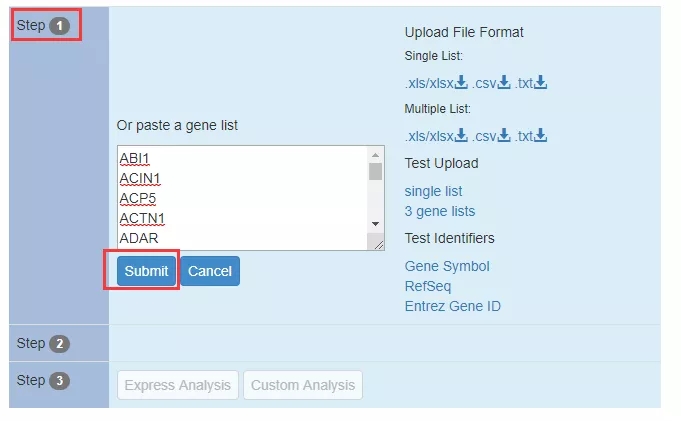
此时,Metascape 会自动识别该输入基因集的物种信息及基因个数。
3. 在进行分析之前,还需要下拉「Any Species」选择自己的输入基因物种,在这里选择「H.sapiens」,选完物种信息之后,点击第三步中的「Express Analysis」。
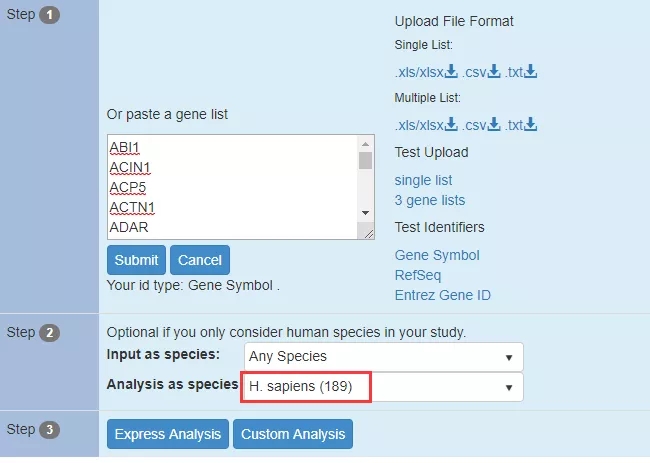
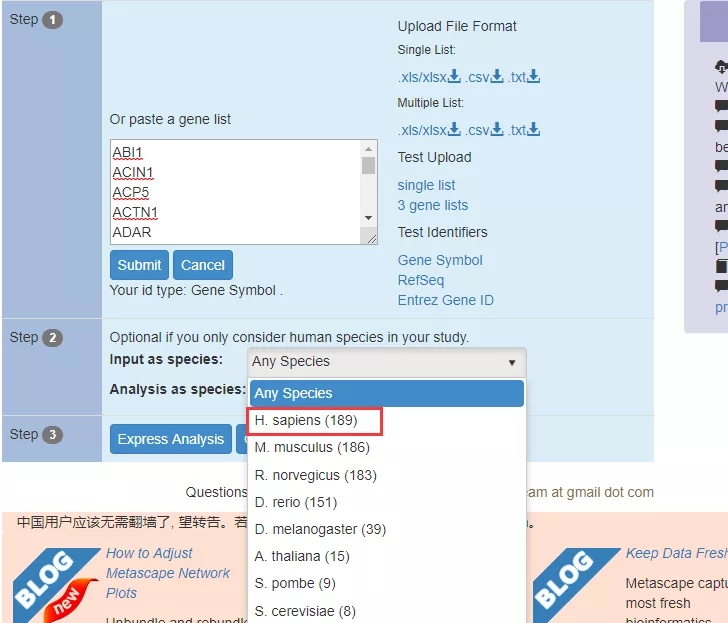
此时,我们会在下方看到分析进度条,等到结束的时候,会看到完成度 100% 的进度条。
4. 点击「Analysis Report Page」,此时会进入另外一个界面。
5. 在新页面的最上方,是分析结果的不同保存形式,你可以下载 Excel 表格,同时 Metascape 还会生成一份 PPT,看上去还是非常高大上的,当然,你也可以选择一股脑将所有注释结果下载,点解「All in One Zip File」,会得到一个压缩文件,解压后会是结果汇总。
除此之外,Metascape 还会提供一个网页版的结果。
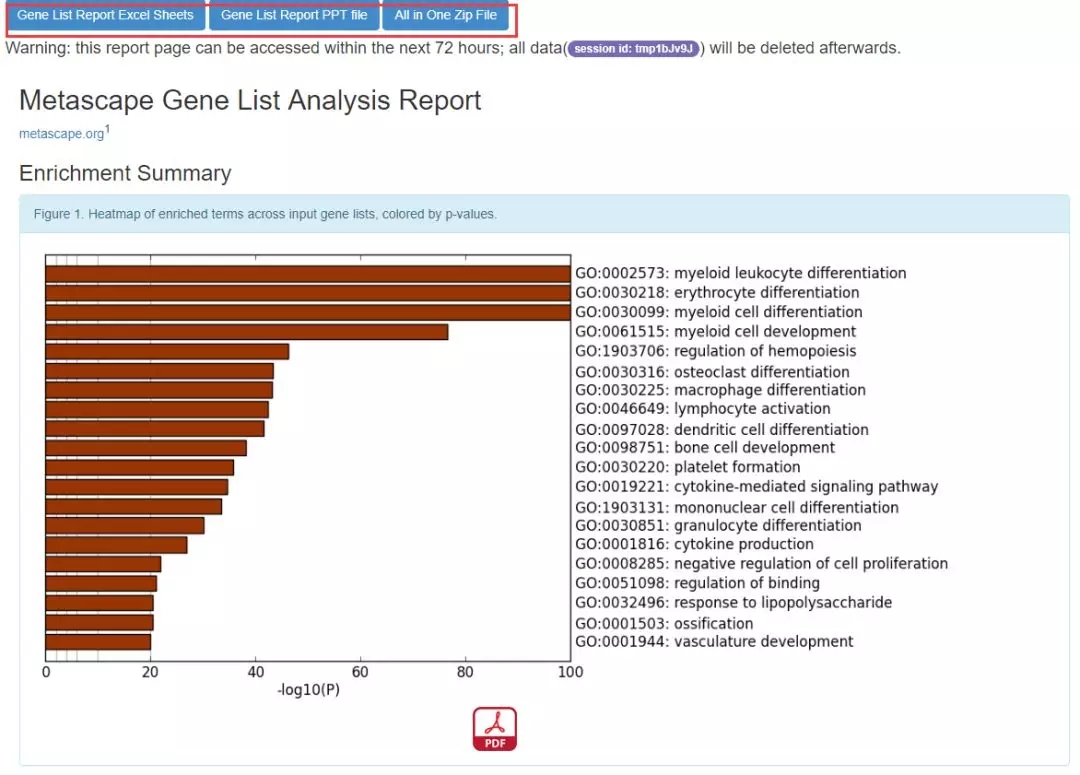
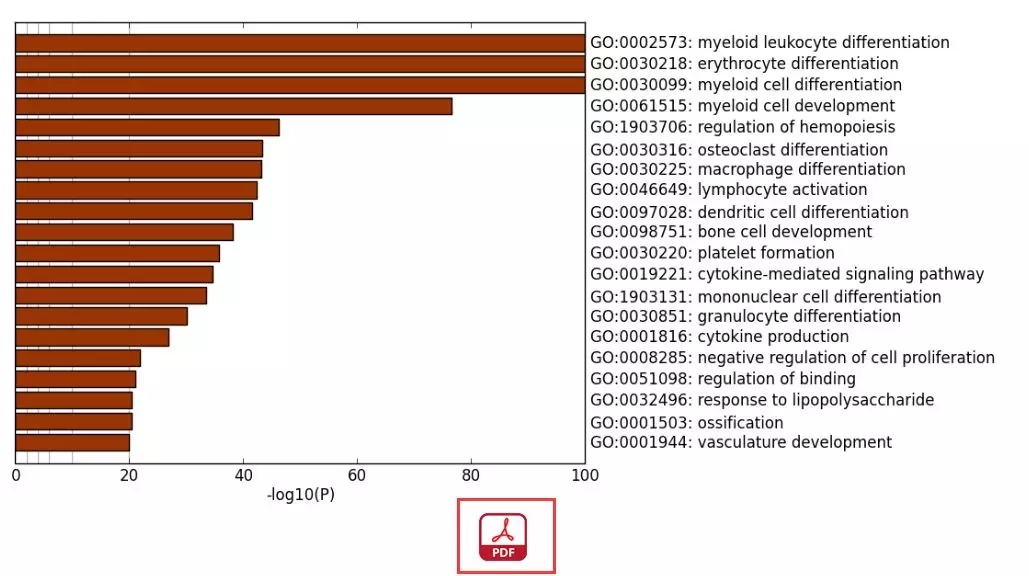
6. 下图中的「Enrichment Summary」中是输入基因集的注释信息汇总,颜色深浅代表每条 GO 的 P 值,在注释结果中,我们可以看到髓系细胞分化发育、红细胞分化、造血调控等信息,这与我们输入的基因集是相一致的。
如果想下载这个图,点击下方的「PDF」即可。
7. 向下滚动页面,会有生物学过程和通路的详细信息,其中 Count 和 % 为输入基因集中富集到该条 GO 的数量及比例,之后的 Log10 (P) 、Log10 (q) 为该条 GO 的统计学意义,数值越小代表越特异。
做到这儿,其实就能够达到基因注释的基本目的。但是, Metascape 提供的要远远超过这些。
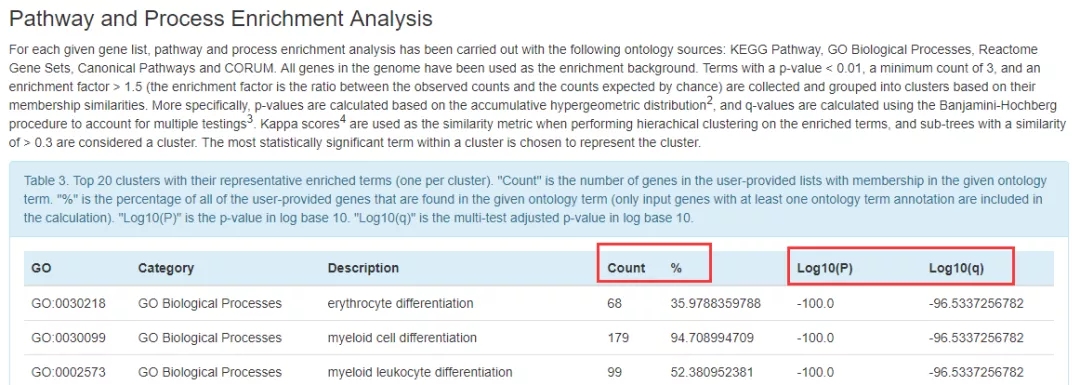
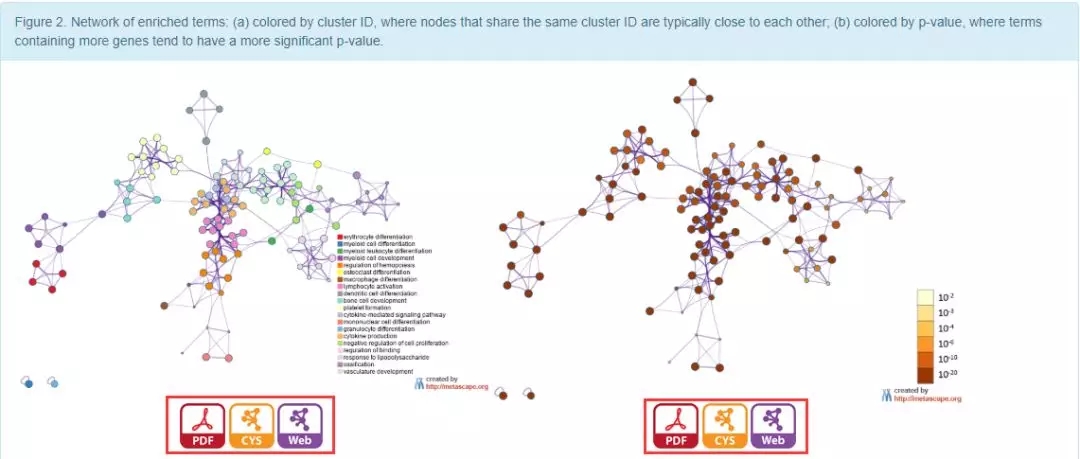
8. 继续向下滚动页面,在第二张图中,做出的是图一富集的各个生物过程及通路的相关关系,左边是以每个生物学过程定义颜色,显示的是各个通路之间的相互关系;右侧是以 P 值定义颜色,揭示的是各个通路之间联系的特异性。
该图不仅仅可以下载为 PDF 版本,同时也可下载为.cys 格式,以方便在 Cytoscape 中进行调整和完善。
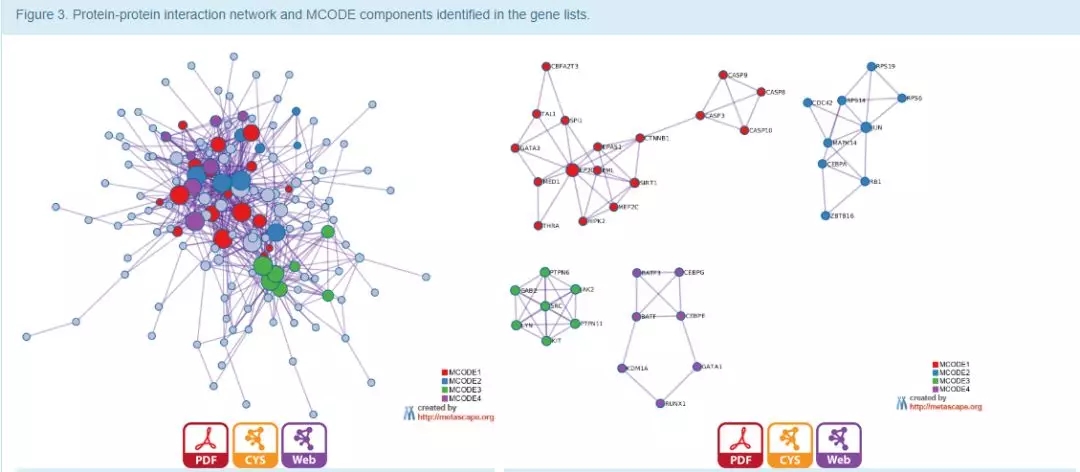
9. 除此之外,Metascape 还会对输入基因集进行蛋白互作富集分析,该分析基于的数据库有 BioGrid, InWeb_IM 和 OmniPath8。
蛋白子集之间会彼此之间相连以构成互作网络,当该网络中的蛋白数量为 3~500 之间时,Molecular Complex Detection (MCODE) 算法会对这些蛋白进行互作密度检测,并将结果展示在下图中。
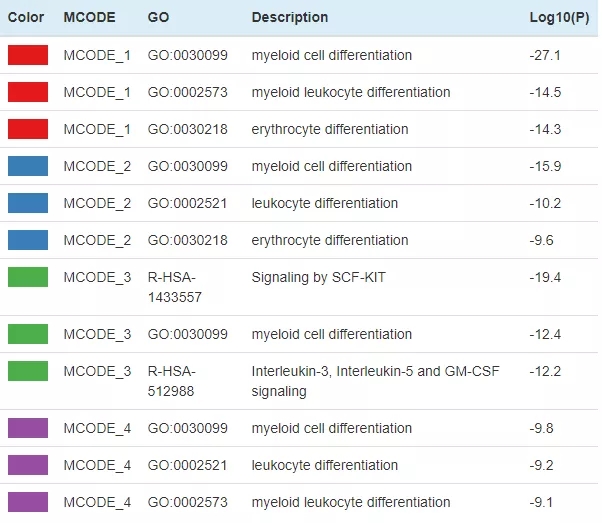
下图是对上图蛋白互作网络中颜色的解释,上图中,左边是各个模块之间的相互关系,右边是每个模块内包含的基因。下图是每个模块包含的 GO,以及利用其进行互作分析时得出的 P 值。
10. 至此,我们就完成了对目标基因集的注释。
从拿到基因集到看到基因集注释结果,整个过程绝对不会超过十分钟,可以说非常快了,另外,Metascape 是基于网页的基因注释分析,所以说非常的方便、易懂。
最后,恭喜你又获得了一项技能!