基因组注释不用愁,这款在线神器送给你!
当我们得到一株菌株的全基因组序列后首先要做什么?基因组注释!那有什么方便又好用的注释神器吗?
下面为大家介绍一款超好用的在线基因组注释神器 ——RAST,让大家随时随地都可以自己进行注释分析。
RAST 网址为 rast.nmpdr.org,是基于 Subsystem 技术的一个针对细菌和古菌基因组的快速注释工具。它可以用来预测 ORF、rRNA、tRNA 以及相应的功能基因,并可以利用这些信息构建代谢网络。
首先我们通过邮箱注册一个账号,登陆,开始一个新的项目。点击 Your Jobs 后再点击 Upload New Job,就可以上传序列了。
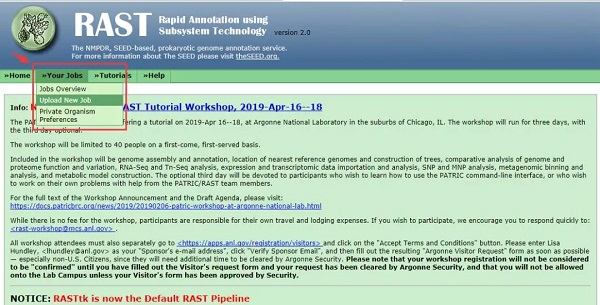
可以从 NCBI 上下载你所需要的(未进行注释的)基因组数据(FASTA 或者 GenBank 格式)。上传后,进入下一步。
输入分类 ID
搜索网址:
www.ncbi.nlm.nih.gov/Taxonomy/taxonomyhome.html
输入了正确的 ID 之后,下面的信息会自己显示出来。如果不知道该菌株的分类或者找不到该菌株的 ID,可以自己来手动填写下面的分类。此处 Genetic code 大多数的细菌都选择 11。
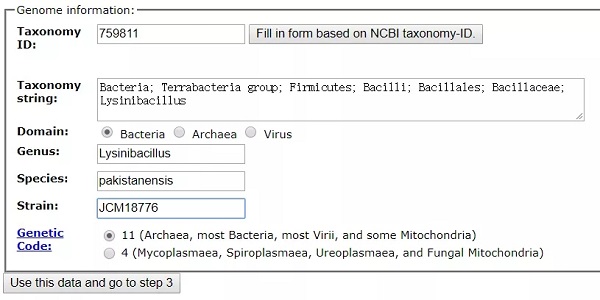
这一步的选择可以根据自己的需要进行设置。设置完成后点击 Finish the upload。

注释完成后将给你的邮箱发送通知。结果可以通过 Your Jobs 中的 Jobs Overview 查看。在 Annotation Progress 中显示满格绿色时代表注释已经完成,点击 View details 查看详细结果。

结果页面如下,可以查看和下载注释结果,分享注释结果,也可以查看相近的菌株。
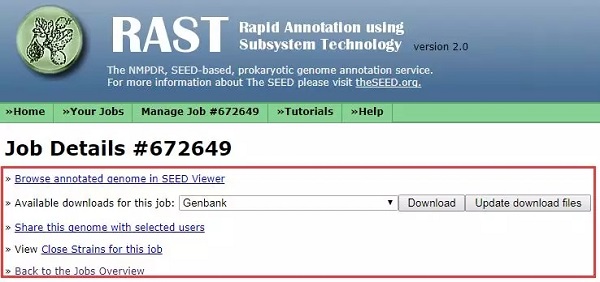
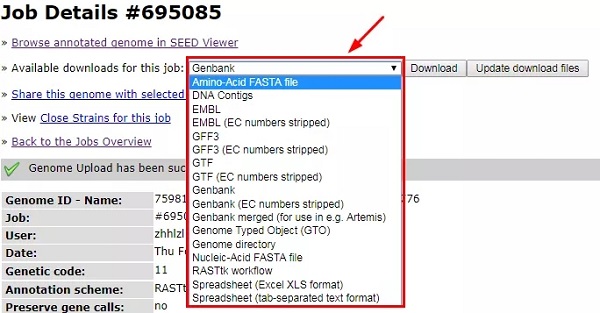
下载注释结果时,RAST 提供了多种格式供你选择,方便你之后的分析。
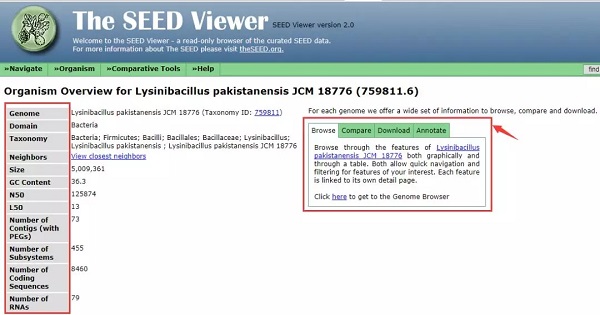
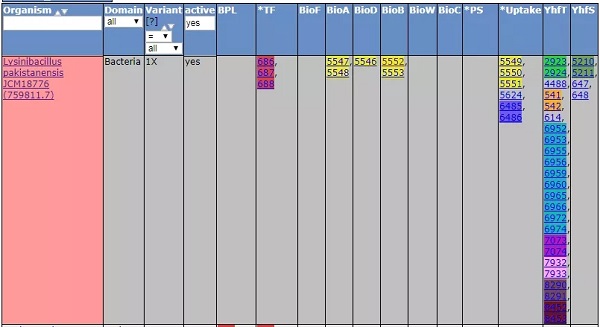
查看注释结果,这一页显示了上传菌株的基本信息,包括基因组大小、GC 含量、N50 和 L50(这两个数值主要用来评估序列组装的质量)、contigs 数、Subsystems 数、CDS 数和 RNA 数。右边的框里可以查看一些其他信息,一会儿进行详细介绍。
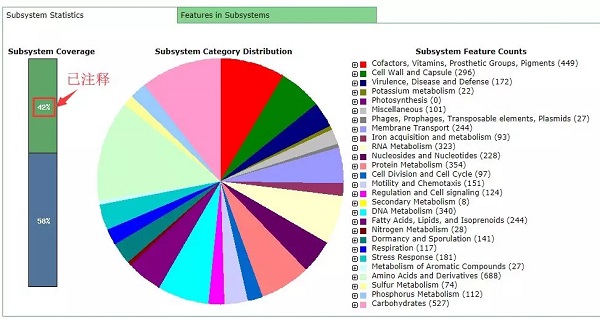
下面的饼图显示被注释上的基因占全基因组的比例以及各个功能所占的比例。右边的 Subsystem Feature Counts 中的信息都可以展开进行查看。
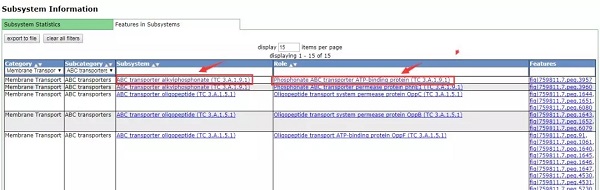
除了饼图分布之外右边的「Features in Subsystems」列举了详细信息,可以进行查找浏览。点击 Subsystem 和 Role 下面的选项可以看到很多该功能的详细信息。
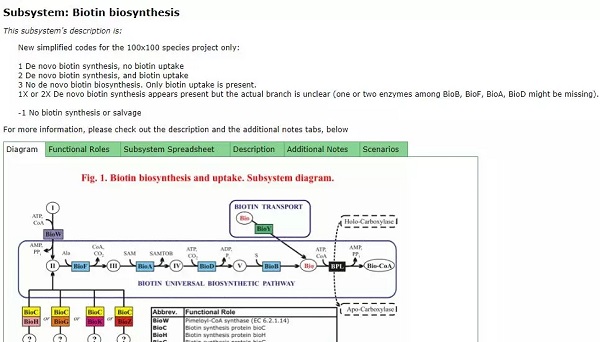
以及关于基因和基因簇的总体展示,相同颜色的代表基因处于同一个基因簇内。
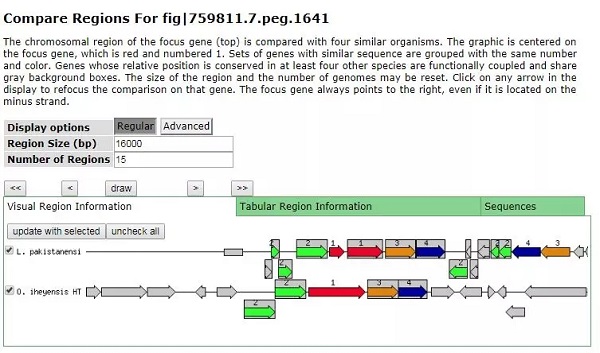
点进去还可以查看基因的序列及详细信息和上下游区域的比较(可以调整窗口覆盖的范围)。
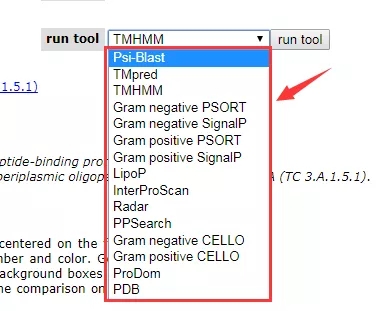
还有非常好用的地方就是从 RAST 查看基因的界面可以直接链接到别的网站做分析,包括分析跨膜区(TMHMM、TMpred)、比对蛋白序列数据库(Psi-Blast、InterProScan)及结构数据库(PDB)、亚细胞定位(PSORT、CELLO)、寻找信号肽(SignalP)、脂蛋白鉴定(LipoP)、结构域分析(ProDom)等。以运行 Psi-Blast 为例,直接「run tool」就会直接进入 NCBI 的 Blast 页面。
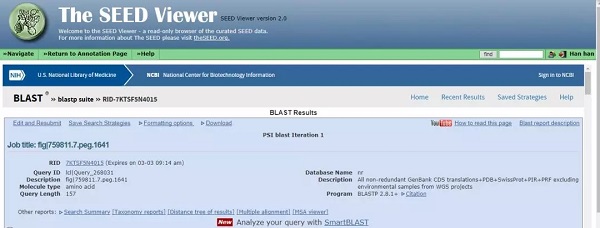
Blast 结果页面。
返回初页面选择「Genome Browser」,可以自主选择展示基因组的特定区域进行可视化。
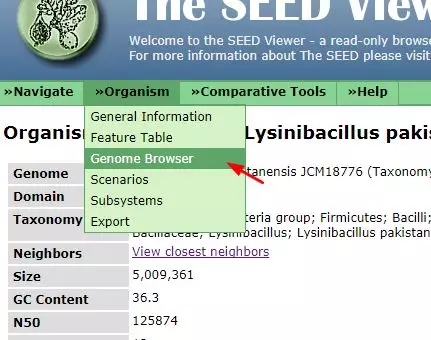

RAST 除了能注释出功能基因并做一个简单的代谢网络以外,还可以用进行基因组间基于功能(function)、序列(sequence)或者是 KEGG 代谢通路的比较。

基于功能(function based)的比较(一对一):
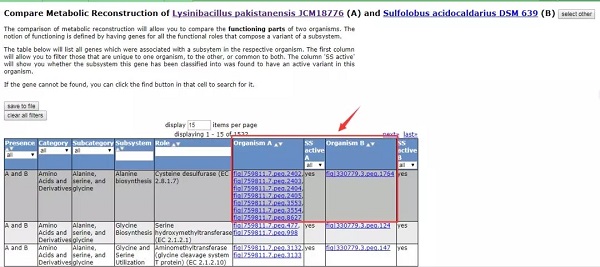
基于序列(sequence based)的比较(一对一)。结果会出现一个圈图。展现了两个菌株的相关性,圆圈附近有个小红点,移动它可以看不同区域的基因展示情况,左边的表格会随着圆点的移动改变。
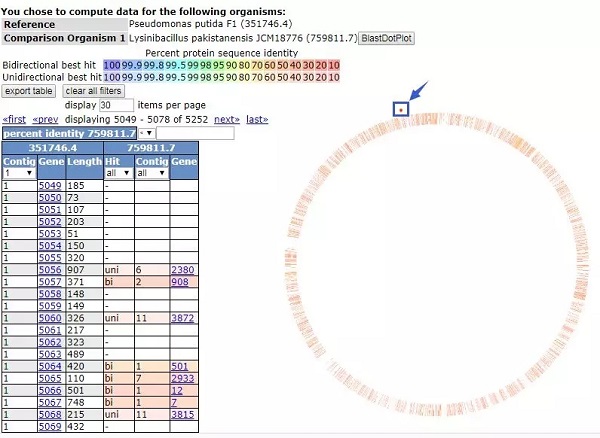
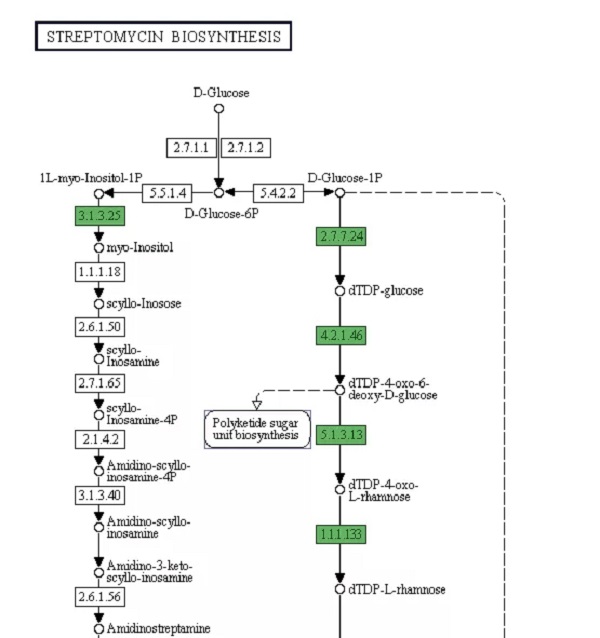
还有就是 KEGG 代谢通路注释及比较,选定一个代谢通路,再选定菌株。结果会把这条代谢通路中参与的酶都标注出来。绿色的方框就表示该菌株存在这个酶,点开它就会跳转到基因详细信息的页面。
以上就是简单介绍了 RAST 的使用方法以及结果页面,RAST 是一个非常方便的在线注释工具,使用界面友好简洁,功能十分强大,年均引用量已突破 400,大家有兴趣地可以继续摸索和研究。希望这个工具可以帮助大家更加了解自己的菌株!(封面来源:站酷海洛 Plus)
参考文献:
1.Aziz, R. K., Bartels, D., Best, A. A., Dejongh, M., Disz, T., & Edwards, R. A., et al. (2008). The rast server: rapid annotations using subsystems technology. Bmc Genomics, 9(1), 75-75.
2.Overbeek, R., Olson, R., Pusch, G. D., Olsen, G. J., Davis, J. J., & Disz, T., et al. (2014). The seed and the rapid annotation of microbial genomes using subsystems technology (rast). Nucleic Acids Research,42(Database issue), D206.
3.Brettin, T., Davis, J. J., Disz, T., Edwards, R. A., Gerdes, S., & Olsen, G. J., et al. (2015). Rasttk: a modular and extensible implementation of the rast algorithm for building custom annotation pipelines and annotating batches of genomes. Sci Rep, 5(8365), 8365.
图片来源:作者提供