掌握这些基因组 MGE 识别数据库,分析工作不用愁!
丁香园
移动遗传元件 (Mobile genetic elements, MGEs) 如质粒、噬菌体、基因组岛、转座子和插入序列等是能够在细胞内外移动、携带编码特定功能基因的基因组片段,在生物体基因转移和基因重组方面以及物种进化方面起着不可小觑的推动和调节作用,并且一直是研究的热点。
MGE 也是横向基因转移事件的指示物,代表着生物体对不同环境的主要适应性机制。
如何准确预测基因组中的 MGE 并进行分析一直是大家关注的问题,今天就为大家介绍一些可以识别 MGE 的数据库:
IslandViewer4
ISFinder
PHASTER
ACLAME
CENSOR、RepeatMasker 与 HTT-DB
熟练使用后将方便大家之后的分析工作。
一、使用 IslandViewer4 识别基因组岛
基因组岛是细菌或古细菌基因组中位置相邻的外源基因集群,通常与 tRNA 基因整合在一起,通常编码病原体的毒力因子如抗菌素耐药性基因,编码与环境适应性相关的新基因如金属抗性基因,以及毒物代谢基因如芳烃降解基因等。
IslandViewer4 服务器集成了利用序列组分预测的 IslandPath-DIMOB 算法和 SIGI-HMM 算法,以及利用比较基因岛的 IslandPick 算法,可以识别基因组内潜在的基因组岛。
登录 IslandViewer4 主页,可以直接查看和下载所有已发表的已预测的细菌和古细菌基因组序列的基因组岛,或者上传自己未发表的基因组进行分析。
具体分析步骤:
1、点击 GENOME UPLOAD -> 选择文件 -> 输入任务名 -> Upload(提交任务)。

2、选择 IslandViewer4 数据库里已有的与上传基因组亲缘关系较近的物种以方便分析,选好后点击 Start Analysis。

IslandViewer4 就会按照预定流程进行分析。

3、任务完成后会显示如下界面,其中圈图展示基因组上预测出的基因岛的分布、位置、不同颜色表示对应的分析方法,圈图的内圈是基因组 G+C 含量的变化,一般基因岛的 G+C 含量会显著偏离基因组 G+C 含量的平均水平。
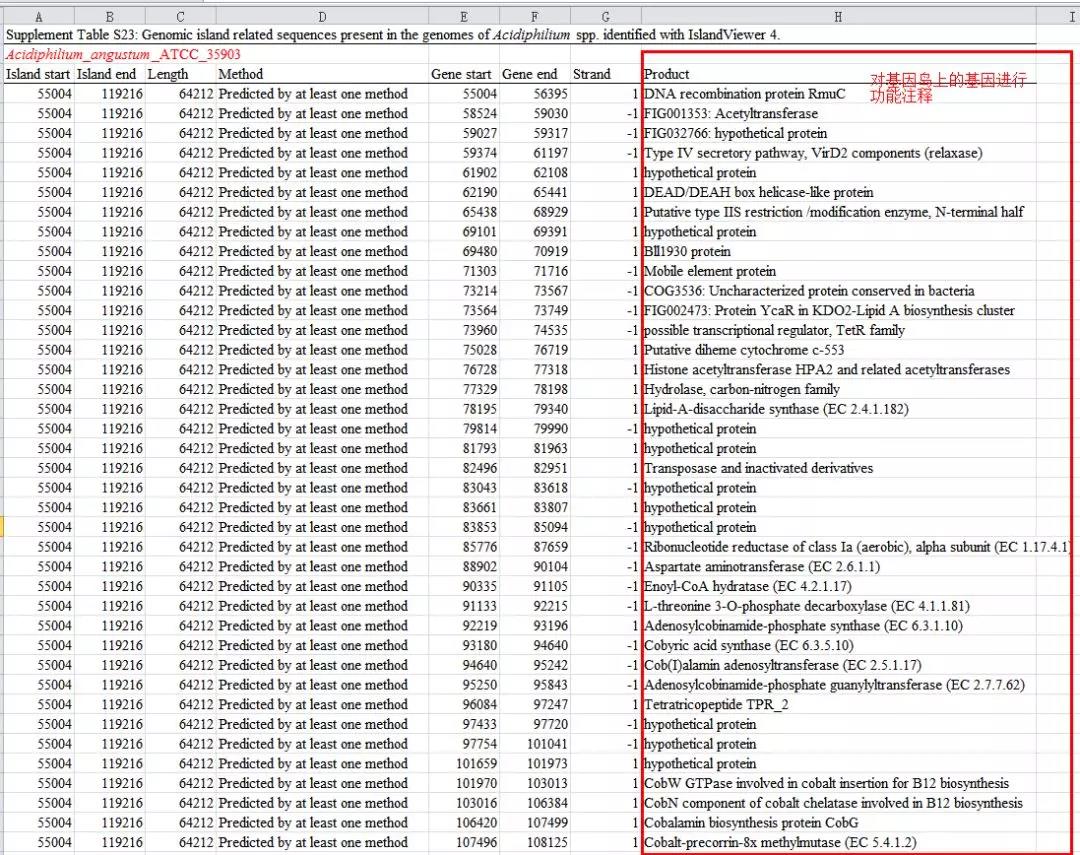
下面是基因组岛区域的详细展示,条形基因组可以用鼠标滑轮缩放查看,下面的表格具体展示基因岛的数量、范围、起始位置、预测方法等,点击进去还会显示基因岛上基因的具体功能。
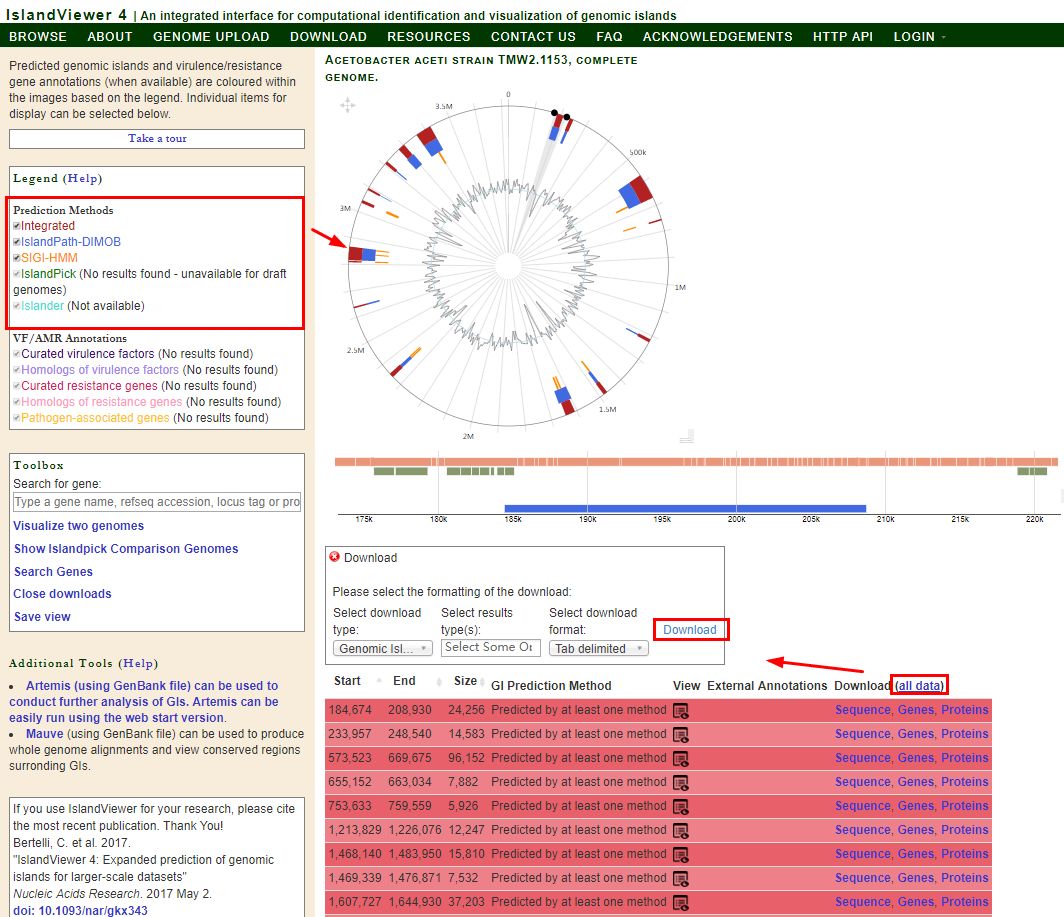
也可以选择下载整个表格,用 Excel 打开并整理成三线表的格式即可以作为文章的支撑材料。
二、使用 ISFinder 预测和归类基因组上的插入序列
插入序列是相对较短的紧凑的 DNA 片段(0.7 到 2.5 kb 之间),它们只编码与移动性有关的功能,是 MGE 的一大类群。
许多插入序列末端携带短的(< 40bp)不完全反向重复序列,并在插入点的两侧通常存在一个 2 - 14bp 的短 DNA 复制。ISFinder 服务器可以帮助我们鉴别基因组内的插入序列。
具体操作步骤:
1、打开 ISFinder 主页,点击 Tool -> Blast 选项卡。

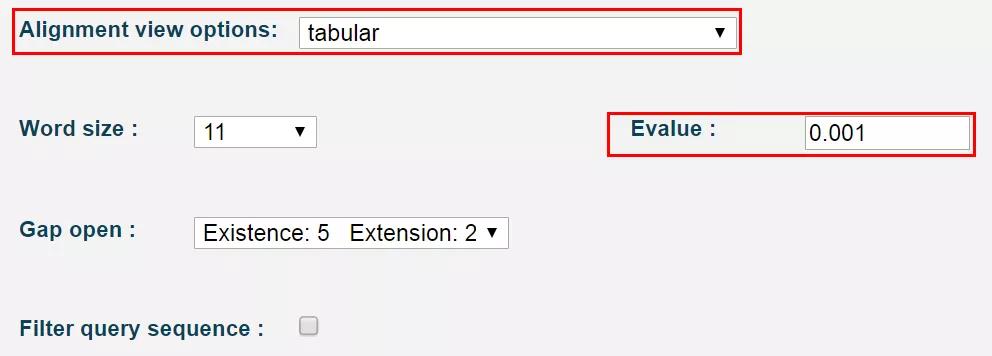
2、可以选择上传基因组核酸序列或蛋白质序列,并选择对应的 Blast 算法。
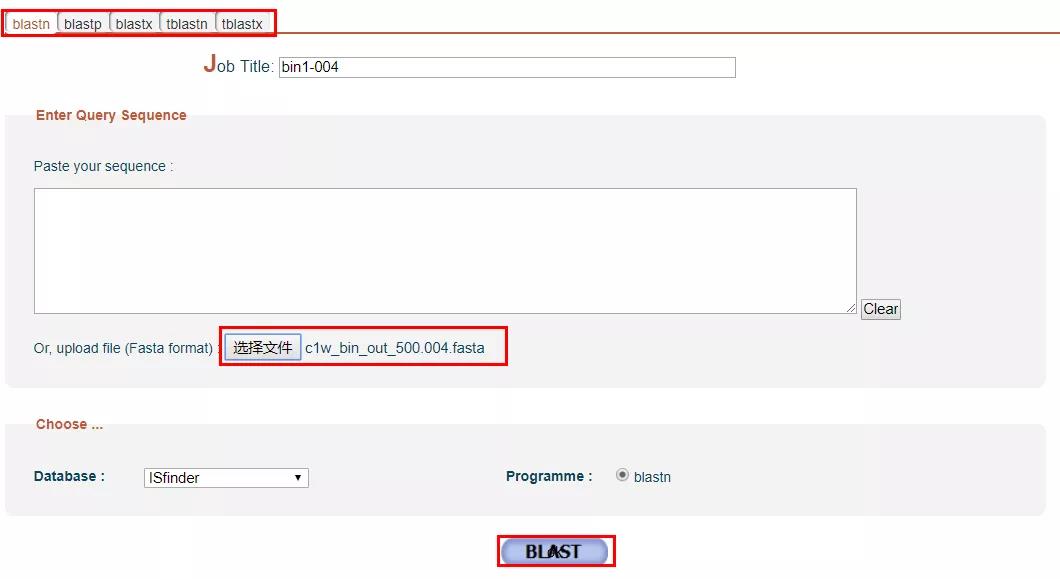
3、参数列表里,注意一下输出结果形式以及比对 E 值的选择。推荐输出表格形式的结果。
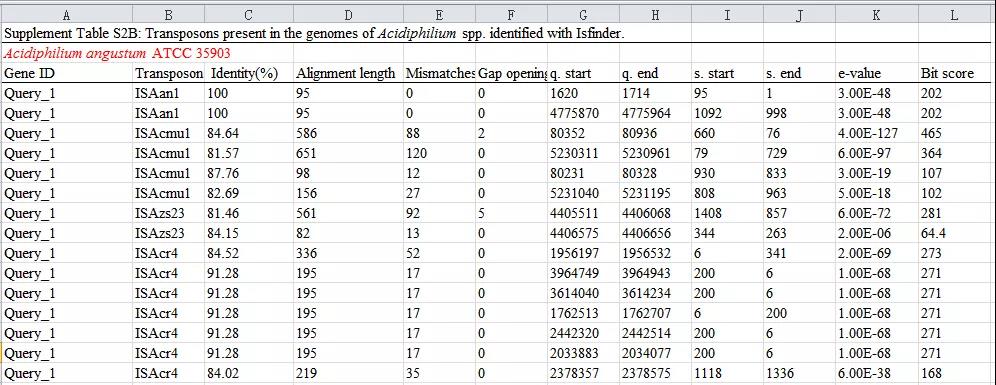
4、运行完成后整理结果到 Excel 表格里,并进行统计
三、使用 PHASTER 鉴定和注释噬菌体序列
一部分微生物的横向转移基因是通过噬菌体的侵染而获得的。如果细菌中存在连续的并且完整的噬菌体蛋白包括溶菌酶、蛋白酶、连接位点、外壳蛋白、整合酶、末端酶、通道蛋白,说明是一个完整的前噬菌体。
PHASTER (Phage Search Tool - Enhanced Release) 通过与噬菌体数据库进行比对可以快速鉴定和注释微生物基因组内的噬菌体序列。
具体操作步骤:
1、登录 PHASTER 的主页,根据实际情况选择上传文件以及参数,提交任务。

2、结果界面首页显示完整或不完整的噬菌体序列数目、位置等信息。

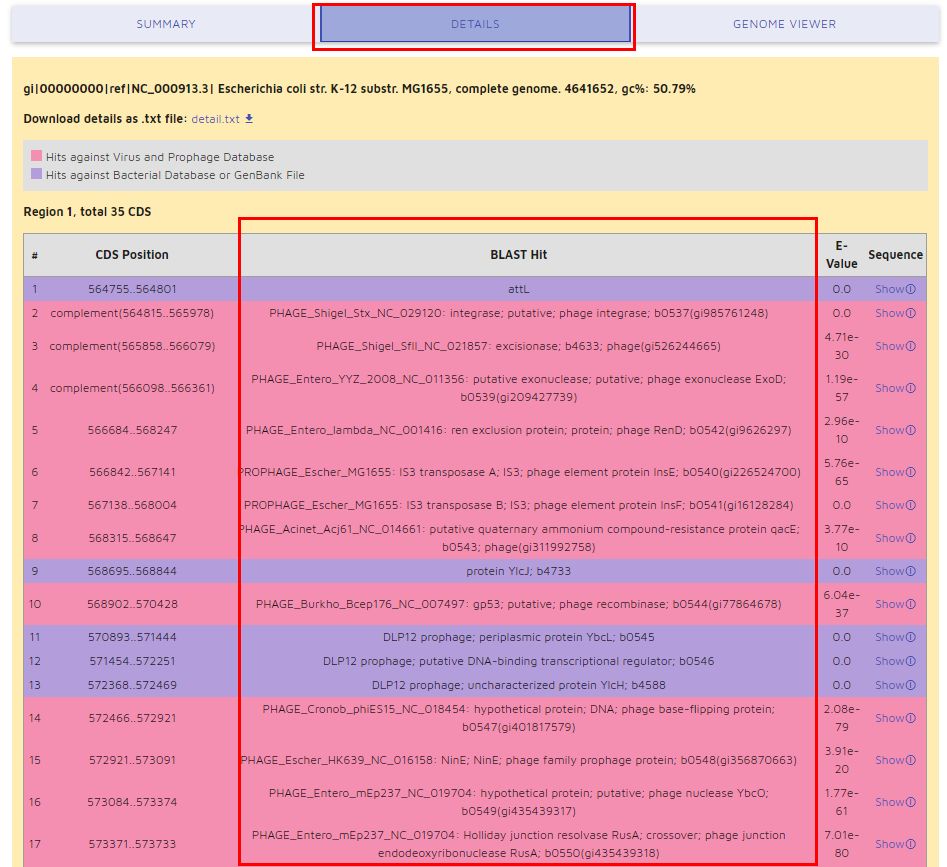
3、详细结果界面(Details)显示噬菌体序列上基因编码的功能。
4、GENOME VIEWER 选项卡可以直观查看噬菌体序列在微生物基因组上的数量及位置,点击圈图上相应的噬菌体,会显示更详细的区域展示,其中不同功能的基因以不同颜色标示。所有结果都可以下载。

四、综合数据库 ACLAME
ACLAME 是一个致力于收集和归类来自不同来源的 MGE 的综合数据库,收集了近 33 万条 MGE 序列,主要包括已知的噬菌体基因、质粒和其他病毒。

可以进行在线分析,或者下载对应数据库进行本地化分析。

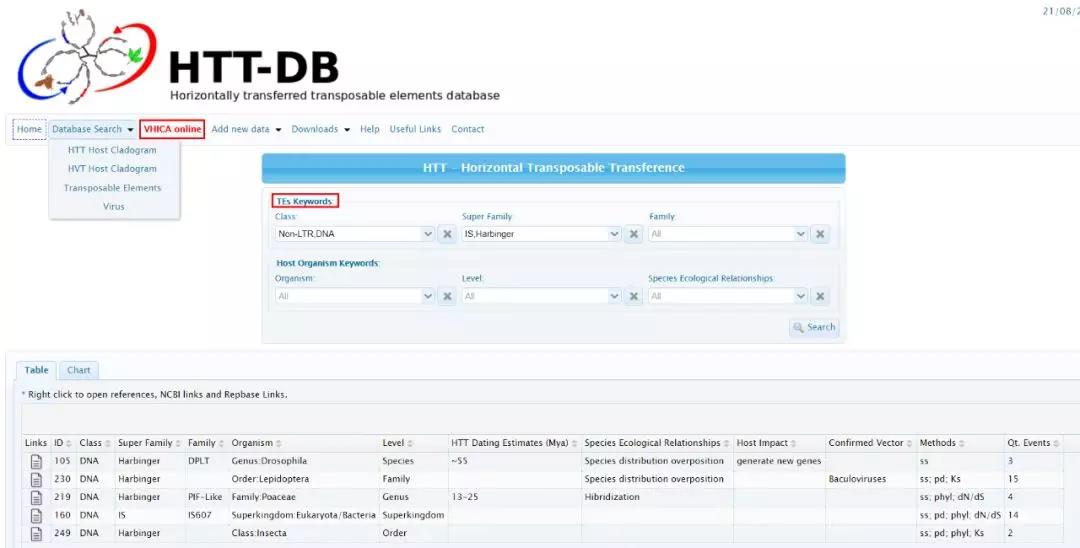
五、CENSOR、RepeatMasker 与 HTT-DB:检测重复序列与转座子
基因组的很大一部分是由多个重复 DNA 拷贝组成的,可以分为两大类:
第一类是主要由宿主复制机制或重组过程生成的串联重复序列;
第二类包括由逆转录因子和 DNA 转座子衍生的零星重复,统称为转座子或转座子元件。
它们能够通过非同源重组从基因组上的某个位置移动到另一个位置的「移动基因」,可分为通过 RNA 或 DNA 中间体进行转移的转座子,但是这种移运是由转座子自己介导的,并不会对物种的适应性生存做出贡献,所以它们又被称为自私基因。
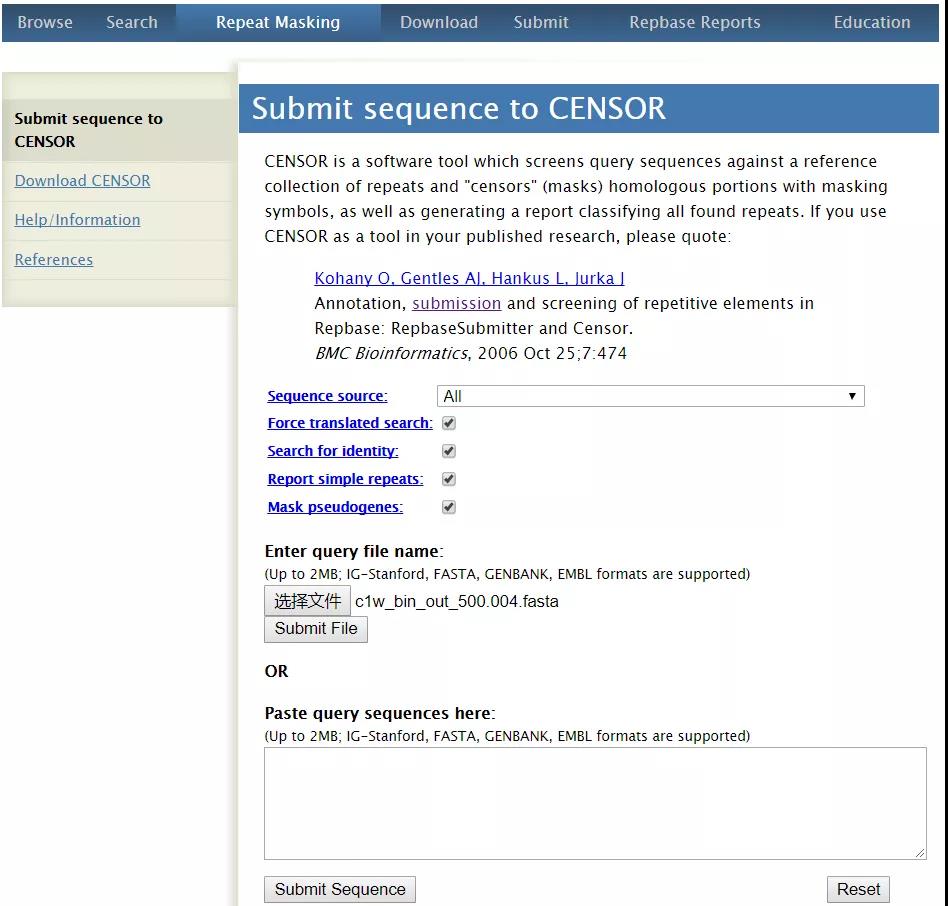
CENSOR 通过比对数据库 Repbase 识别标注基因组重复序列,还可以进行简单重复的从头检测(denovo detection,包括由几个序列基序组成的简单重复)。
RepeatMasker 可以通过比对 Repbase 及 Dfam 数据库检测 DNA 序列中的间隔重复序列和低复杂度序列。

HTT 数据库可以根据关键词检索不同物种转座子横向转移事件,而垂直和水平遗传一致性分析 (VHICA) 可以检测上传的目标基因组内的转座子横向转移事件。