Sci Adv: 打开重编程「黑箱」,这个团队发现关键因子,提高多能干细胞诱导效率
2012 年,山中伸弥因诱导多功能干细胞(iPSCs)获得了当年的诺贝尔医学奖。之后,科学界在 iPSCs 领域投入了大量的资源以兑现其巨大的临床价值。2017 年,日本理研所的高桥雅代在 NEJM 上报道了使用 iPSC 疗法治疗视网膜病变的人体试验结果。两年后,美国国立眼科研究所也进行了自体 iPSC 治疗视网膜病变的临床试验。
截止至 2020 年 5 月,全世界有 53 个正在进行的 iPSC 相关的临床研究。iPSC 离我们的生活已经不再遥远。可尽管如此,人类自体细胞的重编程效率依旧不高,这给 iPSC 的应用带来的极大的困难。如何进一步提高人体细胞的重编程效率?是所有 iPSC 学者都想解决的问题之一。
尽管我们早已知道操纵「山中四因子」能实现细胞的重编程,我们对这个过程中发现的具体变化依旧不甚了解。重编程的过程更像是一个「黑箱」,我们只知其然,却不知其所以然。破译这个「黑箱」,对于进一步解决重编程效率以及安全性等问题解决至关重要。
随着高通量测序技术的兴起以及单细胞技术的日趋成熟,科学家应对复杂系统的分析也越来越多。但遗憾的是,由于重编程过程极度复杂,我们依旧还未完全打开重编程的「黑箱」。
2020 年 9 月 11 号,来自新加坡分子细胞生物学研究院的邢巧瑞博士 (Dr Qiaorui Xing) 与导师 Jonathan Loh Yuin-Han 研究员带领团队在 Science 子刊 Science Advances 发表了题为 Diversification of reprogramming trajectories revealed by parallel single-cell transcriptome and chromatin accessibility sequencing 的论文 [1],报道了他们如何利用单细胞 RNA 测序技术(scRNA-seq)与单细胞可接近性染色质测序技术 (scATAC-seq) 的结合打开重编程的「黑箱」,发现提高诱导多能干细胞效率的方法。在这项研究中,作者发现细胞被重编程为干细胞的潜力在重编程的早期就已出现。人体细胞在重编程过程中存在极高的异质性,最后往往只有少数细胞能成为 iPSC,而单细胞层面的分析正是研究细胞异质性的法宝。
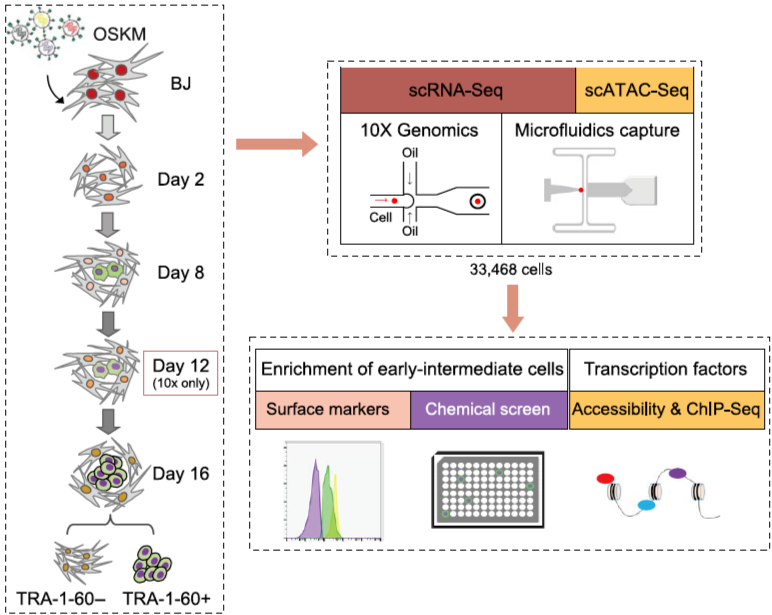
所以,作者使用了 OSKM 经典方法对人源成纤维细胞进行了重编程诱导,再通过基于微流控芯片系统的分离技术,对 33468 个细胞进行了两种单细胞测序,并建立了测序文库。通过该方法获得的测序数据表明,重编程失败的细胞依旧表达高水平的间充质细胞与体细胞标记物 ZEB1 和 COL1A1,而重编程成功的细胞显示出了高水平的 EPCAM ,NANOG 与 LIN28A 表达。这些数据证明通过微流控芯片分离出单细胞能够得到高质量的测序结果。在获取到高质量的数据后,作者进行了参考成分分析(Reference Component Analysis,RCA) 和基因表达差异分析(DGE analysis),发现细胞在重编程过程中表现出了极其丰富的多样性。在几个关键的时间点,如 2 天(D2),8 天 (D8) 以及 16 天 (D16),作者发现细胞中都出现特性截然不同的多个细胞亚群。以 D8 为例,细胞中就出现了 G1、G2 和 G3 三种各种基因表达相去甚远的细胞类型。
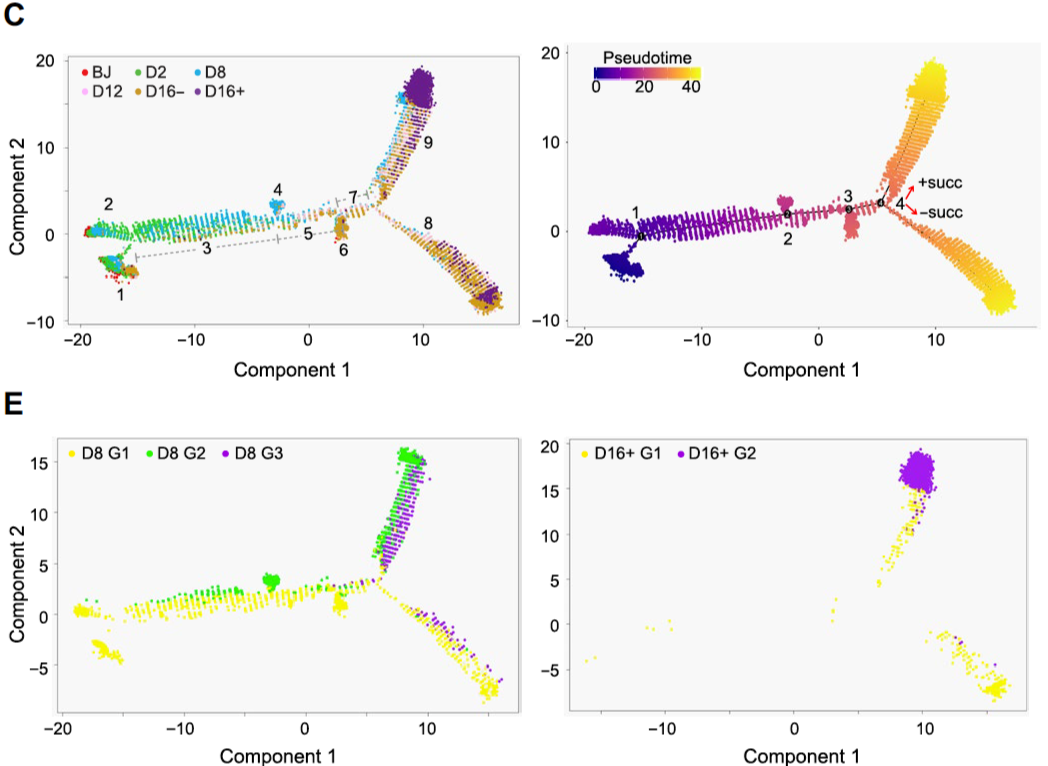
为了知道这些差异产生的原因,作者进一步使用 10X 单细胞转录组库对不同时间点数据进行了细胞轨迹的构建,并在细胞重编程的轨迹中发现了 9 个不同阶段以及 4 个分支事件。令人惊讶的事,作者发现之前在 D8 发现的 G3,以及 D16 的 G2 的细胞亚群是干细胞特征最明显的阶段 9 的主要组成部分,意味着这些细胞亚群成为干细胞的机率远远大于其他细胞。如果我们能在重编程的早期 D8 就将这些细胞分离出来,就可以大大提高重编程的效率。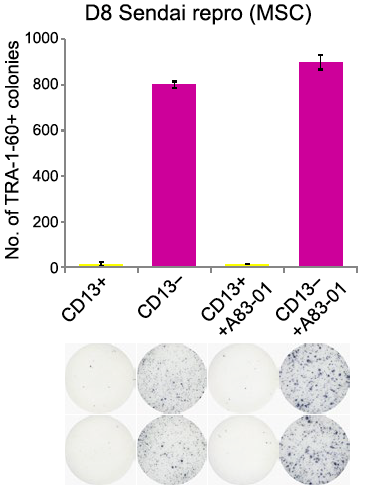
为了找到这些细胞,作者对荧光标记分子库进行了筛选,并发现荧光染料 BDD2-C8 能有效的区分出重编程能力更强的细胞。除此之外,作者通过分析 scRNA-Seq 的数据识别出多个能区分这些细胞的表面标记物。研究团队选取了 CD13,CD44 和 CD201 进行了进一步的验证。结果显示,通过这些表面标记物分离出的 D8 细胞,成功重编程形成干细胞克隆的数量出现了显著的提高。作者并没有满足于识别出这些表面标记物。利用单细胞测序的高精度,他们对重编程「黑盒」还想了解更多。所以他们对经过 CD13 分离的 D8 细胞建立了 10X 单细胞转录组文库,并发现这些 D8 细胞还能被细分为 8 个亚群。通过和另外两个标记物 CD44 和 CD201 交叉分析,作者在这些分离过的细胞中找到一种处于干细胞 / 非干细胞中间状态的 CD13−CD201 + 细胞亚群,同时, CD13−CD201−细胞亚群重编程的能力最强,而 CD13+CD201 + 反之。利用单细胞技术强大的分辨率,作者更加深入的细分精确的找到了重编程潜力更大细胞亚群。

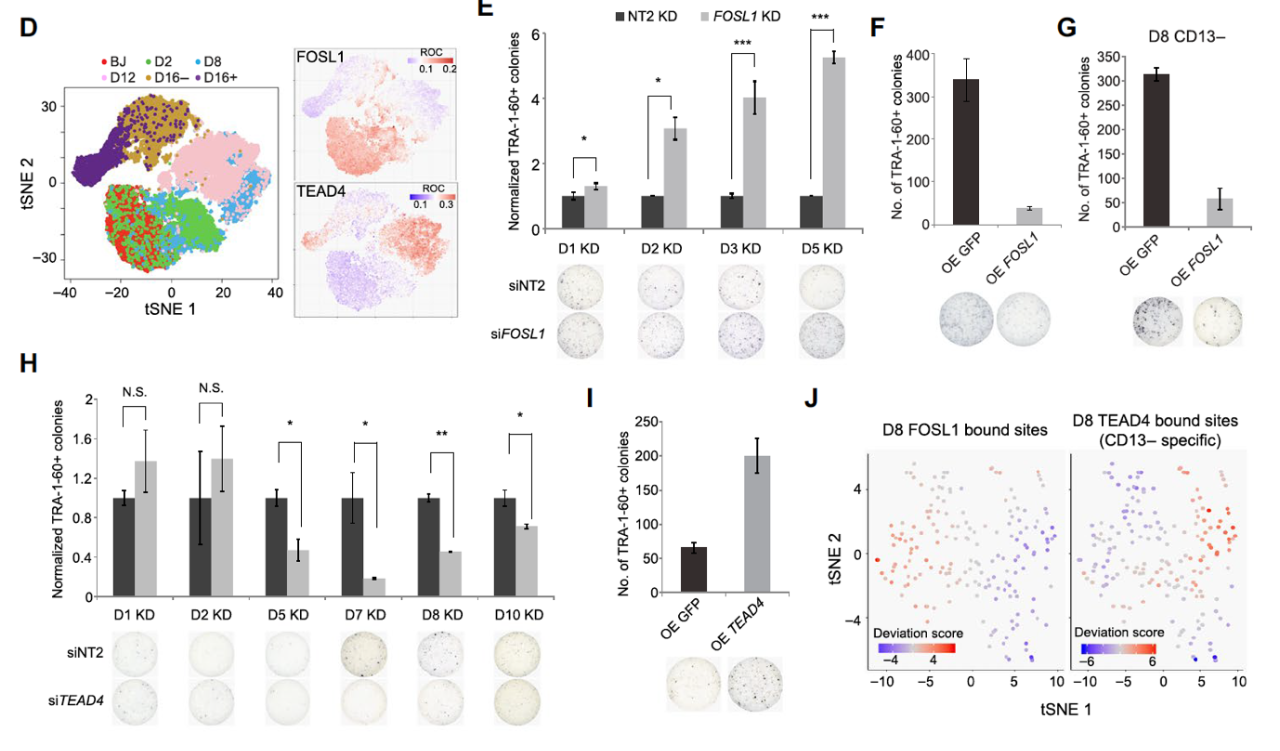
在找到了早期就能分辨出重编程潜力的标记物后,作者开始解决「为什么」的问题。利用单细胞层面的 ATAC-seq,作者探究了是什么转录因子导致了这种重编程能力的差异。在将庞大的 scATAC-seq 数据和此前的 scRNA-seq 结果进行整合后,作者挖掘出了数个决定细胞重编程命运的转录因子,包括 FOSL1, CEBPA, GAPA1:TAL1, TEAD4 和 FOXL1。
最后,作者对在中间状态中「互斥」的转录因子 FOSL1 和 TEAD4 进行了验证。此前的 scRNA-seq 和 scATAC-seq 联用产生的结果显示 FOSL1 与 TEAD4 分别是重编程的抑制因子和促进因子,且表达趋势完全相反。结果不出所料,在敲低 FOSL1 和敲入 TEAD4 后,作者观察到了细胞重编程克隆数的显著上升,证明他们通过双单细胞测序技术联用是一种行之有效的分析手段,并能够找到重编程过程中的关键因子。现在,高通量技术几乎成了顶刊研究中的标配。利用诸如 RNA-seq 的高通量技术,我们能够从样品中抽离出远超从前的信息。可由于生物体的复杂,科学家也逐渐发现单种高通量技术的力不从心,我们需要更加强大的研究工具。所以,将多种高通量手段联用的多组学技术(Multi-Omics)应运而生。该研究将多组学技术应用在单细胞层面,通过结合 scRNA-seq 和 scATAC-seq,成功打开了细胞重编程过程的「黑箱」。在发现能够提高重编程效率方法的同时,也再次证明多组学技术的巨大价值。除此之外,该研究还产生了庞大数据库,作者仅验证了其中非常小的一个部分。相信其余数据也会作为强大的资源来帮助相关研究的开展。本文并不是 Jonathan Loh Yuin-Han 团队将单细胞层面的多组学技术应用在干细胞研究的第一次尝试。

上个月,本篇 Science Advances 的第一作者邢巧瑞博士(Dr Qiaorui Xing)和 Chadi A EI Farran 博士就在生物信息领域著名期刊 Genome Research 发表了题为 Parallel bimodal single-cell sequencing of transcriptome and chromatin accessibility 的研究 [2],报道了一种能够在干细胞中同时获得单细胞转录组和可接近性染色质的测序方法,名为「单细胞转录组与可接近性染色质测序技术」(Assay for Single-cell Transcriptome and Accessibility Regions (ASTAR-seq), ASTAR 也是作者所在新加坡科技研究所的英文缩写)。我们也相信,在单种组学和单细胞技术更加成熟的未来,多组学研究将为生物学研究打开新世界的大门。
![]()










![[精选]SCI论文写作投稿资料包:100+资料& 投稿答疑30问](https://img1.dxycdn.com/p/s14/2023/1105/569/6102685706544601271.jpg!wh200)