关于R语言的亚组分析
青年放疗科医生陈
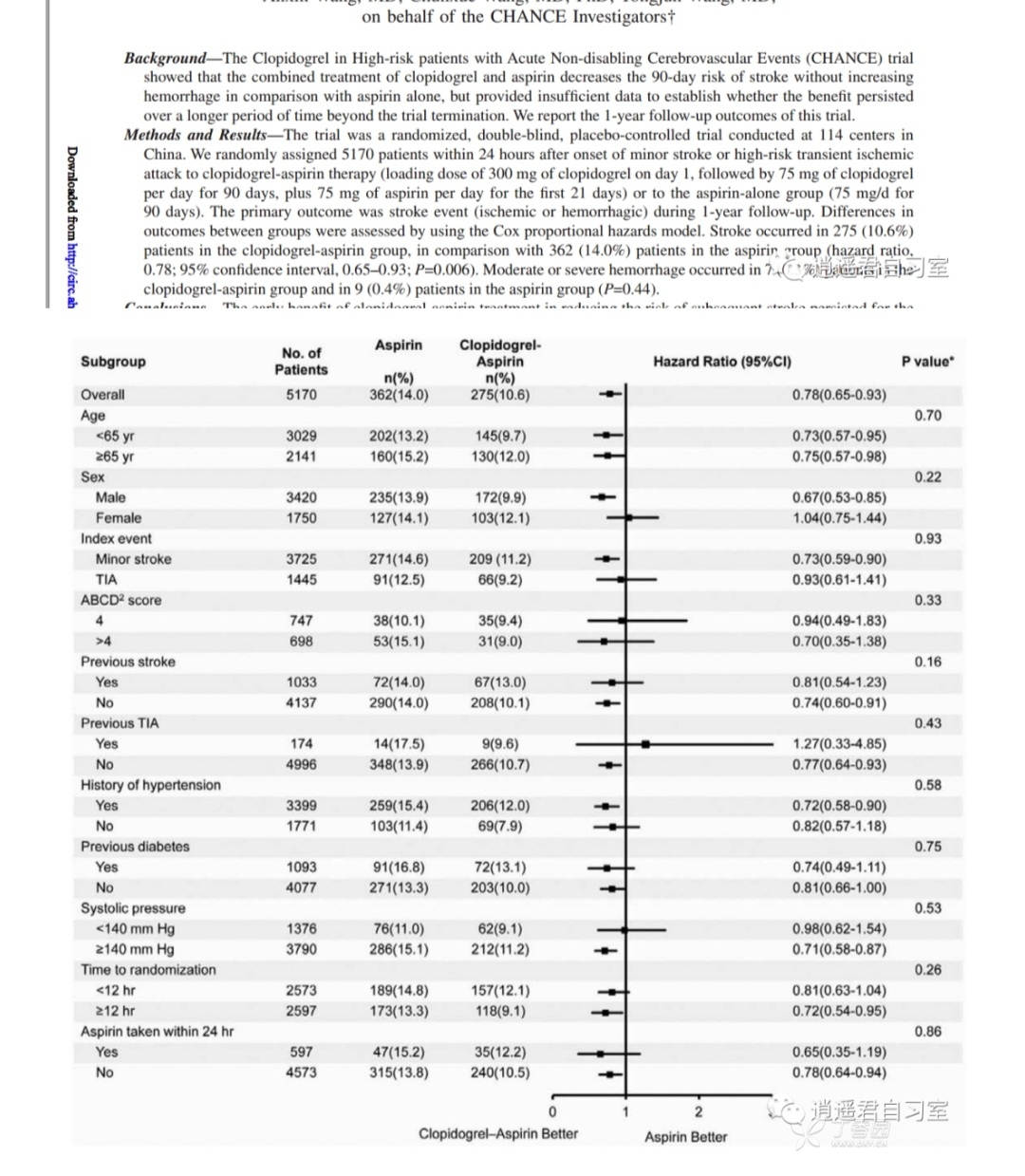 像这个图如果是做总的cox回归并森林图可视化我会做,但是分组对比的还没摸索到,请问是在R语言coxph()函数的参数做分层调整吗?
像这个图如果是做总的cox回归并森林图可视化我会做,但是分组对比的还没摸索到,请问是在R语言coxph()函数的参数做分层调整吗?
2 个回答
汤姆卜丽波
嗯,你先用包把你的临床样本分层然后再画森林图,你是要分高危低危么
青年放疗科医生陈
不是啊,因为单因素多因素分析没有什么意义,想进一步的做亚组/分层分析探索
loveliufudan
在R语言中,您可以使用coxph函数来进行Cox回归分析,并使用参数进行分组对比和分层调整。以下是一些示例代码,可以帮助您进行这样的分析:
首先,您需要加载适当的R包,如survival和forestplot:
```R
library(survival)
library(forestplot)
```
接下来,您可以创建一个Surv对象来定义生存时间和事件状态:
```R
# 假设您的数据框名为data,并包含适当的变量
surv_obj <- with(data, Surv(time, event))
```
然后,您可以使用coxph函数进行Cox回归分析,并进行分层调整。假设您的分组变量为"group",调整变量为"adjust_var":
```R
cox_model <- coxph(surv_obj ~ group + strata(adjust_var), data = data)
```
请注意,通过在模型中使用strata()函数,您可以将"adjust_var"指定为分层变量,以确保在分析中进行正确的分层调整。
接下来,您可以提取Cox回归的结果,包括估计的风险比(Hazard Ratio)和置信区间:
```R
coefficient <- coef(cox_model)
hazard_ratio <- exp(coefficient)
conf_interval <- confint(cox_model)
```
最后,您可以使用forestplot包来可视化分组对比结果:
```R
# 创建一个包含组名、风险比和置信区间的数据框
forest_data <- data.frame(Group = c("Group A", "Group B"),
HR = hazard_ratio,
Lower_CI = conf_interval[, "lower"],
Upper_CI = conf_interval[, "upper"])
# 使用forestplot函数进行可视化
forestplot(forest_data,
xlog = TRUE,
is.summary = c(TRUE, FALSE, FALSE),
clip = c(0.1, 10),
col = fpColors(box = "black", line = "black", summary = "black"))
```
请根据您的实际数据和变量进行适当的调整,并确保您在分析和解释结果时遵循统计学和临床研究的最佳实践。
青年放疗科医生陈
你好朋友,cox_model <- coxph(surv_obj ~ group + strata(adjust_var), data = data),为什么我的数据运行了这句关键代码后没有出现分层的结果,仅p值发生了改变呀😨
相关产品推荐
相关问答