我能不能做生信?生信难吗?
科研论文时间
什么是生信,现在有没有必要学,怎么学?相信一提到生信,很多人是又爱又恨的。爱的是现在流传的一种让人心动的说法,「不用做实验就能发文章,好棒啊」。恨的是「R 语言太难了吧,我不会写代码!」或「我数学不好,统计学差,几万个数据怎么处理看得头都大」。
就像朋友跟我讲的「找到数据库后,后续不知道怎么做了,密密麻麻的看着密集恐惧症都犯了」。
今天,我们就来聊一聊,生信有那么可怕吗?
一、生物信息学的前世今生
「生物信息学」,这个名字听起来特别高大上。看名字除了生物学,又是计算机,又是信息学的。很多人一打开生信文章就被它各种复杂图表吓倒,如下图。
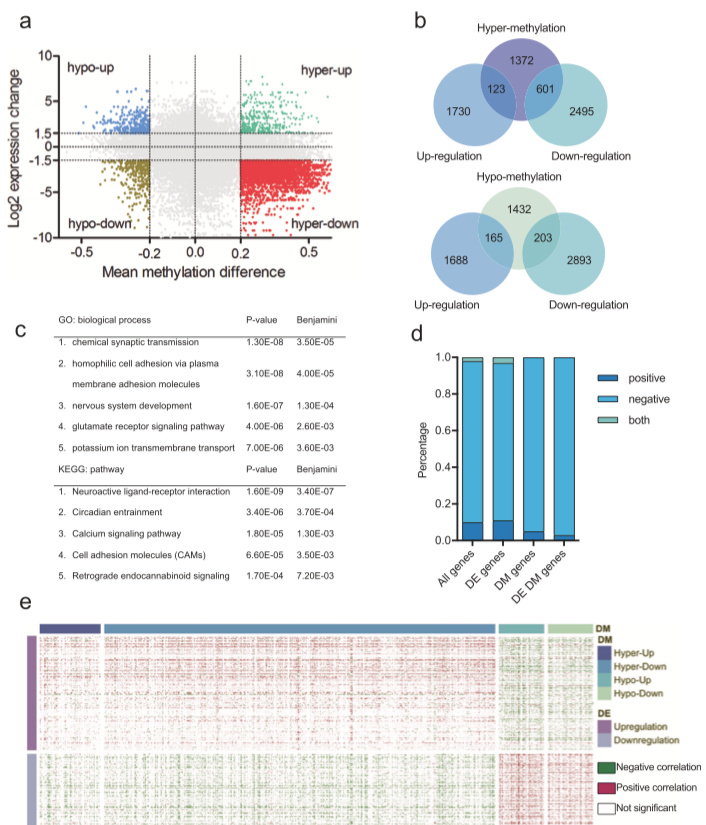
图片来源:文献截图
但实际上不用怕的,阿 Q 一点你甚至可以这么想「如果我学会了,是不是很牛逼?」生信表面上看起来难,但我自己学习时的理念一直都是:生信是生物学和计算机的交叉学科,计算机大神多,很多技术上的难题都已经帮我们解决,跟着大神的脚步走就可以了。
今天,想跟大家简单介绍下生信的前世今生,并通过一个小例子告诉大家,生信并不可怕,生信可以学!
个人十分推荐学新东西的时候花点时间了解它的历史,了解它在什么背景下为解决什么问题而出现,目前进展到什么程度,将来又将去往何方。
如果你想了解一个疾病或一个分子,一份高质量的 Review 是很不错的选择。但对于一项新技术,百度百科或维基百科就够了。
图片来源:文献截图
如维基百科\百度百科的介绍:「Bioinformatics」是 Biology(生物学)+information(信息学)+omics(组学)以及数学和统计学组成的新兴交叉学科。
它伴随着上个世纪八九十年代人类基因组计划的诞生而出现。「人类基因组计划其宗旨在于测定组成人类染色体(指单倍体)中所包含的 30 亿个碱基对组成的核苷酸序列,从而绘制人类基因组图谱,并且辨识其载有的基因及其序列,达到破译人类遗传信息的最终目的(百度百科)」。
30 亿个碱基对这么庞大的数据,做过实验的大家都知道这个叫「原始数据」,本身并没有什么意义,就是些数值而已。得到这些数据后如何处理、分析,以解决现实生活中的问题,是科学家们进一步思考的问题。
由于传统的生物学方法无法处理这么庞大的数据量,利用计算机强大的计算能力,生物信息学运应而生。
这就是生物信息学诞生最初的背景。近年来的蓬勃发展,主要归功于测序技术的飞速迭代。早期人类基因组计划 1 个碱基对测序的价格在 1 美元,全基因组测下来 30 亿美元。而如今,基因组已经进入了千元时代,人人都可以想做就做,因而产生了大量的有待分析数据。
二、生物信息学的基本功能
了解了什么是生物信息学,下一步就是要知道怎么利用它来解决实际问题,包括指导基础实验方向或结合临床数据解决实际问题等。
这里就回到一开始的问题:
「用生信手段辅助自己课题进展,一定要会写代码,会复杂的 R 语言并掌握大量的数学、统计知识才能开始吗?」答案是否定的。
上面提到的知识都懂当然最好,但作为一个临床医生或医学生,显然是不现实的,不然计算机或统计学专业的人早该下岗了。
幸运的是,计算机高手们理解我们的需求并已经帮我们解决了相当一部分问题。诸多在线工具,简单几步操作便能高效产出自己想要的效果。
不信我们来试一试。现在,就让我们看看如何用在线工具——「GEO2R」画一张漂亮又高大上的差异基因表达 Differentially Expressed Genes(DEGs)火山图,并理解其基本含义及价值。
生信的最基本步骤是对从数据库检索出的数据进行 「差异基因筛选」。我们就以此为例,看看统计学家和计算机学家有多贴心。
图片来源:网页截图
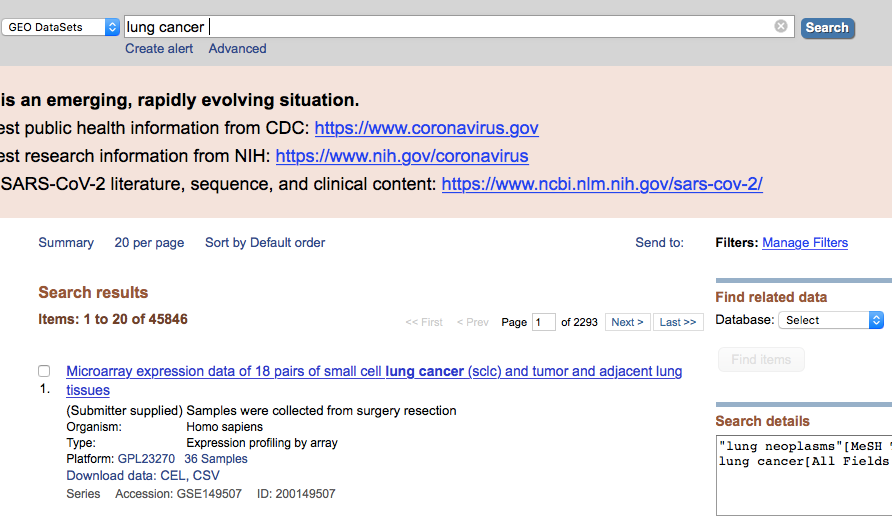
这里我们选用 GEO 数据库,GEO 数据库全称为(GENE EXPRESSION OMNIBUS),感兴趣的可以去了解下,储存了大量肿瘤跟非肿瘤的数据。
首先进入 PubMed,选择 GEO DateSets,在此以「lung cancer」检索出的第一个数据为例。点击打开后,我们会看到对这个数据集的简单介绍,类似文章的摘要,介绍作者用的什么芯片,有多少例病人,以及如何分组等。
我们直接点击「Analyze with GEO2R」试试。
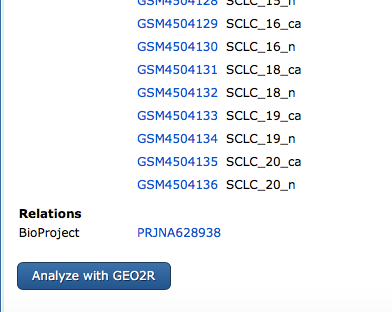
图片来源:网页截图
根据作者定义的「Source name」我们可以点击「Define groups」进行待分析组别的定义。然后,左键点击勾选,得到如下图。
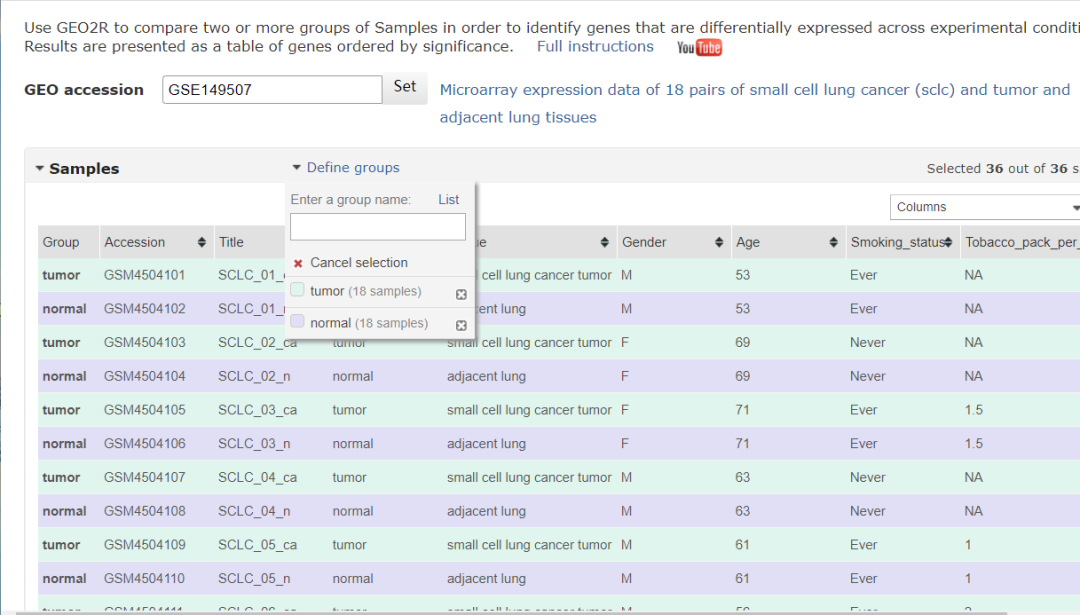
图片来源:网页截图
这时候我们把需要分析的数据都选上了,直接拉到底部点击「Analyze」开始分析。
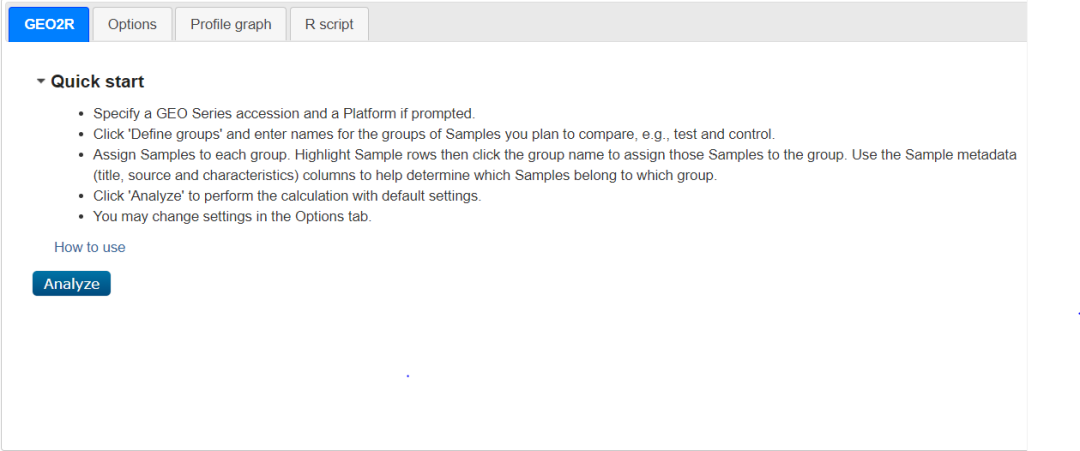
图片来源:网页截图
短暂的等待,神奇的事情就发生啦,我们会得到如下界面,图可以直接保存,数据可以下载!
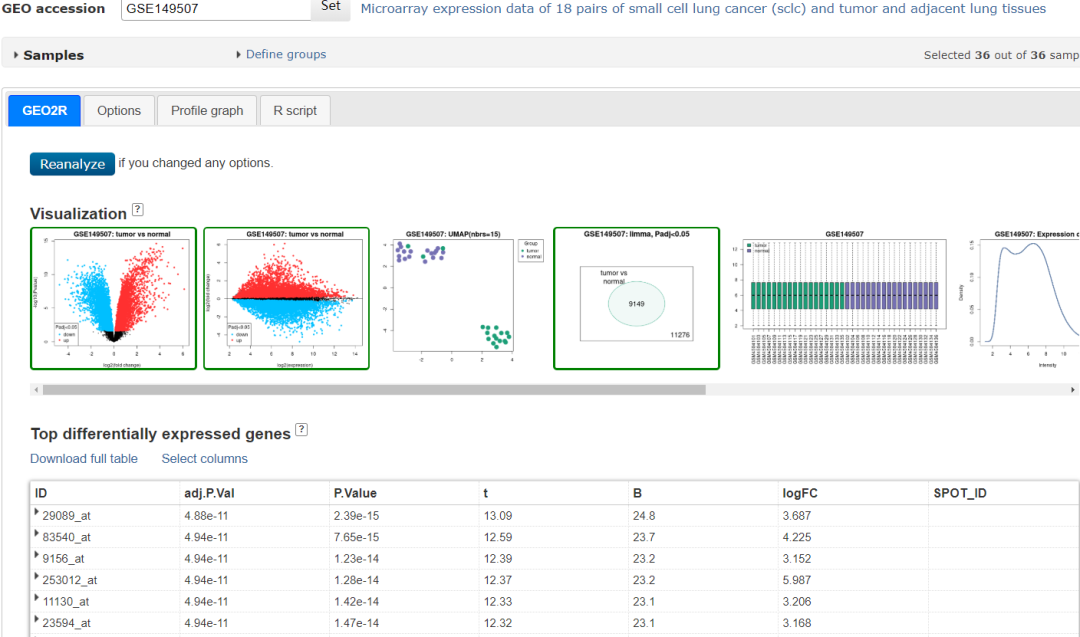
图片来源:网页截图
有没有上面觉得很漂亮,很棒!我们暂时不展开解释这些图的意义,而是来看看统计学家和计算机大神们有多贴心。
刚刚我们直接选择了「Analyze」,会用鼠标,一键点击就能出图。
我们再回过头来「GEO2R」旁边的「Options」,你会看到这个在线工具选用了什么统计方法。
比如这里用的「Benjamini & Hochberg」以及后面几栏内容,相信大多数临床医生是看不懂的。但没关系,工具就是这么傻瓜式,选默认值就可以!
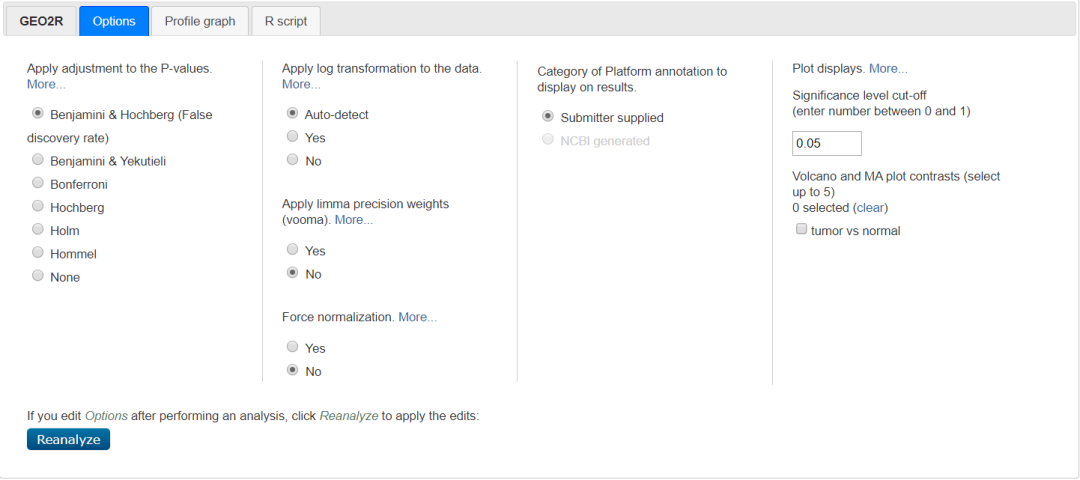
图片来源:网页截图
这些漂亮的图通过 R 语言进行可视化的,不知不觉中你已经用到 R 语言了,是不是自己都没注意到?
点击第四栏「R script」,鼠标滚动一下就能看到密密麻麻的代码。学过编程的都知道,一个空格敲错电脑都会报错,代码别人已经帮你写好了,很幸福有没有!
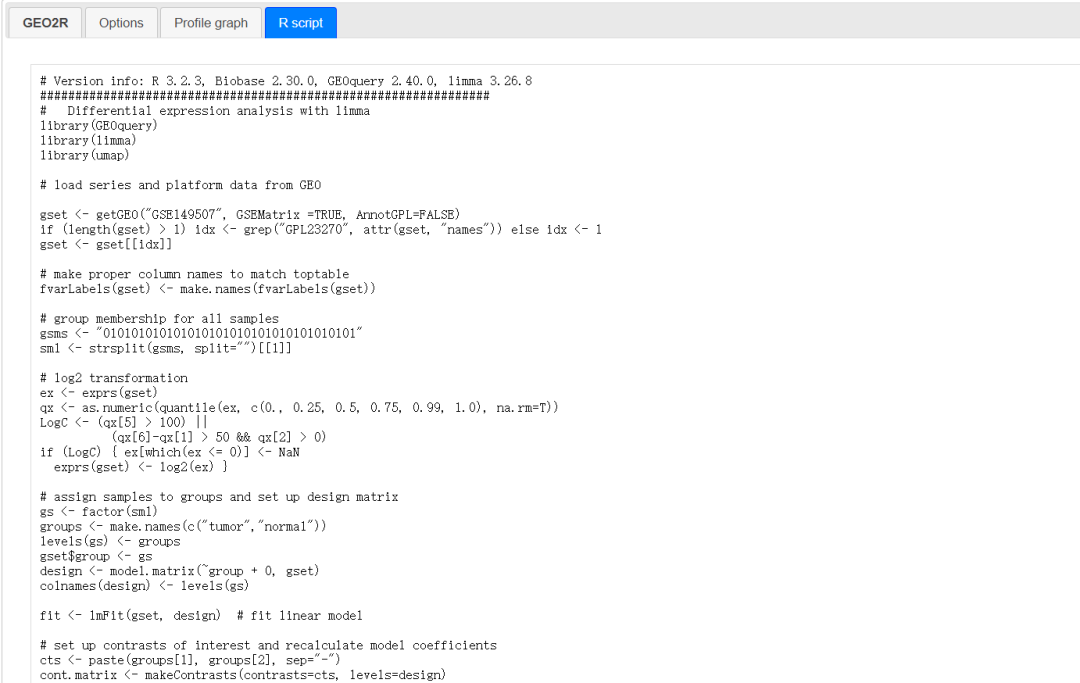
图片来源:网页截图
怎么样,到这里学生信是不是有点信心了?
今天,我们只展示了差异基因筛选的在线插件使用。生信后续的数据处理其实类似,你懂 R 语言最好,但很多时候是可以借助一些公认可以使用的工具跳过的。
具体如何使用有机会后续再交流,总之,生信可以学,要有信心!