【基础知识】生信分析文章套路原来这么简单!
科研论文时间
1. 生信分析文章范例解读
最近在检索文献时发现了 10 月有一篇生信文章,做的是关于三阴性乳腺癌中相关 ceRNA 差异表达谱的综合分析,于 2018 年 10 月 11 日发表在《Cellular physiology and biochemistry》杂志上,IF = 5.5。
由于小编就是做三阴性乳腺癌 non-coding RNA 的相关研究,一直都有关注 ceRNA 的研究,类似这篇文章的工作量投稿分数应为 3-5 分,而此文竟然突破 5 分门槛。
仔细阅读发现,原来这篇文章与常规生信分析文章相比,增加了 30 例样本的 q-PCR 验证,按照小编做实验的效率,大概也就两天工作量。
所以接下来我们一起走进这篇文章,感受他的总体思路,看看是不是你与 5 分只差两天的距离?

三阴性乳腺癌(TNBC)是高度恶性乳腺癌的亚型,预后不良。越来越多的证据表明,长链非编码 RNA(lncRNA)在包括乳腺癌在内的多种癌症的发展和进展中发挥着重要的调节作用。
此研究利用癌症基因组图谱(TCGA)的 RNA-Seq 数据比较了 111 个 TNBC 组织和 104 个非癌组织中 mRNA,lncRNA 和 miRNA 的表达谱。
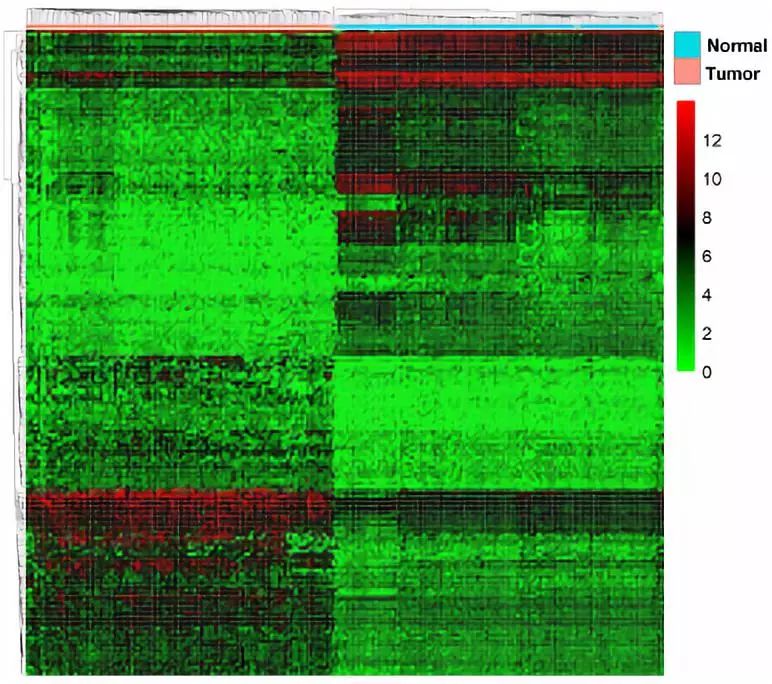
之后进行 GO 和 KEGG 途径分析失调的 mRNA 的主要功能。并进行 Kaplan-Meier 存活分析以确定差异表达的 lncRNA / mRNA / miRNA 对总体存活的影响。
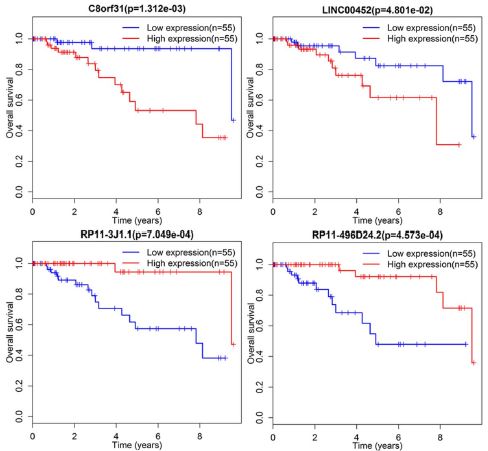
随后,本研究构建了一个竞争性内源 RNA(ceRNA)网络,其中包括66 个失调的 lncRNAs,24 个 miRNA 和 55 个 mRNA 。并通过 qRT-PCR 分别在 30 对样品中在 ceRNA 网络中证实了四种失调的 lncRNA,三种异常表达的 miRNA 和四种 mRNA 。
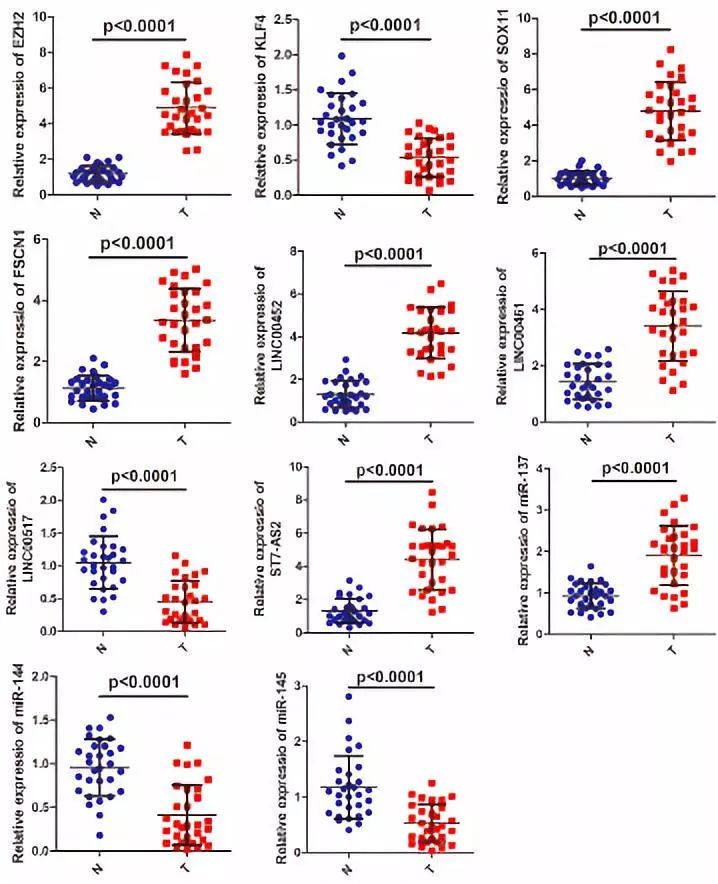
得出以下结论:根据存活分析,109 个 lncRNA 和 124 个 mRNA 可作为 TNBC 患者的预后信号;而功能分析显示,ceRNA 网络中的 19 种 mRNA 在 17 种癌症相关途径中富集。
2. 生信分析文章套路解析
通过对范文的解读,我们可以理出生信分析文章的大致思路:通过数据库筛选,加少量的实验验证得出结论。
TCGA 数据库:
此文章所用的数据库 TCGA 是一个公开可用的数据库,是数据挖掘和生物发现的丰富且有价值的在线资源,迄今为止,它包含超过 10,000 种患者样本,涵盖 36 种癌症类型。
本研究的目的就是在大样本中发现特异性 lncRNA 标记,对 mRNA,lncRNA 和 miRNA 表达谱进行了全面分析,并在 TCGA 数据库中预测了大量 TNBC 患者中代表性 lncRNA 的可能生物学功能和临床结果。
GO 和 KEGG:
GO 和 KEGG 分析是对差异基因进行注释。为什么要进行注释?因为筛选的差异基因可能有很多,那么到底哪个基因比较重要,哪个基因更可能与疾病相关?
通过 GO 和 KEGG 分析,我们通过了解该基因已有的信息,把预测的尺度放得小一点,那么预测的准确性相应得就会提高一些;另外通过功能注释,我们还可以了解到这个基因可能是在疾病发生过程中的哪个环节产生了作用。
3. 总结
生信分析对科研人员来说,有一定难度,但是一旦掌握了这项技能,那无疑就是发文章最省时省力省钱的方式了,小编非常建议那些经费或资源稍欠的研究生,有时间可以学习一下生信分析。
总结生信文章套路,一句话总结:下载别人已经完成的芯片数据,分析筛选差异基因,构建蛋白质互作网络,最后利用 GO 和 KEGG 分析。