「谁」动了我的扩增曲线?
赛默飞
各位老师在做荧光定量 PCR 实验的时候,是否遇到过非标准「S」型曲线的问题呢?俗话说的好,完美的 PCR 扩增曲线「千篇一律」,而有问题的 PCR 扩增曲线则是「千姿百态」。今天给大家分享一个案例:客户反映做绝对定量过程中,扩增曲线在线性增长期出现荧光信号突然下降的情况,在多组分图(Multicomponent Plot)中查看原始信号采集情况时,这种现象更加明显。
 图 1. 实验数据多组分图(MulticomponentPlot)
图 1. 实验数据多组分图(MulticomponentPlot)
好好的扩增曲线怎么就突然「不走寻常路」了,到底是「谁」动了我的扩增曲线呢?
其实,这种现象并不是偶发的,有很多原因可能导致,并且有个专属名称「Hook Effect」,翻译过来叫「鱼钩效应」。「鱼钩效应」指在荧光定量 PCR 过程中,DNA 扩增到指数增长期后出现的一种荧光信号不能保持稳定(或者上升)而出现下降的现象 [1]。
导致扩增曲线出现这一现象的因素比较复杂,这里我们分别从嵌入性染料法与探针法两种定量方式的角度为大家解释:
嵌入性染料法:
嵌入性染料法(例如 SYBR Green I)产生「鱼钩效应」主要是由于模板序列混杂,在 PCR 过程中易形成异源双链,造成同源/异源产物并存的状态,而异源双链熔解温度要低于同源双链。
在 PCR 反应最初的几个循环,引物充足,延伸步骤产生的 DNA 双链完美匹配,100% 互补,熔解温度比较高;随着循环数增加,PCR 产物不断累积,引物退火与产物自退火之间形成竞争,异源双链产生,阻碍了引物的退火和延伸;若异源双链熔解温度低于延伸反应温度(即荧光信号采集温度),则会在荧光采集步骤发生解链反应,导致荧光信号下降 [1]。随着自退火产物的增加,「鱼钩效应」更加明显。
这种「鱼钩效应」我们平时遇到的并不多,究竟什么样的模板能够在扩增过程中产生异源双链呢?主要是那些产物序列中既有一致性序列又有差异性序列的应用,例如:带有统一接头序列的 NGS 文库定量、带有一致保守区序列的 16S 宏基因组定量,以及定量模板中含有突变位点的应用都可能产生异源双链。
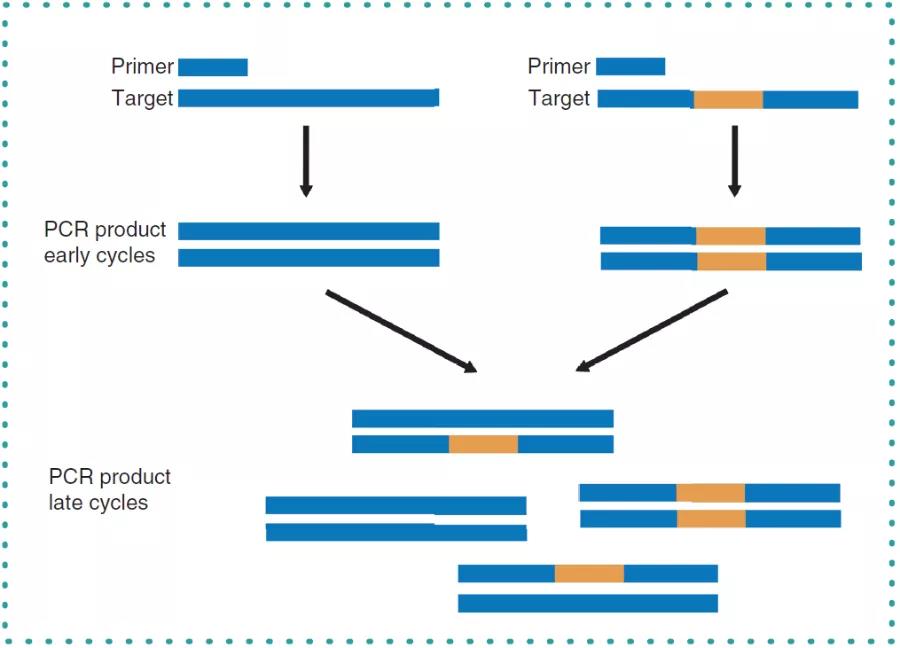 图 2. PCR 扩增异质靶标,产物自退火引发「鱼钩效应」 [1]
图 2. PCR 扩增异质靶标,产物自退火引发「鱼钩效应」 [1]
既然清楚了嵌入性染料法产生「鱼钩效应」的原理,也就找到了对应的解决方案:1. 减少 PCR 反应循环数,比如从 35 个循环下降到 28 个循环,产物自退火尚未发生,引物延伸反应处于优势地位,扩增曲线正常;2. 降低延伸步骤反应温度,使其低于异源双链的熔解温度,比如延伸温度从 77℃ 降到 72℃,扩增曲线也是正常的。
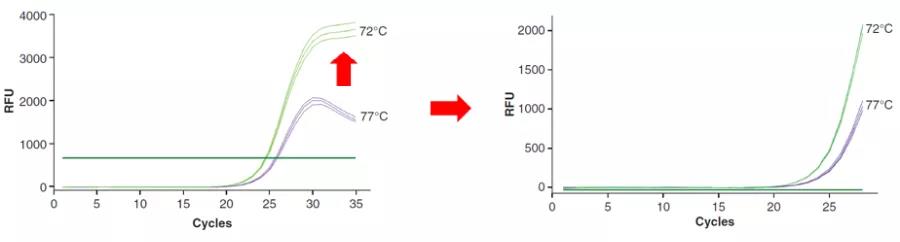 图 3. 嵌入性染料法荧光定量消除「鱼钩效应」的方式 [1]
图 3. 嵌入性染料法荧光定量消除「鱼钩效应」的方式 [1]
虽然能够引起「鱼钩效应」的异源双链错配程度无法估计,但单碱基水平错配还不足以引起 PCR 产物熔解温度发生明显改变。
探针法:
除了染料法,探针法荧光定量 PCR 实验也有可能产生「鱼钩效应」。采用杂交探针,扩增子链和探针之间会形成竞争,荧光信号明显下降,一般发生在模板浓度比较高、单链扩增子重新退火的速度比其与探针结合速度更快的情况下。Barratt 等人采用一步法对 HSV 病毒进行分型,结果发现扩增曲线产生了「鱼钩效应」,为了使反应能更多地产生与探针结合的那条单链,他们采用了不对称 PCR(反向引物浓度高于正向引物浓度)的方式,大大改善了「鱼钩效应」 [2]。
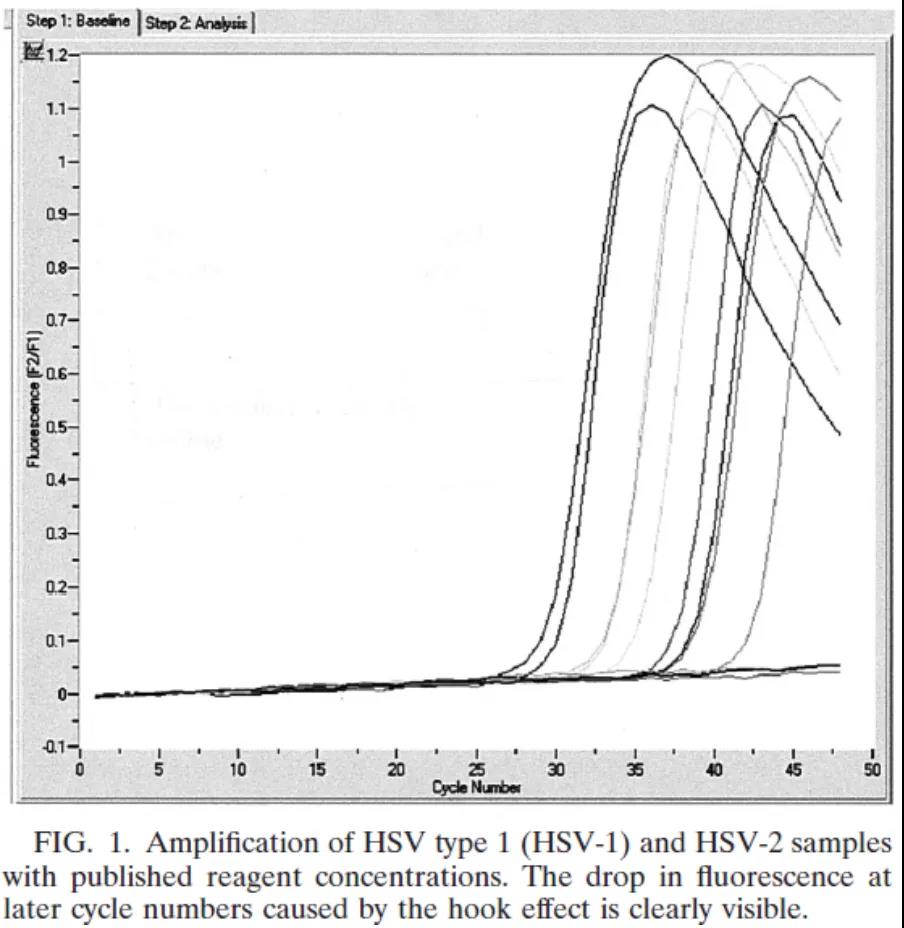
 图 4. 杂交探针法发生「鱼钩效应」及解决方案 [2]
图 4. 杂交探针法发生「鱼钩效应」及解决方案 [2]
「鱼钩效应」对灵敏度、熔解曲线、数据分析有一定的影响,但这种现象一般发生在指数增长期之后,对数据分析着眼点放在指数增长期的绝对定量和相对定量结果影响不大 [3];但如果您是做等位基因分型或者法医相关应用,可以根据实际情况,通过合理设置基线、适当调整模板及 PCR 反应体系其他组分的浓度、降低反应循环数以及进行不对称 PCR 的方式来改善结果。
最后,我们回到文章开头的那个案例,沟通后我们发现客户采用的是 SYBR Green I 染料法对 NGS 文库进行绝对定量,NGS 文库构建过程中,需要在测序片段两端加上可以被测序仪识别的「接头」序列,而这些「接头」大部分序列是一致的,导致文库中存在大量异质但又相互关联的 DNA 序列,在定量过程中形成异质双链,进而引发了「鱼钩效应」。
好了,亲爱的小伙伴儿们,今天的 troubleshooting case 就分享到这里,如果您在实验过程中发现什么有趣的现象也可以联系我们,让我们共同学习、进步。