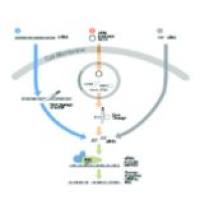
RNAi实验原理、方法和应用
丁香园论坛
6509
我用附件传不上来,只能用这种笨法了,希望大家用的上
近年来的研究表明,将与mRNA对应的正义RNA和反义RNA组成的双链RNA(dsRNA)导入细胞,可以使mRNA发生特异性的降解,导致其相应的基因沉默。这种转录后基因沉默机制(post-transcriptional gene silencing, PTGS)被称为RNA干扰(RNAi)。 一、RNAi的分子机制 通过生化和遗传学研究表明,RNA干扰包括起始阶段和效应阶段(inititation and effector steps)。在起始阶段,加入的小分子RNA被切割为21-23核苷酸长的小分子干扰RNA片段(small interfering RNAs, siRNAs)。证据表明;一个称为Dicer的酶,是RNase III家族中特异识别双链RNA的一员,它能以一种ATP依赖的方式逐步切割由外源导入或者由转基因,病毒感染等各种方式引入的双链RNA,切割将RNA降解为19-21bp的双链RNAs(siRNAs),每个片段的3’端都有2个碱基突出。 在RNAi效应阶段,siRNA双链结合一个核酶复合物从而形成所谓RNA诱导沉默复合物(RNA-induced silencing complex, RISC)。激活RISC需要一个ATP依赖的将小分子RNA解双链的过程。激活的RISC通过碱基配对定位到同源mRNA转录本上,并在距离siRNA3’端12个碱基的位置切割mRNA。尽管切割的确切机制尚不明了,但每个RISC都包含一个siRNA和一个不同于Dicer的RNA酶。 另外,还有研究证明含有启动子区的dsRNA在植物体内同样被切割成21-23nt长的片段,这种dsRNA可使内源相应的DNA序列甲基化,从而使启动子失去功能,使其下游基因沉默.二、如何进行RNAi试验(一)siRNA的设计1. 在设计RNAi实验时,可以先在以下网站进行目标序列的筛选:http://www.genesil.com/business/products/order2.htmhttp://www.ambion.com/techlib/misc/siRNA_finder.htmlhttp://www.ic.sunysb.edu/Stu/shilin/rnai.htmlhttp://design.dharmacon.com/rnadesign/default.aspx?SID=453587102.RNAi目标序列的选取原则:(1)从转录本(mRNA)的AUG起始密码开始,寻找“AA”二连序列,并记下其3'端的19个碱基序列,作为潜在的siRNA靶位点。有研究结果显示GC含量在45%—55%左右的siRNA要比那些GC含量偏高的更为有效。Tuschl等建议在设计siRNA时不要针对5'和3'端的非编码区(untranslated regions,UTRs),原因是这些地方有丰富的调控蛋白结合区域,而这些UTR结合蛋白或者翻译起始复合物可能会影响siRNP核酸内切酶复合物结合mRNA从而影响siRNA的效果。(2)将潜在的序列和相应的基因组数据库(人,或者小鼠,大鼠等等)进行比较,排除那些和其他编码序列/EST同源的序列。例如使用BLAST( www.ncbi.nlm.nih.gov/BLAST/)(3)选出合适的目标序列进行合成。通常一个基因需要设计多个靶序列的siRNA,以找到最有效的siRNA序列。3.阴性对照 一个完整的siRNA实验应该有阴性对照,作为阴性对照的siRNA应该和选中的siRNA序列有相同的组成,但是和mRNA没有明显的同源性。通常的做法是将选中的siRNA序列打乱,同样要检查结果以保证它和目的靶细胞中其他基因没有同源性。4.目前已证实的siRNA可以在下面的网页找到:http://design.dharmacon.com/catalog/category.aspx?key=49http://www.ambion.com/techlib/tb/tb_502.htmlhttp://web.mit.edu/mmcmanus/www/siRNADB.htmlhttp://python.penguindreams.net/Order_Entry/jsp/BrowseCatalog.jsp?Category=Published(二)siRNA的制备 目前为止较为常用的方法有通过化学合成,体外转录,长片断dsRNAs经RNase III 类降解 (e.g. Dicer, E. coli, RNase III)体外制备siRNA,以及通过siRNA表达载体或者病毒载体,PCR制备的siRNA表达框在细胞中表达产生siRNA。体外制备1.化学合成 许多国外公司都可以根据用户要求提供高质量的化学合成siRNA。主要的缺点包括价格高,定制周期长,特别是有特殊需求的。由于价格比其他方法高,为一个基因合成3—4对siRNAs 的成本就更高了,比较常见的做法是用其他方法筛选出最有效的序列再进行化学合成。最适用于:已经找到最有效的siRNA的情况下,需要大量siRNA进行研究不适用于:筛选siRNA等长时间的研究,主要原因是价格因素2.体外转录 以DNA Oligo为模版,通过体外转录合成siRNAs,成本相对化学合成法而言比较低,而且能够比化学合成法更快的得到siRNAs。不足之处是实验的规模受到限制,虽然一次体外转录合成能提供足够做数百次转染的siRNAs,但是反应规模和量始终有一定的限制。而且和化学合成相比,还是需要占用研究人员相当的时间。值得一提的是体外转录得到的siRNAs毒性小,稳定性好,效率高,只需要化学合成的siRNA量的1/10就可以达到化学合成siRNA所能达到的效果,从而使转染效率更高。最适用于:筛选siRNAs,特别是需要制备多种siRNAs,化学合成的价格成为障碍时。不适用于:实验需要大量的,一个特定的siRNA。长期研究。3.用RNase III 消化长片断双链RNA制备siRNA 其他制备siRNA的方法的缺陷是需要设计和检验多个siRNA序列以便找到一个有效的siRNA。而用这种方法——制备一份混合有各种siRNAs “混合鸡尾酒” 就可以避免这个缺陷。选择通常是200—1000碱基的靶mRNA模版,用体外转录的方法制备长片断双链dsRNA ,然后用RNase III (or Dicer) 在体外消化,得到一种siRNAs“混合鸡尾酒”。在除掉没有被消化的dsRNA后,这个siRNA混合物就可以直接转染细胞,方法和单一的siRNA转染一样。由于siRNA混合物中有许多不同的siRNAs,通常能够保证目的基因被有效地抑制。 dsRNA消化法的主要优点在于可以跳过检测和筛选有效siRNA序列的步骤,为研究人员节省时间和金钱(注意:通常用RNAse III通常比用Dicer要便宜)。不过这种方法的缺点也很明显,就是有可能引发非特异的基因沉默,特别是同源或者是密切相关的基因。现在多数的研究显示这种情况通常不会造成影响。最适用于:快速而经济地研究某个基因功能缺失的表型不适用于:长时间的研究项目,或者是需要一个特定的siRNA进行研究,特别是基因治疗体内表达 前面的3种方法主要都是体外制备siRNAs,并且需要专门的RNA转染试剂将siRNAs转到细胞内。而采用siRNA表达载体和基于PCR的表达框架则属于:从转染到细胞的DNA模版中在体内转录得到siRNAs。这两种方法的优点在于不需要直接操作RNA。4. siRNA表达载体 多数的siRNA表达载体依赖三种RNA聚合酶III 启动子(pol III)中的一种,操纵一段小的发夹RNA(short hairpin RNA, shRNA)在哺乳动物细胞中的表达。这三类启动子包括大家熟悉的人源和鼠源的U6启动子和人H1启动子。之所以采用RNA pol III启动子是由于它可以在哺乳动物细胞中表达更多的小分子RNA,而且它是通过添加一串(3到6个)U来终止转录的。要使用这类载体,需要订购2段编码短发夹RNA序列的DNA单链,退火,克隆到相应载体的pol III 启动子下游。由于涉及到克隆,这个过程需要几周甚至数月的时间,同时也需要经过测序以保证克隆的序列是正确的。 siRNA表达载体的优点在于可以进行较长期研究——带有抗生素标记的载体可以在细胞中持续抑制靶基因的表达,持续数星期甚至更久。病毒载体也可用于siRNA表达,其优势在于可以直接高效率感染细胞进行基因沉默的研究,避免由于质粒转染效率低而带来的种种不便,而且转染效果更加稳定。最适用于:已知一个有效的siRNA序列,需要维持较长时间的基因沉默。不适用于:筛选siRNA序列(其实主要是指需要多个克隆和测序等较为费时、繁琐的工作)。5. siRNA表达框架 siRNA表达框架(siRNA expression cassettes,SECs)是一种由PCR得到的siRNA表达模版,包括一个RNA pol III启动子,一段发夹结构siRNA,一个RNA pol III终止位点,能够直接导入细胞进行表达而无需事前克隆到载体中。和siRNA表达载体不同的是,SECs不需要载体克隆、测序等颇为费时的步骤,可以直接由PCR得到,不用一天的时间。因此,SECs成为筛选siRNA的最有效工具,甚至可以用来筛选在特定的研究体系中启动子和siRNA的最适搭配。如果在PCR两端添加酶切位点,那么通过SECs筛选出的最有效的siRNA后,可以直接克隆到载体中构建siRNA表达载体。构建好的载体可以用于稳定表达siRNA和长效抑制的研究。这个方法的主要缺点是①PCR产物很难转染到细胞中②不能进行序列测定,PCR和DNA合成时可能差生的误读不能被发现导致结果不理想。最适用于:筛选siRNA序列,在克隆到载体前筛选最佳启动子不适用于:长期抑制研究。(如果克隆到载体后就可以了)(三)siRNA的转染 将制备好的siRNA,siRNA表达载体或表达框架转导至真核细胞中的方法主要有以下几种:1.磷酸钙共沉淀 将氯化钙,RNA(或DNA)和磷酸缓冲液混合,沉淀形成包含DNA且极小的不溶的磷酸钙颗粒。磷酸钙-DNA复合物粘附到细胞膜并通过胞饮进入目的细胞的细胞质。沉淀物的大小和质量对于磷酸钙转染的成功至关重要。在实验中使用的每种试剂都必须小心校准,保证质量,因为甚至偏离最优条件十分之一个pH都会导致磷酸钙转染的失败。2.电穿孔法 电穿孔通过将细胞暴露在短暂的高场强电脉冲中转导分子。将细胞悬浮液置于电场中会诱导沿细胞膜的电压差异,据认为这种电压差异会导致细胞膜暂时穿孔。电脉冲和场强的优化对于成功的转染非常重要,因为过高的场强和过长的电脉冲时间会不可逆地伤害细胞膜而裂解细胞。一般,成功的电穿孔过程都伴随高水平(50%或更高)的毒性。3.DEAE-葡聚糖和polybrene 带正电的DEAE-葡聚糖或polybrene多聚体复合物和带负电的DNA分子使得DNA可以结合在细胞表面。通过使用DMSO或甘油获得的渗透休克将DNA复合体导入。两种试剂都已成功用于转染。DEAE-葡聚糖仅限于瞬时转染。4.机械法 转染技术也包括使用机械的方法,比如显微注射和基因枪(biolistic particle)。显微注射使用一根细针头将DNA,RNA或蛋白直接转入细胞质或细胞核。基因枪使用高压microprojectile将大分子导入细胞。5.阳离子脂质体试剂 在优化条件下将阳离子脂质体试剂加入水中时,其可以形成微小的(平均大小约100-400nm)单层脂质体。这些脂质体带正电,可以靠静电作用结合到DNA的磷酸骨架上以及带负电的细胞膜表面。因此使用阳离子脂质体转染的原理与以前利用中性脂质体转染的原理不同。使用阳离子脂质体试剂,DNA并没有预先包埋在脂质体中,而是带负电的DNA自动结合到带正电的脂质体上,形成DNA-阳离子脂质体复合物。据称,一个约5kb的质粒会结合2-4个脂质体。被俘获的DNA就会被导入培养的细胞。现存对DNA转导原理的证据来源于内吞体和溶酶体。 为了达到高的转染效率,在转染实验过程中,需要注意以下几点:1.纯化siRNA 在转染前要确认siRNA的大小和纯度。为得到高纯度的siRNA,推荐用玻璃纤维结合,洗脱或通过15-20%丙烯酰胺胶除去反应中多余的核苷酸,小的寡核苷酸,蛋白和盐离子。注意:化学合成的RNA通常需要跑胶电泳纯化(即PAGE胶纯化)。 2.避免RNA酶污染 微量的RNA酶将导致siRNA实验失败。由于实验环境中RNA酶普遍存在,如皮肤,头发,所有徒手接触过的物品或暴露在空气中的物品等,此保证实验每个步骤不受RNA酶污染非常重要。3.健康的细胞培养物和严格的操作确保转染的重复性 通常,健康的细胞转染效率较高。此外,较低的传代数能确保每次实验所用细胞的稳定性。为了优化实验,推荐用50代以下的转染细胞,否则细胞转染效率会随时间明显下降。 4.避免使用抗生素 Ambion公司推荐从细胞种植到转染后72小时期间避免使用抗生素。抗生素会在穿透的细胞中积累毒素。有些细胞和转染试剂在siRNA转染时需要无血清的条件。这种情况下,可同时用正常培养基和无血清培养基做对比实验,以得到最佳转染效果。5.选择合适的转染试剂 针对siRNA制备方法以及靶细胞类型的不同,选择好的转染试剂和优化的操作对siRNA实验的成功至关重要。6.通过合适的阳性对照优化转染和检测条件 对大多数细胞,看家基因是较好的阳性对照。将不同浓度的阳性对照的siRNA转入靶细胞(同样适合实验靶siRNA),转染48小时后统计对照蛋白或mRNA相对于未转染细胞的降低水平。过多的siRNA将导致细胞毒性以至死亡。7.通过标记siRNA来优化实验 荧光标记的siRNA能用来分析siRNA稳定性和转染效率。标记的siRNA还可用作siRNA胞内定位及双标记实验(配合标记抗体)来追踪转染过程中导入了siRNA的细胞,将转染与靶蛋白表达的下调结合起来。三、RNAi的应用前景1. 研究基因功能的新工具 已有研究表明RNAi能够在哺乳动物中灭活或降低特异性基因的表达,制作多种表型,而且抑制基因表达的时间可以随意控制在发育的任何阶段,产生类似基因敲除的效应。线虫和果蝇的全部基因组序列已测试完毕,发现大量未知功能的新基因,RNAi将大大促进对这些新基因功能的研究。与传统的基因敲除技术相比,这一技术具有投入少,周期短,操作简单等优势,近来RNAi成功用于构建转基因动物模型的报道日益增多,标志着RNAi将成为研究基因功能不可或缺的工具。2. 研究信号传导通路的新途径 联合利用传统的缺失突变技术和RNAi技术可以很容易地确定复杂的信号传导途径中不同基因的上下游关系,Clemensy等应用RNAi研究了果蝇细胞系中胰岛素信息传导途径,取得了与已知胰岛素信息传导通路完全一致的结果,在此基础上分析了DSH3PX1与DACK之间的关系, 证实了DACK是位于DSH3PX1磷酸化的上游激酶. RNAi技术较传统的转染实验简单、快速、重复性好,克服了转染实验中重组蛋白特异性聚集和转染效率不高的缺点, 因此认为RNAi技术将可能成为研究细胞信号传导通路的新途径。3.开展基因治疗的新策略 RNAi具有抵抗病毒入侵,抑制转座子活动,防止自私基因序列过量增殖等作用,因此可以利用RNAi现象产生抗病毒的植物和动物,并可利用不同病毒转录序列中高度同源区段相应的dsRNA抵抗多种病毒。 肿瘤是多个基因相互作用的基因网络调控的结果,传统技术诱发的单一癌基因的阻断不可能完全抑制或逆转肿瘤的生长, 而RNAi可以利用同一基因家族的多个基因具有一段同源性很高的保守序列这一特性, 设计针对这一区段序列的dsRNA分子,只注射一种dsRNA即可以产生多个基因同时剔除的表现,也可以同时注射多种dsRNA而将多个序列不相关的基因同时剔除。 尽管目前RNAi技术在哺乳动物中的应用还处于探索阶段,但它在斑马鱼和老鼠等脊椎动物中的成功应用预示着RNAi将成为基因治疗中重要的组成部分,人工合成的dsRNA寡聚药物的开发将可能成为极具发展前途的新兴产业。
RNAi技术在病毒性疾病和肿瘤治疗中的应用前景
摘要 RNA干扰是内源性或外源性双链RNA诱发的mRNA水平上的基因沉默机制,具有高度特异性。21 nt干扰性小RNA在哺乳动物细胞中可以诱导强有力的特异性RNAi作用,这一突破性研究进展开创了人类疾病治疗的新天地,RNAi 技术用于人类疾病治疗已受到极大关注,尤其在AIDS、乙肝和丙肝等病毒感染性疾病和肿瘤治疗中的应用研究最为活跃和深入,并取得了令人鼓舞的成果。本文对siRNA的设计、制备,及其在病毒性疾病和肿瘤治疗中的研究进展作一综述。 关键词:siRNA RNAi 肿瘤治疗 HIV 病毒性肝炎 RNAi (RNA interference,RNAi)是高度特异的在mRNA水平上的基因沉默机制,由内源性或外源性dsRNA(double-stranded RNA,dsRNA)诱发。dsRNA 在细胞内被切割成21-25nt 干扰性小RNA(small interfering RNA,siRNA), siRNA介导识别并靶向切割同源性靶mRNA分子,从而导致该基因不表达[1]。RNAi发现仅短短5年的时间,由于其高效性和高度特异性,RNAi 成为颇为理想的细胞水平基因敲除工具,并迅速成为后基因组时代功能基因组学研究中不可或缺的重要手段。然而,由于长的dsRNA(38-1000bp)在哺乳动物细胞中产生非特异的基因表达抑制,使得RNAi 技术用于哺乳动物中的研究相对滞后。直至2001年,Tuschl研究组通过采取21 nt siRNA 在哺乳动物细胞中诱导了强有力的特异性RNA干扰作用[3],这一突破性研究进展开创了人类疾病治疗的新天地,在AIDS、乙肝和丙肝等病毒感染性疾病和肿瘤治疗中的应用研究最为活跃和深入,并取得了令人鼓舞的成果。本文对siRNA的设计、制备,以及在病毒性疾病和肿瘤治疗中的研究进展做一综述。 siRNA的设计、制备 在哺乳动物细胞中引发特异的基因沉默最有效的siRNA是21 nt siRNA[3],其中19个碱基互补,两边3΄ 端各有2个碱基突出(通常是AA或UU)。基因抑制的有效性关键在于siRNA靶基因序列片段的选择,然而,目前为止人们并不知道mRNA的哪一个部位对siRNA更敏感。通常随机选择3-4个靶位点进行设计siRNA,然后根据体外实验结果,筛选出最为有效的siRNA。考虑到mRNA 5΄ 端非编码区(untranslated regions,UTRs)通常有丰富的调控蛋白结合位点,调控蛋白与目的mRNA UTR区结合后可能会影响RISC复合物(RNA-induced silencing complex, RISC)与mRNA结合降低抑制效果,通常人们避开此区域设计siRNA,但也有研究者持相反态度。 选出合适的靶序列后要同Genebank中的编码序列进行blast 序列分析是非常必要的,这可以避免与无关的mRNA序列同源[4]。目前有多种在线siRNA设计软件可供使用,详细情况请搜索相关网站(www.dharmacon.com, www.qiagen.com, www.ambion.com, )。 化学合成法制备siRNA是目前最常用的方法,Dharmacon、QIAGEN 、Ambion等公司均可以提供高质量的化学合成siRNA,但费用昂贵。其他体外制备siRNA的方法还包括体外转录法和Rnase III(如Silencer siRNA Construction Kit,Ambion公司) 或Dicer 酶消化dsRNA法[5]。体外制备的siRNA需要专门的RNA转染试剂将其转到细胞内,在不同细胞内的转染效率存在较大差异,在原代细胞和T淋巴细胞中的转染效率低,转染到细胞内后作用时间较短,不能持久抑制基因表达。针对以上不足,结合过去20多年基因治疗的经验,人们构建了各种siRNA 表达载体或病毒载体,siRNA通过转染到细胞内的DNA模板转录而获得,在细胞内形成发卡结构的小RNA(short hairpin RNA, shRNA)。siRNA病毒载体的应用研究是目前研究的热点。目前RNAi常用的病毒载体包括逆转录病毒载体、慢病毒载体和腺病毒载体[6,7]。 RNAi 在抗病毒感染中的应用前景 1. RNAi 技术用于抗HIV治疗 2001年Tuschl研究组的突破性研究开创了RNAi技术治疗人类疾病的新途径,siRNA迅速成为治疗HIV的新武器。美国费城的Thomas Jefferson 大學生化及分子药理研究所Roger J. Pomerantz博士指出:将这项新颖的技术应用于人类疾病的治疗上可能有莫大的潜力。在AIDS的治疗上,RNA干扰可能成为对抗HIV病毒感染、复制的最有利方式。人们针对HIV生活周期的不同阶段设计了多种siRNA,针对HIV病毒gag、tat、rev、nef等基因的siRNA可用以控制病毒复制,针对宿主细胞的表面HIV受体基因的siRNA可用以控制病毒进入宿主细胞内,抑制病毒的感染过程[8]。 1.1 针对HIV基因组RNA的siRNA Jacque等针对HIV病毒基因组的长末端重复序列(long terminal repeat ,LTR)、病毒vif和nef基因,设计合成了siRNA。他们将siRNA和HIV前病毒DNA共同转染CD4受体阳性的Hela细胞,病毒感染阳性细胞的比率同对照组相比降低了95%以上。诺贝尔生理医学奖得主Philip Sharp博士研究组设计合成了针对HIV gag 基因的anti-gag siRNA。研究证实针对病毒基因組的gag区域的siRNA可以有效的將病毒的基因“沉默”,抑制HIV病毒的复制能力[9]。Bauer 研究组通过载体在细胞内表达anti-rev siRNA,可明显降低HIV复制水平,作用持续时间达数天之久[10]。在体外细胞株培养条件下,人们利用RNAi技术成功的控制HIV在细胞内的复制能力,然而,将该项技术用于临床治疗尚有一定距离。首先,siRNA转染到原代T淋巴细胞比较困难是anti-HIV siRNA在临床应用上的一大障碍。其次,RNAi具有高度特异性,靶序列上一个碱基的差异即可导致抑制效果明显不同,而HIV逆转录酶的错配率较高(每个复制周期可达1/1000nt),可能会迅速产生病毒突变株逃避siRNA的抑制作用。再者,不同个体甚至同一个体内的HIV病毒基因组呈现序列复杂多样性,给siRNA的设计带来困难。 1.2 针对宿主细胞HIV受体的siRNA 由于HIV病毒基因组的复杂多样性和易突变性,单独针对某一病毒基因的siRNA可能无法达到令人满意的抑制病毒复制的效果,采取针对宿主细胞HIV受体的siRNA以阻止病毒进入细胞内也许更具实用价值。CD4是HIV病毒与之结合并进入宿主细胞内的主要受体,Philip Sharp博士研究组发现,针对宿主细胞受体CD4的anti- CD4 siRNA可以有效抑制HIV病毒的感染能力,并进而影响其复制水平[10]。然而,CD4是人体正常免疫功能不可缺少的分子,它的表达抑制势必影响细胞免疫功能。在以往的研究中发现,T细胞共同受体CCR5的突变可以有效的保护细胞免受HIV-1的攻击,而不影响正常的免疫功能。由此可以设想针对CCR5、CCR4等共同受体的siRNA或许具有更大的意义。此外,我们可以针对HIV感染和复制的不同阶段设计多种siRNA,多种siRNA联合应用很可能增强病毒抑制效果,也可以同其他抗HIV的药物合用。 2.RNAi技术用于治疗其他病毒性疾病的研究 RNAi技术除用于抗HIV研究以外,还用于抗其他多种病毒的探索。它们包括乙型肝炎病毒(HBV)、丙型肝炎病毒(HCV)、呼吸道合胞病毒(RSV)、流感病毒(influenza virus)、脊髓灰质炎病毒(poliovirus)等[11,12]。以上研究均取得了另人欣喜的体外病毒抑制作用,但体内清除病毒的效果尚须在动物实验模型中进一步验证。RNAi技术可能会给病毒治疗带来一场革命,但真正用于抗病毒治疗还有待进一步验证。 RNAi 在抗肿瘤治疗中的应用前景 1.RNAi用于抑制癌基因表达 原癌基因是细胞基因组中的正常组成成分,在自然状态下无致癌作用,主要作用是通过编码生长因子、受体和细胞内信息传递物质调节正常细胞的生长、分裂和分化。原癌基因的活化主要包括点突变、插入突变、基因扩增和染色体重排。以上各种类型的活化的癌基因的mRNA均可以采取RNAi 技术得到抑制,从而抑制肿瘤的生长。 1.1 RNAi 技术用于敲除点突变激活的癌基因 RNAi 技术是敲除点突变激活的癌基因的理想武器。K- ras 基因成为第一个采用RNAi 技术敲除的癌基因。ras基因是目前已知在人类肿瘤中最普遍呈活化状态的致癌基因,此基因的突变現象存在于30-50%的肿瘤组织中,在胰腺癌可达85%。突变的ras 基因与正常细胞中的野生型ras 基因通常只有一个碱基的不同,但由于RNAi 的高度特异性,可以针对肿瘤细胞中突变的ras产生抑制作用,而对正常细胞中的野生型ras不产生抑制作用。Agami研究组采用逆转录病毒载体shRNA表达系统不仅特异性地在体外抑制了CAPAN-1胰腺癌细胞内的K- ras 基因的表达,也成功的抑制了裸鼠移植瘤的生长[13]。 1.2 RNAi 技术用于抑制基因扩增 基因扩增是指原癌基因通过某种机制在原来染色体上复制出多个拷贝数,导致表达产物异常增多,细胞增殖。EGFR(erbB1)在乳腺癌和肺癌等多种上皮性肿瘤中发生基因扩增,在肿瘤发生中起重要作用。德国Jovin研究组通过化学合成的anti-erbB1 siRNA成功的抑制了A431细胞内erbB1的表达,导致细胞凋亡增加和肿瘤增殖减弱[14]。 1.3 RNAi 技术用于抑制融合基因表达 染色体重排是指癌基因从所在染色体的正常位置上易位至另一个染色体的某一位置上,使起调控发生改变转为激活状态,产生异常基因产物,导致细胞恶性增殖。BCR/ABL融合基因是定位于人9号染色体9q34上的C-ABL基因和22号染色体22q11 上的BCR基因发生t(9:22)易位,使相应的无关的基因发生融合而形成的,它是Ph 染色体的分子基础,在慢性粒细胞性白血病(CML)发病中起到重要作用。Borkhardt等采用RNAi 技术成功的抑制了K562白血病细胞中的BCR/ABL融合基因mRNA的表达[15]。 2.RNAi用于抑制其他与肿瘤发生发展相关基因的表达 肿瘤发生发展过程中除癌基因激活外,还涉及到多种形式的基因改变。bcl-2 是与血液系统恶性肿瘤密切相关的抗凋亡基因,可以阻遏化疗药物诱导的肿瘤细胞的凋亡。raf-1和白血病的发生和耐药性也有密切联系。抑制bcl-2 和raf-1 mRNA的表达可以抑制白血病细胞的增殖[[16]。抑制肿瘤血管的生成是肿瘤治疗的又一策略,anti- VEGF siRNA可以特异性的抑制血管内皮生长因子(vascular endothelial growth factor,VEGF)的表达,抑制肿瘤的生长[17]。肿瘤的耐药性和多药耐药基因(multidrug resistance,MDR)的表达有关,通过anti- MDR siRNA可以抑制细胞的耐药产生[18]。 如何在体内实现靶基因siRNA转移到所有肿瘤细胞并稳定持久的抑制癌基的表达是RNAi技术治疗肿瘤的关键。此外,从目前RNAi的研究现状来看,在哺乳动物细胞中,RNAi并不能完全阻断基因的表达,尤其是异常高表达的基因[3], 这也将影响对肿瘤的治疗疗效。尽管如此,但RNAi的高效特异性抑制癌基因表达以及明显优于反义核酸的效果的特征显示: RNAi将是一项令人不可忽视的抗肿瘤新技术。 2002年12月20日, Small RNA & RNAi 被Science杂志评为年度十大科技成就之首,Nature杂志亦将Small RNA评为年度重大科技成果之一。RNA研究的突破性进展,是生物医学领域近20年来,可与人类基因组计划(human genomics program,HGP)相提并论的最重大成果之一。RNAi最主要的功能在于可以调节和关闭基因的表达,进而调控细胞的各种高级生命活动。人们对小RNA分子的深入研究必将大大推进基因功能的研究,更为各种病毒性疾病和肿瘤等疾病的根治,开辟新的治疗途径。Small RNA与RNAi的研究与应用,具有极其重要的理论和实际意义,它必将对生物学和医学的发展产生深远的影响。
近年来的研究表明,将与mRNA对应的正义RNA和反义RNA组成的双链RNA(dsRNA)导入细胞,可以使mRNA发生特异性的降解,导致其相应的基因沉默。这种转录后基因沉默机制(post-transcriptional gene silencing, PTGS)被称为RNA干扰(RNAi)。 一、RNAi的分子机制 通过生化和遗传学研究表明,RNA干扰包括起始阶段和效应阶段(inititation and effector steps)。在起始阶段,加入的小分子RNA被切割为21-23核苷酸长的小分子干扰RNA片段(small interfering RNAs, siRNAs)。证据表明;一个称为Dicer的酶,是RNase III家族中特异识别双链RNA的一员,它能以一种ATP依赖的方式逐步切割由外源导入或者由转基因,病毒感染等各种方式引入的双链RNA,切割将RNA降解为19-21bp的双链RNAs(siRNAs),每个片段的3’端都有2个碱基突出。 在RNAi效应阶段,siRNA双链结合一个核酶复合物从而形成所谓RNA诱导沉默复合物(RNA-induced silencing complex, RISC)。激活RISC需要一个ATP依赖的将小分子RNA解双链的过程。激活的RISC通过碱基配对定位到同源mRNA转录本上,并在距离siRNA3’端12个碱基的位置切割mRNA。尽管切割的确切机制尚不明了,但每个RISC都包含一个siRNA和一个不同于Dicer的RNA酶。 另外,还有研究证明含有启动子区的dsRNA在植物体内同样被切割成21-23nt长的片段,这种dsRNA可使内源相应的DNA序列甲基化,从而使启动子失去功能,使其下游基因沉默.二、如何进行RNAi试验(一)siRNA的设计1. 在设计RNAi实验时,可以先在以下网站进行目标序列的筛选:http://www.genesil.com/business/products/order2.htmhttp://www.ambion.com/techlib/misc/siRNA_finder.htmlhttp://www.ic.sunysb.edu/Stu/shilin/rnai.htmlhttp://design.dharmacon.com/rnadesign/default.aspx?SID=453587102.RNAi目标序列的选取原则:(1)从转录本(mRNA)的AUG起始密码开始,寻找“AA”二连序列,并记下其3'端的19个碱基序列,作为潜在的siRNA靶位点。有研究结果显示GC含量在45%—55%左右的siRNA要比那些GC含量偏高的更为有效。Tuschl等建议在设计siRNA时不要针对5'和3'端的非编码区(untranslated regions,UTRs),原因是这些地方有丰富的调控蛋白结合区域,而这些UTR结合蛋白或者翻译起始复合物可能会影响siRNP核酸内切酶复合物结合mRNA从而影响siRNA的效果。(2)将潜在的序列和相应的基因组数据库(人,或者小鼠,大鼠等等)进行比较,排除那些和其他编码序列/EST同源的序列。例如使用BLAST( www.ncbi.nlm.nih.gov/BLAST/)(3)选出合适的目标序列进行合成。通常一个基因需要设计多个靶序列的siRNA,以找到最有效的siRNA序列。3.阴性对照 一个完整的siRNA实验应该有阴性对照,作为阴性对照的siRNA应该和选中的siRNA序列有相同的组成,但是和mRNA没有明显的同源性。通常的做法是将选中的siRNA序列打乱,同样要检查结果以保证它和目的靶细胞中其他基因没有同源性。4.目前已证实的siRNA可以在下面的网页找到:http://design.dharmacon.com/catalog/category.aspx?key=49http://www.ambion.com/techlib/tb/tb_502.htmlhttp://web.mit.edu/mmcmanus/www/siRNADB.htmlhttp://python.penguindreams.net/Order_Entry/jsp/BrowseCatalog.jsp?Category=Published(二)siRNA的制备 目前为止较为常用的方法有通过化学合成,体外转录,长片断dsRNAs经RNase III 类降解 (e.g. Dicer, E. coli, RNase III)体外制备siRNA,以及通过siRNA表达载体或者病毒载体,PCR制备的siRNA表达框在细胞中表达产生siRNA。体外制备1.化学合成 许多国外公司都可以根据用户要求提供高质量的化学合成siRNA。主要的缺点包括价格高,定制周期长,特别是有特殊需求的。由于价格比其他方法高,为一个基因合成3—4对siRNAs 的成本就更高了,比较常见的做法是用其他方法筛选出最有效的序列再进行化学合成。最适用于:已经找到最有效的siRNA的情况下,需要大量siRNA进行研究不适用于:筛选siRNA等长时间的研究,主要原因是价格因素2.体外转录 以DNA Oligo为模版,通过体外转录合成siRNAs,成本相对化学合成法而言比较低,而且能够比化学合成法更快的得到siRNAs。不足之处是实验的规模受到限制,虽然一次体外转录合成能提供足够做数百次转染的siRNAs,但是反应规模和量始终有一定的限制。而且和化学合成相比,还是需要占用研究人员相当的时间。值得一提的是体外转录得到的siRNAs毒性小,稳定性好,效率高,只需要化学合成的siRNA量的1/10就可以达到化学合成siRNA所能达到的效果,从而使转染效率更高。最适用于:筛选siRNAs,特别是需要制备多种siRNAs,化学合成的价格成为障碍时。不适用于:实验需要大量的,一个特定的siRNA。长期研究。3.用RNase III 消化长片断双链RNA制备siRNA 其他制备siRNA的方法的缺陷是需要设计和检验多个siRNA序列以便找到一个有效的siRNA。而用这种方法——制备一份混合有各种siRNAs “混合鸡尾酒” 就可以避免这个缺陷。选择通常是200—1000碱基的靶mRNA模版,用体外转录的方法制备长片断双链dsRNA ,然后用RNase III (or Dicer) 在体外消化,得到一种siRNAs“混合鸡尾酒”。在除掉没有被消化的dsRNA后,这个siRNA混合物就可以直接转染细胞,方法和单一的siRNA转染一样。由于siRNA混合物中有许多不同的siRNAs,通常能够保证目的基因被有效地抑制。 dsRNA消化法的主要优点在于可以跳过检测和筛选有效siRNA序列的步骤,为研究人员节省时间和金钱(注意:通常用RNAse III通常比用Dicer要便宜)。不过这种方法的缺点也很明显,就是有可能引发非特异的基因沉默,特别是同源或者是密切相关的基因。现在多数的研究显示这种情况通常不会造成影响。最适用于:快速而经济地研究某个基因功能缺失的表型不适用于:长时间的研究项目,或者是需要一个特定的siRNA进行研究,特别是基因治疗体内表达 前面的3种方法主要都是体外制备siRNAs,并且需要专门的RNA转染试剂将siRNAs转到细胞内。而采用siRNA表达载体和基于PCR的表达框架则属于:从转染到细胞的DNA模版中在体内转录得到siRNAs。这两种方法的优点在于不需要直接操作RNA。4. siRNA表达载体 多数的siRNA表达载体依赖三种RNA聚合酶III 启动子(pol III)中的一种,操纵一段小的发夹RNA(short hairpin RNA, shRNA)在哺乳动物细胞中的表达。这三类启动子包括大家熟悉的人源和鼠源的U6启动子和人H1启动子。之所以采用RNA pol III启动子是由于它可以在哺乳动物细胞中表达更多的小分子RNA,而且它是通过添加一串(3到6个)U来终止转录的。要使用这类载体,需要订购2段编码短发夹RNA序列的DNA单链,退火,克隆到相应载体的pol III 启动子下游。由于涉及到克隆,这个过程需要几周甚至数月的时间,同时也需要经过测序以保证克隆的序列是正确的。 siRNA表达载体的优点在于可以进行较长期研究——带有抗生素标记的载体可以在细胞中持续抑制靶基因的表达,持续数星期甚至更久。病毒载体也可用于siRNA表达,其优势在于可以直接高效率感染细胞进行基因沉默的研究,避免由于质粒转染效率低而带来的种种不便,而且转染效果更加稳定。最适用于:已知一个有效的siRNA序列,需要维持较长时间的基因沉默。不适用于:筛选siRNA序列(其实主要是指需要多个克隆和测序等较为费时、繁琐的工作)。5. siRNA表达框架 siRNA表达框架(siRNA expression cassettes,SECs)是一种由PCR得到的siRNA表达模版,包括一个RNA pol III启动子,一段发夹结构siRNA,一个RNA pol III终止位点,能够直接导入细胞进行表达而无需事前克隆到载体中。和siRNA表达载体不同的是,SECs不需要载体克隆、测序等颇为费时的步骤,可以直接由PCR得到,不用一天的时间。因此,SECs成为筛选siRNA的最有效工具,甚至可以用来筛选在特定的研究体系中启动子和siRNA的最适搭配。如果在PCR两端添加酶切位点,那么通过SECs筛选出的最有效的siRNA后,可以直接克隆到载体中构建siRNA表达载体。构建好的载体可以用于稳定表达siRNA和长效抑制的研究。这个方法的主要缺点是①PCR产物很难转染到细胞中②不能进行序列测定,PCR和DNA合成时可能差生的误读不能被发现导致结果不理想。最适用于:筛选siRNA序列,在克隆到载体前筛选最佳启动子不适用于:长期抑制研究。(如果克隆到载体后就可以了)(三)siRNA的转染 将制备好的siRNA,siRNA表达载体或表达框架转导至真核细胞中的方法主要有以下几种:1.磷酸钙共沉淀 将氯化钙,RNA(或DNA)和磷酸缓冲液混合,沉淀形成包含DNA且极小的不溶的磷酸钙颗粒。磷酸钙-DNA复合物粘附到细胞膜并通过胞饮进入目的细胞的细胞质。沉淀物的大小和质量对于磷酸钙转染的成功至关重要。在实验中使用的每种试剂都必须小心校准,保证质量,因为甚至偏离最优条件十分之一个pH都会导致磷酸钙转染的失败。2.电穿孔法 电穿孔通过将细胞暴露在短暂的高场强电脉冲中转导分子。将细胞悬浮液置于电场中会诱导沿细胞膜的电压差异,据认为这种电压差异会导致细胞膜暂时穿孔。电脉冲和场强的优化对于成功的转染非常重要,因为过高的场强和过长的电脉冲时间会不可逆地伤害细胞膜而裂解细胞。一般,成功的电穿孔过程都伴随高水平(50%或更高)的毒性。3.DEAE-葡聚糖和polybrene 带正电的DEAE-葡聚糖或polybrene多聚体复合物和带负电的DNA分子使得DNA可以结合在细胞表面。通过使用DMSO或甘油获得的渗透休克将DNA复合体导入。两种试剂都已成功用于转染。DEAE-葡聚糖仅限于瞬时转染。4.机械法 转染技术也包括使用机械的方法,比如显微注射和基因枪(biolistic particle)。显微注射使用一根细针头将DNA,RNA或蛋白直接转入细胞质或细胞核。基因枪使用高压microprojectile将大分子导入细胞。5.阳离子脂质体试剂 在优化条件下将阳离子脂质体试剂加入水中时,其可以形成微小的(平均大小约100-400nm)单层脂质体。这些脂质体带正电,可以靠静电作用结合到DNA的磷酸骨架上以及带负电的细胞膜表面。因此使用阳离子脂质体转染的原理与以前利用中性脂质体转染的原理不同。使用阳离子脂质体试剂,DNA并没有预先包埋在脂质体中,而是带负电的DNA自动结合到带正电的脂质体上,形成DNA-阳离子脂质体复合物。据称,一个约5kb的质粒会结合2-4个脂质体。被俘获的DNA就会被导入培养的细胞。现存对DNA转导原理的证据来源于内吞体和溶酶体。 为了达到高的转染效率,在转染实验过程中,需要注意以下几点:1.纯化siRNA 在转染前要确认siRNA的大小和纯度。为得到高纯度的siRNA,推荐用玻璃纤维结合,洗脱或通过15-20%丙烯酰胺胶除去反应中多余的核苷酸,小的寡核苷酸,蛋白和盐离子。注意:化学合成的RNA通常需要跑胶电泳纯化(即PAGE胶纯化)。 2.避免RNA酶污染 微量的RNA酶将导致siRNA实验失败。由于实验环境中RNA酶普遍存在,如皮肤,头发,所有徒手接触过的物品或暴露在空气中的物品等,此保证实验每个步骤不受RNA酶污染非常重要。3.健康的细胞培养物和严格的操作确保转染的重复性 通常,健康的细胞转染效率较高。此外,较低的传代数能确保每次实验所用细胞的稳定性。为了优化实验,推荐用50代以下的转染细胞,否则细胞转染效率会随时间明显下降。 4.避免使用抗生素 Ambion公司推荐从细胞种植到转染后72小时期间避免使用抗生素。抗生素会在穿透的细胞中积累毒素。有些细胞和转染试剂在siRNA转染时需要无血清的条件。这种情况下,可同时用正常培养基和无血清培养基做对比实验,以得到最佳转染效果。5.选择合适的转染试剂 针对siRNA制备方法以及靶细胞类型的不同,选择好的转染试剂和优化的操作对siRNA实验的成功至关重要。6.通过合适的阳性对照优化转染和检测条件 对大多数细胞,看家基因是较好的阳性对照。将不同浓度的阳性对照的siRNA转入靶细胞(同样适合实验靶siRNA),转染48小时后统计对照蛋白或mRNA相对于未转染细胞的降低水平。过多的siRNA将导致细胞毒性以至死亡。7.通过标记siRNA来优化实验 荧光标记的siRNA能用来分析siRNA稳定性和转染效率。标记的siRNA还可用作siRNA胞内定位及双标记实验(配合标记抗体)来追踪转染过程中导入了siRNA的细胞,将转染与靶蛋白表达的下调结合起来。三、RNAi的应用前景1. 研究基因功能的新工具 已有研究表明RNAi能够在哺乳动物中灭活或降低特异性基因的表达,制作多种表型,而且抑制基因表达的时间可以随意控制在发育的任何阶段,产生类似基因敲除的效应。线虫和果蝇的全部基因组序列已测试完毕,发现大量未知功能的新基因,RNAi将大大促进对这些新基因功能的研究。与传统的基因敲除技术相比,这一技术具有投入少,周期短,操作简单等优势,近来RNAi成功用于构建转基因动物模型的报道日益增多,标志着RNAi将成为研究基因功能不可或缺的工具。2. 研究信号传导通路的新途径 联合利用传统的缺失突变技术和RNAi技术可以很容易地确定复杂的信号传导途径中不同基因的上下游关系,Clemensy等应用RNAi研究了果蝇细胞系中胰岛素信息传导途径,取得了与已知胰岛素信息传导通路完全一致的结果,在此基础上分析了DSH3PX1与DACK之间的关系, 证实了DACK是位于DSH3PX1磷酸化的上游激酶. RNAi技术较传统的转染实验简单、快速、重复性好,克服了转染实验中重组蛋白特异性聚集和转染效率不高的缺点, 因此认为RNAi技术将可能成为研究细胞信号传导通路的新途径。3.开展基因治疗的新策略 RNAi具有抵抗病毒入侵,抑制转座子活动,防止自私基因序列过量增殖等作用,因此可以利用RNAi现象产生抗病毒的植物和动物,并可利用不同病毒转录序列中高度同源区段相应的dsRNA抵抗多种病毒。 肿瘤是多个基因相互作用的基因网络调控的结果,传统技术诱发的单一癌基因的阻断不可能完全抑制或逆转肿瘤的生长, 而RNAi可以利用同一基因家族的多个基因具有一段同源性很高的保守序列这一特性, 设计针对这一区段序列的dsRNA分子,只注射一种dsRNA即可以产生多个基因同时剔除的表现,也可以同时注射多种dsRNA而将多个序列不相关的基因同时剔除。 尽管目前RNAi技术在哺乳动物中的应用还处于探索阶段,但它在斑马鱼和老鼠等脊椎动物中的成功应用预示着RNAi将成为基因治疗中重要的组成部分,人工合成的dsRNA寡聚药物的开发将可能成为极具发展前途的新兴产业。
RNAi技术在病毒性疾病和肿瘤治疗中的应用前景
摘要 RNA干扰是内源性或外源性双链RNA诱发的mRNA水平上的基因沉默机制,具有高度特异性。21 nt干扰性小RNA在哺乳动物细胞中可以诱导强有力的特异性RNAi作用,这一突破性研究进展开创了人类疾病治疗的新天地,RNAi 技术用于人类疾病治疗已受到极大关注,尤其在AIDS、乙肝和丙肝等病毒感染性疾病和肿瘤治疗中的应用研究最为活跃和深入,并取得了令人鼓舞的成果。本文对siRNA的设计、制备,及其在病毒性疾病和肿瘤治疗中的研究进展作一综述。 关键词:siRNA RNAi 肿瘤治疗 HIV 病毒性肝炎 RNAi (RNA interference,RNAi)是高度特异的在mRNA水平上的基因沉默机制,由内源性或外源性dsRNA(double-stranded RNA,dsRNA)诱发。dsRNA 在细胞内被切割成21-25nt 干扰性小RNA(small interfering RNA,siRNA), siRNA介导识别并靶向切割同源性靶mRNA分子,从而导致该基因不表达[1]。RNAi发现仅短短5年的时间,由于其高效性和高度特异性,RNAi 成为颇为理想的细胞水平基因敲除工具,并迅速成为后基因组时代功能基因组学研究中不可或缺的重要手段。然而,由于长的dsRNA(38-1000bp)在哺乳动物细胞中产生非特异的基因表达抑制,使得RNAi 技术用于哺乳动物中的研究相对滞后。直至2001年,Tuschl研究组通过采取21 nt siRNA 在哺乳动物细胞中诱导了强有力的特异性RNA干扰作用[3],这一突破性研究进展开创了人类疾病治疗的新天地,在AIDS、乙肝和丙肝等病毒感染性疾病和肿瘤治疗中的应用研究最为活跃和深入,并取得了令人鼓舞的成果。本文对siRNA的设计、制备,以及在病毒性疾病和肿瘤治疗中的研究进展做一综述。 siRNA的设计、制备 在哺乳动物细胞中引发特异的基因沉默最有效的siRNA是21 nt siRNA[3],其中19个碱基互补,两边3΄ 端各有2个碱基突出(通常是AA或UU)。基因抑制的有效性关键在于siRNA靶基因序列片段的选择,然而,目前为止人们并不知道mRNA的哪一个部位对siRNA更敏感。通常随机选择3-4个靶位点进行设计siRNA,然后根据体外实验结果,筛选出最为有效的siRNA。考虑到mRNA 5΄ 端非编码区(untranslated regions,UTRs)通常有丰富的调控蛋白结合位点,调控蛋白与目的mRNA UTR区结合后可能会影响RISC复合物(RNA-induced silencing complex, RISC)与mRNA结合降低抑制效果,通常人们避开此区域设计siRNA,但也有研究者持相反态度。 选出合适的靶序列后要同Genebank中的编码序列进行blast 序列分析是非常必要的,这可以避免与无关的mRNA序列同源[4]。目前有多种在线siRNA设计软件可供使用,详细情况请搜索相关网站(www.dharmacon.com, www.qiagen.com, www.ambion.com, )。 化学合成法制备siRNA是目前最常用的方法,Dharmacon、QIAGEN 、Ambion等公司均可以提供高质量的化学合成siRNA,但费用昂贵。其他体外制备siRNA的方法还包括体外转录法和Rnase III(如Silencer siRNA Construction Kit,Ambion公司) 或Dicer 酶消化dsRNA法[5]。体外制备的siRNA需要专门的RNA转染试剂将其转到细胞内,在不同细胞内的转染效率存在较大差异,在原代细胞和T淋巴细胞中的转染效率低,转染到细胞内后作用时间较短,不能持久抑制基因表达。针对以上不足,结合过去20多年基因治疗的经验,人们构建了各种siRNA 表达载体或病毒载体,siRNA通过转染到细胞内的DNA模板转录而获得,在细胞内形成发卡结构的小RNA(short hairpin RNA, shRNA)。siRNA病毒载体的应用研究是目前研究的热点。目前RNAi常用的病毒载体包括逆转录病毒载体、慢病毒载体和腺病毒载体[6,7]。 RNAi 在抗病毒感染中的应用前景 1. RNAi 技术用于抗HIV治疗 2001年Tuschl研究组的突破性研究开创了RNAi技术治疗人类疾病的新途径,siRNA迅速成为治疗HIV的新武器。美国费城的Thomas Jefferson 大學生化及分子药理研究所Roger J. Pomerantz博士指出:将这项新颖的技术应用于人类疾病的治疗上可能有莫大的潜力。在AIDS的治疗上,RNA干扰可能成为对抗HIV病毒感染、复制的最有利方式。人们针对HIV生活周期的不同阶段设计了多种siRNA,针对HIV病毒gag、tat、rev、nef等基因的siRNA可用以控制病毒复制,针对宿主细胞的表面HIV受体基因的siRNA可用以控制病毒进入宿主细胞内,抑制病毒的感染过程[8]。 1.1 针对HIV基因组RNA的siRNA Jacque等针对HIV病毒基因组的长末端重复序列(long terminal repeat ,LTR)、病毒vif和nef基因,设计合成了siRNA。他们将siRNA和HIV前病毒DNA共同转染CD4受体阳性的Hela细胞,病毒感染阳性细胞的比率同对照组相比降低了95%以上。诺贝尔生理医学奖得主Philip Sharp博士研究组设计合成了针对HIV gag 基因的anti-gag siRNA。研究证实针对病毒基因組的gag区域的siRNA可以有效的將病毒的基因“沉默”,抑制HIV病毒的复制能力[9]。Bauer 研究组通过载体在细胞内表达anti-rev siRNA,可明显降低HIV复制水平,作用持续时间达数天之久[10]。在体外细胞株培养条件下,人们利用RNAi技术成功的控制HIV在细胞内的复制能力,然而,将该项技术用于临床治疗尚有一定距离。首先,siRNA转染到原代T淋巴细胞比较困难是anti-HIV siRNA在临床应用上的一大障碍。其次,RNAi具有高度特异性,靶序列上一个碱基的差异即可导致抑制效果明显不同,而HIV逆转录酶的错配率较高(每个复制周期可达1/1000nt),可能会迅速产生病毒突变株逃避siRNA的抑制作用。再者,不同个体甚至同一个体内的HIV病毒基因组呈现序列复杂多样性,给siRNA的设计带来困难。 1.2 针对宿主细胞HIV受体的siRNA 由于HIV病毒基因组的复杂多样性和易突变性,单独针对某一病毒基因的siRNA可能无法达到令人满意的抑制病毒复制的效果,采取针对宿主细胞HIV受体的siRNA以阻止病毒进入细胞内也许更具实用价值。CD4是HIV病毒与之结合并进入宿主细胞内的主要受体,Philip Sharp博士研究组发现,针对宿主细胞受体CD4的anti- CD4 siRNA可以有效抑制HIV病毒的感染能力,并进而影响其复制水平[10]。然而,CD4是人体正常免疫功能不可缺少的分子,它的表达抑制势必影响细胞免疫功能。在以往的研究中发现,T细胞共同受体CCR5的突变可以有效的保护细胞免受HIV-1的攻击,而不影响正常的免疫功能。由此可以设想针对CCR5、CCR4等共同受体的siRNA或许具有更大的意义。此外,我们可以针对HIV感染和复制的不同阶段设计多种siRNA,多种siRNA联合应用很可能增强病毒抑制效果,也可以同其他抗HIV的药物合用。 2.RNAi技术用于治疗其他病毒性疾病的研究 RNAi技术除用于抗HIV研究以外,还用于抗其他多种病毒的探索。它们包括乙型肝炎病毒(HBV)、丙型肝炎病毒(HCV)、呼吸道合胞病毒(RSV)、流感病毒(influenza virus)、脊髓灰质炎病毒(poliovirus)等[11,12]。以上研究均取得了另人欣喜的体外病毒抑制作用,但体内清除病毒的效果尚须在动物实验模型中进一步验证。RNAi技术可能会给病毒治疗带来一场革命,但真正用于抗病毒治疗还有待进一步验证。 RNAi 在抗肿瘤治疗中的应用前景 1.RNAi用于抑制癌基因表达 原癌基因是细胞基因组中的正常组成成分,在自然状态下无致癌作用,主要作用是通过编码生长因子、受体和细胞内信息传递物质调节正常细胞的生长、分裂和分化。原癌基因的活化主要包括点突变、插入突变、基因扩增和染色体重排。以上各种类型的活化的癌基因的mRNA均可以采取RNAi 技术得到抑制,从而抑制肿瘤的生长。 1.1 RNAi 技术用于敲除点突变激活的癌基因 RNAi 技术是敲除点突变激活的癌基因的理想武器。K- ras 基因成为第一个采用RNAi 技术敲除的癌基因。ras基因是目前已知在人类肿瘤中最普遍呈活化状态的致癌基因,此基因的突变現象存在于30-50%的肿瘤组织中,在胰腺癌可达85%。突变的ras 基因与正常细胞中的野生型ras 基因通常只有一个碱基的不同,但由于RNAi 的高度特异性,可以针对肿瘤细胞中突变的ras产生抑制作用,而对正常细胞中的野生型ras不产生抑制作用。Agami研究组采用逆转录病毒载体shRNA表达系统不仅特异性地在体外抑制了CAPAN-1胰腺癌细胞内的K- ras 基因的表达,也成功的抑制了裸鼠移植瘤的生长[13]。 1.2 RNAi 技术用于抑制基因扩增 基因扩增是指原癌基因通过某种机制在原来染色体上复制出多个拷贝数,导致表达产物异常增多,细胞增殖。EGFR(erbB1)在乳腺癌和肺癌等多种上皮性肿瘤中发生基因扩增,在肿瘤发生中起重要作用。德国Jovin研究组通过化学合成的anti-erbB1 siRNA成功的抑制了A431细胞内erbB1的表达,导致细胞凋亡增加和肿瘤增殖减弱[14]。 1.3 RNAi 技术用于抑制融合基因表达 染色体重排是指癌基因从所在染色体的正常位置上易位至另一个染色体的某一位置上,使起调控发生改变转为激活状态,产生异常基因产物,导致细胞恶性增殖。BCR/ABL融合基因是定位于人9号染色体9q34上的C-ABL基因和22号染色体22q11 上的BCR基因发生t(9:22)易位,使相应的无关的基因发生融合而形成的,它是Ph 染色体的分子基础,在慢性粒细胞性白血病(CML)发病中起到重要作用。Borkhardt等采用RNAi 技术成功的抑制了K562白血病细胞中的BCR/ABL融合基因mRNA的表达[15]。 2.RNAi用于抑制其他与肿瘤发生发展相关基因的表达 肿瘤发生发展过程中除癌基因激活外,还涉及到多种形式的基因改变。bcl-2 是与血液系统恶性肿瘤密切相关的抗凋亡基因,可以阻遏化疗药物诱导的肿瘤细胞的凋亡。raf-1和白血病的发生和耐药性也有密切联系。抑制bcl-2 和raf-1 mRNA的表达可以抑制白血病细胞的增殖[[16]。抑制肿瘤血管的生成是肿瘤治疗的又一策略,anti- VEGF siRNA可以特异性的抑制血管内皮生长因子(vascular endothelial growth factor,VEGF)的表达,抑制肿瘤的生长[17]。肿瘤的耐药性和多药耐药基因(multidrug resistance,MDR)的表达有关,通过anti- MDR siRNA可以抑制细胞的耐药产生[18]。 如何在体内实现靶基因siRNA转移到所有肿瘤细胞并稳定持久的抑制癌基的表达是RNAi技术治疗肿瘤的关键。此外,从目前RNAi的研究现状来看,在哺乳动物细胞中,RNAi并不能完全阻断基因的表达,尤其是异常高表达的基因[3], 这也将影响对肿瘤的治疗疗效。尽管如此,但RNAi的高效特异性抑制癌基因表达以及明显优于反义核酸的效果的特征显示: RNAi将是一项令人不可忽视的抗肿瘤新技术。 2002年12月20日, Small RNA & RNAi 被Science杂志评为年度十大科技成就之首,Nature杂志亦将Small RNA评为年度重大科技成果之一。RNA研究的突破性进展,是生物医学领域近20年来,可与人类基因组计划(human genomics program,HGP)相提并论的最重大成果之一。RNAi最主要的功能在于可以调节和关闭基因的表达,进而调控细胞的各种高级生命活动。人们对小RNA分子的深入研究必将大大推进基因功能的研究,更为各种病毒性疾病和肿瘤等疾病的根治,开辟新的治疗途径。Small RNA与RNAi的研究与应用,具有极其重要的理论和实际意义,它必将对生物学和医学的发展产生深远的影响。