染色质结构与基因表达调控
丁香园论坛
5012
主题:
染色质结构与基因表达调控
胡建广 杨金水
Chromatin Structure and Gene Expression
HU Jian-Guang YANG Jin-Shui
(Institute of Genetics, Fudan University, Shanghai 200433)
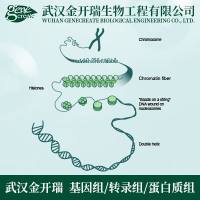
真核细胞中DNA与蛋白质结合形成染色质和染色体是生物进化的必然结果〔1〕。
核小体(Nucleosome)是染色质的基本结构单位,其为长度约160bp的DNA绕由H2A, H2B, H3和H4各两分子形成的八聚体约两圈的粒状结构, 其中H3和H4各两分子形成的四聚体位于八聚体的中间, 其两端均与H2A和H2B各一分子形成的二聚体结合。第五种组蛋白H1或称H5与连接两核小体之间的DNA结合, 并与相邻两核小体首尾相联, 以稳定核心组蛋白与DNA双链的结合。Crane等(1997)对H1与核小体的结合位置提出新的看法〔2〕, 认为染色质的高级结构是以核小体为基本结构单位的一级结构经过进一步的盘绕、折叠而形成。由于染色质是真核细胞遗传信息的载体, 真核基因的表达调控一定与染色质密切相关。研究染色质的结构及其在基因表达调控中的作用, 对于揭示真核生物基因表达调控的机理有重要意义。
1 转录区染色质对DNaseI的敏感性增加
真核生物的染色质结构在不同物种间无显著差异, 核小体的体积也基本恒定, 但核小体间连接DNA的长度是可变的, 它在不同的物种中, 以及不同的组织, 有时不同的发育阶段均有可能不同。例如, 菜豆(Phaseolus vulgaris)的子叶和叶片组织中, 染色质核小体的重复长度有差异, 叶片中为191bp, 而子叶中缩短到177bp, 但没有发现这种差异与基因的特异表达有关〔3〕。Murray和kennard的进一步研究却发现, 菜豆子叶特异表达的基因-菜豆蛋白基因(phaseolin)的染色质区对DNaseⅠ的敏感性比叶片中强, 说明染色质对DNaseⅠ敏感与基因的转录有关。在豌豆(Pisum sativum)中, Steinmuller 等(1986)〔4〕研究了另一储藏蛋白-豆球蛋白(Legumin)基因的染色质在子叶和叶片中变化。在子叶中靠近豆球蛋白基因3'端的染色质对DNaseⅠ的敏感度比叶片中高5倍。大麦(Hordeum vulgare)中, 大麦醇溶蛋白(Hordein)基因, 捕光叶绿素a/b蛋白(light harvesting chlorophyll a/b protein, LHCP) 基因和15~16 kD多肽基因的染色质对DNaseⅠ的敏感度也随着这些基因的表达而显著的增加〔4〕。
在动物组织细胞中的研究同样显示, 基因的转录伴随着其染色质对DNaseⅠ的敏感性显著增加。母鸡中, 球蛋白基因在网织红细胞中表达, 而卵清蛋白基因则于输卵管中有转录; 研究发现, 网织红细胞中编码球蛋白mRNA的染色质DNA更易被DNaseⅠ降解, 输卵管细胞中卵清蛋白基因区域的染色质比球蛋白基因区对DNaseⅠ更敏感〔5,6〕。
关于染色质对DNaseⅠ敏感与该区基因被转录的关系, 研究者认为, 基因的转录造成染色质易被DNaseⅠ消化, 而非前者是后者的原因。
2 染色质中DNaseⅠ超敏感点与基因的表达调控有关
与由于基因的转录而使转录区染色质对DNaseⅠ的敏感性增加不同, 染色质中还存在一些短的区段, 一般为50~200bp之间, 对DNaseⅠ消化更敏感, 称DNaseⅠ超敏感点 (DNaseⅠHypersensivity sites DH sites) , 为无核小体区〔7〕。直接的证据来自Choder等的实验结果, 他们用电镜观察SV40微小染色体时发现其中存在无核小体区段, 该区段与限制内切酶分析定位的超敏感点在同一位置。经过对很多基因染色质的研究,发现超敏感点是广泛存在的,一般位于基因的5'端启动子之内或启动子的周围。玉米中有两个基因位点(Adh1和Adh2)编码乙醇脱氢酶(alcohol dehydrogenase), 它们均受厌氧协迫而表达。Paul等(1987)〔8〕发现,Adh1的5'端区域存在两种类型的DNaseⅠ超敏感点, 即组成型和诱导型。前者位于基因更上游位置, 大约为-160~170之间, 经厌氧诱导后, 出现两个新的DNaseⅠ超敏感点, 主要者中心位于-40, 次要者中心位于-140, 两者之间为厌氧反应域(anaerobic response regions), 其中至少有两个顺序, -30位的TATAA和-90位的CAAT, 被认为对基因的调控非常重要。厌氧环境除了诱导产生两个超敏感点以外, 还使组成型DNaseⅠ超敏感点区域的范围扩大。
玉米Adh2 基因5'端存在三个超敏感点均为组成型的, 它们的中心位置分别为-55, -305, 和-455, 当该基因受诱导而表达时, 超敏感点的敏感度增加, 并且靠近TATAA序列的超敏感点(即-55位)向3'端方向延伸, 使TATAA框暴露。
存在于基因调控区(包括启动子和增强子)的DNaseⅠ超敏感点(组成型或经诱导产生)对基因的表达是必要的。在人类β-球蛋白基因的5'端的上游, 大约10~20kb位置, 存在着位点控制区(Local control regions, LCRs)或称位点激活区 (Local activating regions, LARS), 它可以使其下游的基因的表达不受其所在染色体位置的影响。LCRs中存在4个超敏感点, 如果LCRs与人类β-球蛋白基因(包括启动子和其他一些顺式调控元件)一起转化小鼠, 则球蛋白mRNA在转化小鼠的合成量与整合的基因的拷贝数成正相关, 与其整合的位置无关。但如果其中LCRs的两个超敏感点(即5'-SH和HS-3')没有被整合, 则球蛋白的表达量很少, LCRs失去了其原有的功能〔9〕。
3 染色质无核小体区形成的机理
在染色质的DNaseⅠ超敏感点内没有完整的核小体结构形成。体外实验表明,虽然基因编码区的核小体对基因的转录影响较小, 但核心启动子区形成核小体后, 转录将无法起始〔10〕。基因的转录要求其核心启动子区和上游调控原件(Core promoter and upstream regulatory elements)为无核小体区或该区核小体被去除(Displacement) 。研究基因自然状态下及基因转录状态下染色质的结构, 发现真核细胞无核小体区的形成至少采取两种策略: 核小体的持久型去除(Persistant displacement) 和诱导去除 (Induced displacement)〔11〕。
持久型去除主要指在启动子元件(Promoter element)区自然状态下不形成核小体, 它们主要存在于组成型转录的启动子中, 某些诱导表达的基因在被诱导转录前其启动子中也发现这种类型的无核小体区。
持久型无核小体区的形成可能涉及组成型因子的作用。在DNA复制过程中, 核小体未装配之前可以出现短暂的无核小体染色质区, 有些组成型因子优先与该区DNA结合, 并阻碍随后该区核小体的形成。在果蝇热休克基因中, 通用转录因子(General factors)和GAGA因子(CT因子)很可能参与了核心启动子区(即TATA盒和转录起始点)持久型无核小体区的形成, 并使上游调控元件维持对热击因子的易接近性〔12〕。SP1因子也可能参与持久型无核小体区的形成。很多“管家基因(Housekeeping genes)” 均含有多个SP1结合位点。SP1与这些位点结合后, 阻碍了组蛋白H1的结合。在酵母(S. cerevisiae)中,有一类组成型表达的蛋白质, 称为通用调节因子(General regulatory factors, GRFs), 它们在酵母细胞中的含量特别丰富, 每一种均与特定序列的DNA结合, 并使该区为无核小体区。当GRF结合点突变后,研究发现GRF与其结合点的结合为该无核小体区的形成所必需, 并对相邻区域核小体的形成产生重要影响。在his4基因的HSE区有RAP1(GRF中的一种)的结合位点, RAP1与其结合位点结合后产生无核小体区, 该无核小体区为诱导因子GCN4和BAS1与BAS2等基本因子(basal factors)在该区起作用时所必需〔13〕。
诱导去除是指基因在表达前其上游因子结合位点(Upstream factors-binding sites)以及核心启动子区均形成核小体, 经诱导后, 调节因子直接或间接地引起核小体的去除。
pho5基因在酵母中编码酸性磷酸酶, 当酵母生长的培养基中磷酸缺乏时, 该基因被诱导而表达。pho5启动子区在抑制状态下形成6个核小体, 在倒数第二个核小体上(从转录起始点算起)有PHO4和PHO2两个反式作用蛋白的结合位点, 第一个结合位点位于TATA框在倒数第一个核小体上, PHO4的另一结合点位于倒数第二和第三个核小体间的连接DNA上〔14〕。突变分析发现, PHO4和PHO2两蛋白为产生具有转录活性的染色质和pho5基因表达所必需, 在磷酸缺乏时, PHO4蛋白先与连接DNA的靶位点结合, 在PHO2的协同作用下, 引起第二位核小体的解体, 暴露PHO4的第二个结合点。Svaren(1997)等〔15〕的研究还表明, PHO4与其结合位点结合后, 其他蛋白复合体又与PHO4蛋白结合, 使该区的染色质的结构改变。后结合上的蛋白复合体具有ATP酶的活性, 说明染色质结构的改变是一个耗能的过程。PHO4的功能同时还受pho80和pho85 两个基因产物的调控, PHO80和PHO85两蛋白组成激酶复合体, 对PHO4蛋白进行磷酸化使其激活功能被抑制〔16〕。PHO5的表达需要倒数前四个核小体的解体。现在还不知道其他三个核小体被替代的具体方式, 但能量平衡是一个重要原因, 第二个核小体上DNA序列的变化, 将使启动子的诱导和核小体解体更容易。
与pho5基因相似, 鼠乳腺瘤病毒(mouse mammary tumor virus MMTV)长末端重复(long terminal repeat LTR)顺序的启动子受糖皮质激素受体(glucocorticoid receptor GR)的调控。该启动子也形成6个位置恒定的核小体,在倒数第二个核小体表面有四个糖皮质激素受体的识别位点, 分别位于-175, -119, -98和-83, 其中-175 和-83两结合位点DNA的主沟面对溶液, 可以与GR结合, 这两个位点相距92bp。由于核小体的形成,这两个位点正好位于同一核小体的同一侧, 有利于GR的结合和后来的激活过程。另两位点其主沟面对组蛋白,因受空间的阻碍与GR没有结合能力〔17〕。 在被诱导条件下, GR以二聚体形式与核小体上的识别位点结合, 但似乎并没有使其结合区核心组蛋白被去除, 而是形成了GR-核小体复合体。体内研究表明, 具有转录活性的启动子几乎无例外的为DNaseI超敏感区, 指示它们缺乏正常的核小体结构。GR结合后可能促进其他蛋白复合体(其中包括SWI1、2、3 蛋白在哺乳动物中的类似物)与核心组蛋白作用, 引起核小体的解体〔18〕。SWI1、2、3 是酵母基因编码的蛋白质,为GAL1和GAL10等基因的转录以及酵母GAL4蛋白起作用时所必需, 它们可能与另两种蛋白SNF5和SNF6形成多亚基复合体, 辅佐基因专一性蛋白起作用〔19〕。由GR诱导的转录是短暂的, 几个小时以后, 基本转录复合体, NF1, GR和八聚体因子又被核心组蛋白替代, 它们的结合位点又形成核小体, 启动子转录功能被抑制。转录因子被替代的机理还不清楚,组蛋白和转录因子与启动子区的结合很可能是一个动态过程。 Wong等〔20〕对甲状腺激素β-受体的突变研究发现, 该激素受体与甲状腺结合后, 可以引起其靶位点染色质的解体和调控基因转录起始, 但这两个过程分别与激素受体的不同功能区有关,染色质的解体不足以引起基因的转录起始。
4 组蛋白与基因的转录调控有关
核心组蛋白明显分为两个区域——羧基末端的疏水区和氨基末端的亲水区, 羧基端形成球形结构参与核小体核心的形成, 氨基端带正电荷位于核小体的外部, 与周围环境作用, 为基因的表达调控所必需, 并且不同核心组蛋白的氨基末端所起作用不同。
Kayne 等(1988)〔21〕发现, H4的N-末端的缺失可以解除酵母沉默交配位点 (silent mating loci)HMLα和HMRa的抑制作用,使邻近酵母β2启动子位置的核小体解体。H4末端的4个带正电荷氨基酸残基(Lys16, Arg17, His18或Arg19)的其中之一被甘氨酸替代, 均可以使交配频律降低5 000倍〔22〕。H3和H4的氨基末端对酵母端粒区沉默也有重要作用, 它们N-末端的缺失或一些氨基酸的替代均可以解除端粒沉默(telomeric silencing) 。H3和H4的N-末端对GAL1启动子也有重要的调节功能, 但它们所起的作用并不相同。H4组蛋白N-末端第4~23位氨基酸的缺失或该区段乙酰化位点的半胱氨酸残基(即第5、8、12和16位)被其他氨基酸替代, 将大大降低GAL1启动子的活性;与此相反, H3组蛋白N-末端4~15位氨基酸区段的缺失以及位于该区或邻近区域的半胱氨酸残基(即第9、14、18和23位)的替代, 可以使GAL1启动子活性增加。Fisher-Adams等(1995)〔23〕在H3和H4 N-末端缺失对GAL1启动子转录调控的影响方面进行了很精细的研究,也得到相似的结论。
组蛋白N-末端乙酰化对染色质结构和基因表达调控有重要影响。 Allfery等(1964)〔24〕认为, 染色质组蛋白N-末端半胱氨酸残基的乙酰化,可以作为该染色质具有转录活性的标志。组蛋白乙酰化减弱了染色质对DNA 的限制, 使核小体的构象发生变化, 转录因子接近核小体的机会增加,因而对基因的转录是有利的。当组蛋白的乙酰化去除后, 伴随着转录的关闭, 同时,组蛋白的乙酰化程度又决定于乙酰化酶和去乙酰化酶的平衡, 因此, Wolffe(1996)认为〔25〕, 对某一特定的基因而言, 其染色质的结构和基因转录与否决定于乙酰化酶和去乙酰化酶的比例。目前, 在多种生物中, 乙酰转移酶基因和去乙酰化酶基因均已被克隆。P/CAF为人类中被克隆的乙酰转移酶基因,研究表明, P/CAF基因产物与已知的转录共激活子(coactivator)CBP和p300结合, 后者与转录因子(例如CREB, c-JUN和FOS)结合, 因此, Sternglanz(1996)等〔26〕认为, 乙酰转移酶通过与转录因子和共激活子结合, 催化启动子区核心组蛋白的乙酰化。
5 核小体与基因调控
核小体是染色质的基本结构单位。体内外实验均证实, 核小体为基因转录的一个通用抑制子(general repressor)。由于缺少H4组蛋白,核小体不能形成的酵母细胞系中, 很多基因变为组成型表达,而在正常的细胞株系中, 它们均处于抑制状态。
但大量的实验结果证明, 核小体的形成和在染色质的精确定位为真核基因的表达所必需〔27〕。在MMTVLTR启动子中, 两个GR结合位点(第-175和-83)相距92bp, 由于核小体的形成, 它们正好位于核小体的同一侧面, 在空间上非常接近, 这显然有利于GR的结合, 因而对基因的诱导表达有利。
果蝇hsp26启动子中,第-51位和第-34位为热休克转录因子(heat shock transcription factor, HSTF) 的识别位点, 由于在第-330至-140区段形成一个核小体,也使得HSTF两个位点在空间上非常接近〔28〕。相似的情况也存在于果蝇ADH(alcohol dehydrogenase)基因和青蛙的卵黄原基因。卵黄原基因DNA的-300至-140区段与组蛋白结合形成核小体,转录促进因子(stimulatory transcription factors)、雌激素受体和NF1的结合点分别位于该核小体两侧的-300至-330 和-120至-110位, 核小体的形成或该区段的人工缺失, 均可以使该基因的转录量提高5~10倍。因此, Wolffe(1994)〔29〕认为, 核小体的形成及其在染色质上的精确定位有以下两方面的作用:(1) 提供一个框架(scaffold), 使转录因子之间的信息传递更有效; (2) 染色质结构的不均一性,即其某些区域不形成核小体, 从而保证了转录因子易于接近染色质模板。
染色质是真核生物遗传物质的载体, 真核基因的表达调控显然与染色质的结构改变有关。染色质的结构在具有转录活性的基因区域确实与基因活性惰性区域有很大的不同,同时, 其结构也存在不均一性,即在启动子或增强子区存在DNaseI超敏感点, 它们为基因的表达调控所必需。染色质的重要组成成分-组蛋白,在真核基因的表达调控中也起着非常重要的作用,核小体的形成和正确定位为基因的时空表达提供了重要保证。虽然这里只简单介绍染色质的结构和部分组成成分与基因表达的关系,但仍然可以领悟染色质结构和功能的高度一致。
作者单位:复旦大学遗传学研究所, 上海 200433
参考文献
[1] Eissenberg J C et al. Selected topics in chromatin structure. Annu. Rev. Genet., 1985, 19: 485~536
[2] Crane-Rodinson C. Where is the grobular domain if linking histone located in the nucleosome. TIBS, 1997, 22(3): 75~77
[3] Murray M G et al. #FSAltered chromatin structure of higher plant gene phaseolin.Biochemistry, 1984, 23: 4225~4232
[4] Steinmuller K et al. A simple and efficient procedure for isolating plant chromatin which is suitable for studies of DNaseI sensitive domains and hypersensitive sites. Plant Mol. Biol., 1986, 7: 87~94
[5] Fritton H P et al. Alterative sets of DNaseI hypersensitive sites characterize the various functional sites of the chicken lysozyme gene. Nature, 1984, 311: 163~165
[6] Elgin S C R. Anatony of hypersensitive sites. Nature, 1984, 309: 213~214
[7] Paul A L et al. Constitutive and anaerobically induced DNaseI hypersensitive sites in the 5' region of the maize Adh1 gene. Proc. Natl. Acad. Sci. USA, 1987, 84: 799~803
[8] Felsenfeld G. Chromatin as an essential part of the transcriptional mechanism. Nature, 1992, 355: 219~224
[9] Lorch Y et al. Nucleosomes inhibit the initiation of transcription but allow chain elongation with the displacement of histones. Cell, 1987, 49: 203~210
[10] Workman J et al. Multiple functions of nucleosomes and regulatory factors in transcription. TIBS, 1993, 18(3): 90~95
[11] Wolffe A P. Implication of DNA replication for eukaryotic gene. J. Cell Sci., 1991, 99(2): 201~206
[12] Gilmour D S et al. Drosophila nuclear proteins bind to regions of alteracting C and T residues in gene promoters. Science, 1989, 245(4944): 1487~1490
[13] Chasman D et al. A yeast protein that influences the chromatin structure of UASG and functions as a powerful auxiliary gene activator. Gene Dev., 1990, 4: 503~514
[14] Almer A et al. Remove of positioned nucleosomes from the yeast PHO5 induction release additional upstream activating DNA elements. EMBO J., 1986, 5: 2689~2696
[15] Svaren J et al. Transcription factors vs nucleosomes regulation of the PHO5 promoter in yeast. TIBS, 1997, 22(3): 93~97
[16] Kaffman A et al. Phosphorylation of the transcription pho4 by a cylin-CDK coplex pho80-pho85, Science, 1994, 263: 1153~1156
[17] Pina B et al. nucleosome positioning modulates accessibility of regulatory proteins to the mouse mammary tumor virus promoter. Cell, 1990, 60: 719~731
[18] Muchardt G et al. A human homologue of Saccharomyces cerevisiae SNF2/SWI2 and Drosophila brm genes potentiates transcriptional activation by the glucocortcoid receptor. EMBO J., 1993, 12: 4279~4293
[19] Peterson G L et al. Characterizaion of the yeast SwI1, Swl2 and Swl3 genes which encode a global activator of transcription. Cell, 1992, 68: 573~583
[20] Wong J et al. Determinants of chromatin disruption and transcriptional regulation instigated by the thyroid hormone receptor: hormone-regulated chromatin disruption is not sufficient for transcriptional activation. EMBO, J., 1997, 16(11): 3158~3171
[21] Thompson J S et al. Histone H3 amino terminus is required for telomeric and silent mating locus repression in yeast. Nature, 1994, 369: 245~247
[22] Fisher-Adams G et al. Yeast histone H4 and H3 N-termini have different effects on the chromatin structure of the GAL1 promoter. EMBO J., 1995, 14(7): 1468~1477
[23] Allfrey G et al. Acetylation and methylation of histone and their possible role in the regulation of RNA synthesis. Proc. Natl. Acad. Sci. USA, 1964, 51(5): 786~790
[24] Bauer W R et al. Nucleosome structural changes due to acetylation. J. Mol. Biol., 1994, 236: 685~690
[25] Wolffe A P. Histone deacetylase: A regulator of transcription. Science, 1996, 272: 371~372
[26] Sternglanz R. Histone acetylation: A gateway to transcriptional activation. TIBS, 1996, 21(10): 375~358
[27] Schild C et al. A nuclosome-dependent static loop potentiates estrogen-regulated transcription from the Xenops viteuogenin Bi promoter in vitro. EMBO J., 1993, 12: 423~433
[28] Jackson J R et al. DNA-histone interactions are sufficient to position a single nucleosome juxtaosing Drosophila Adh adult enhancer and distal promoter. Nucleic Acids Res., 1993, 21: 975~967
[29] Woffle A P. Nucleosome positioning and modification: Chromatin structure that potentiate transcription. TIBS, 1994, 19(6): 240~244
染色质结构与基因表达调控
胡建广 杨金水
Chromatin Structure and Gene Expression
HU Jian-Guang YANG Jin-Shui
(Institute of Genetics, Fudan University, Shanghai 200433)
真核细胞中DNA与蛋白质结合形成染色质和染色体是生物进化的必然结果〔1〕。
核小体(Nucleosome)是染色质的基本结构单位,其为长度约160bp的DNA绕由H2A, H2B, H3和H4各两分子形成的八聚体约两圈的粒状结构, 其中H3和H4各两分子形成的四聚体位于八聚体的中间, 其两端均与H2A和H2B各一分子形成的二聚体结合。第五种组蛋白H1或称H5与连接两核小体之间的DNA结合, 并与相邻两核小体首尾相联, 以稳定核心组蛋白与DNA双链的结合。Crane等(1997)对H1与核小体的结合位置提出新的看法〔2〕, 认为染色质的高级结构是以核小体为基本结构单位的一级结构经过进一步的盘绕、折叠而形成。由于染色质是真核细胞遗传信息的载体, 真核基因的表达调控一定与染色质密切相关。研究染色质的结构及其在基因表达调控中的作用, 对于揭示真核生物基因表达调控的机理有重要意义。
1 转录区染色质对DNaseI的敏感性增加
真核生物的染色质结构在不同物种间无显著差异, 核小体的体积也基本恒定, 但核小体间连接DNA的长度是可变的, 它在不同的物种中, 以及不同的组织, 有时不同的发育阶段均有可能不同。例如, 菜豆(Phaseolus vulgaris)的子叶和叶片组织中, 染色质核小体的重复长度有差异, 叶片中为191bp, 而子叶中缩短到177bp, 但没有发现这种差异与基因的特异表达有关〔3〕。Murray和kennard的进一步研究却发现, 菜豆子叶特异表达的基因-菜豆蛋白基因(phaseolin)的染色质区对DNaseⅠ的敏感性比叶片中强, 说明染色质对DNaseⅠ敏感与基因的转录有关。在豌豆(Pisum sativum)中, Steinmuller 等(1986)〔4〕研究了另一储藏蛋白-豆球蛋白(Legumin)基因的染色质在子叶和叶片中变化。在子叶中靠近豆球蛋白基因3'端的染色质对DNaseⅠ的敏感度比叶片中高5倍。大麦(Hordeum vulgare)中, 大麦醇溶蛋白(Hordein)基因, 捕光叶绿素a/b蛋白(light harvesting chlorophyll a/b protein, LHCP) 基因和15~16 kD多肽基因的染色质对DNaseⅠ的敏感度也随着这些基因的表达而显著的增加〔4〕。
在动物组织细胞中的研究同样显示, 基因的转录伴随着其染色质对DNaseⅠ的敏感性显著增加。母鸡中, 球蛋白基因在网织红细胞中表达, 而卵清蛋白基因则于输卵管中有转录; 研究发现, 网织红细胞中编码球蛋白mRNA的染色质DNA更易被DNaseⅠ降解, 输卵管细胞中卵清蛋白基因区域的染色质比球蛋白基因区对DNaseⅠ更敏感〔5,6〕。
关于染色质对DNaseⅠ敏感与该区基因被转录的关系, 研究者认为, 基因的转录造成染色质易被DNaseⅠ消化, 而非前者是后者的原因。
2 染色质中DNaseⅠ超敏感点与基因的表达调控有关
与由于基因的转录而使转录区染色质对DNaseⅠ的敏感性增加不同, 染色质中还存在一些短的区段, 一般为50~200bp之间, 对DNaseⅠ消化更敏感, 称DNaseⅠ超敏感点 (DNaseⅠHypersensivity sites DH sites) , 为无核小体区〔7〕。直接的证据来自Choder等的实验结果, 他们用电镜观察SV40微小染色体时发现其中存在无核小体区段, 该区段与限制内切酶分析定位的超敏感点在同一位置。经过对很多基因染色质的研究,发现超敏感点是广泛存在的,一般位于基因的5'端启动子之内或启动子的周围。玉米中有两个基因位点(Adh1和Adh2)编码乙醇脱氢酶(alcohol dehydrogenase), 它们均受厌氧协迫而表达。Paul等(1987)〔8〕发现,Adh1的5'端区域存在两种类型的DNaseⅠ超敏感点, 即组成型和诱导型。前者位于基因更上游位置, 大约为-160~170之间, 经厌氧诱导后, 出现两个新的DNaseⅠ超敏感点, 主要者中心位于-40, 次要者中心位于-140, 两者之间为厌氧反应域(anaerobic response regions), 其中至少有两个顺序, -30位的TATAA和-90位的CAAT, 被认为对基因的调控非常重要。厌氧环境除了诱导产生两个超敏感点以外, 还使组成型DNaseⅠ超敏感点区域的范围扩大。
玉米Adh2 基因5'端存在三个超敏感点均为组成型的, 它们的中心位置分别为-55, -305, 和-455, 当该基因受诱导而表达时, 超敏感点的敏感度增加, 并且靠近TATAA序列的超敏感点(即-55位)向3'端方向延伸, 使TATAA框暴露。
存在于基因调控区(包括启动子和增强子)的DNaseⅠ超敏感点(组成型或经诱导产生)对基因的表达是必要的。在人类β-球蛋白基因的5'端的上游, 大约10~20kb位置, 存在着位点控制区(Local control regions, LCRs)或称位点激活区 (Local activating regions, LARS), 它可以使其下游的基因的表达不受其所在染色体位置的影响。LCRs中存在4个超敏感点, 如果LCRs与人类β-球蛋白基因(包括启动子和其他一些顺式调控元件)一起转化小鼠, 则球蛋白mRNA在转化小鼠的合成量与整合的基因的拷贝数成正相关, 与其整合的位置无关。但如果其中LCRs的两个超敏感点(即5'-SH和HS-3')没有被整合, 则球蛋白的表达量很少, LCRs失去了其原有的功能〔9〕。
3 染色质无核小体区形成的机理
在染色质的DNaseⅠ超敏感点内没有完整的核小体结构形成。体外实验表明,虽然基因编码区的核小体对基因的转录影响较小, 但核心启动子区形成核小体后, 转录将无法起始〔10〕。基因的转录要求其核心启动子区和上游调控原件(Core promoter and upstream regulatory elements)为无核小体区或该区核小体被去除(Displacement) 。研究基因自然状态下及基因转录状态下染色质的结构, 发现真核细胞无核小体区的形成至少采取两种策略: 核小体的持久型去除(Persistant displacement) 和诱导去除 (Induced displacement)〔11〕。
持久型去除主要指在启动子元件(Promoter element)区自然状态下不形成核小体, 它们主要存在于组成型转录的启动子中, 某些诱导表达的基因在被诱导转录前其启动子中也发现这种类型的无核小体区。
持久型无核小体区的形成可能涉及组成型因子的作用。在DNA复制过程中, 核小体未装配之前可以出现短暂的无核小体染色质区, 有些组成型因子优先与该区DNA结合, 并阻碍随后该区核小体的形成。在果蝇热休克基因中, 通用转录因子(General factors)和GAGA因子(CT因子)很可能参与了核心启动子区(即TATA盒和转录起始点)持久型无核小体区的形成, 并使上游调控元件维持对热击因子的易接近性〔12〕。SP1因子也可能参与持久型无核小体区的形成。很多“管家基因(Housekeeping genes)” 均含有多个SP1结合位点。SP1与这些位点结合后, 阻碍了组蛋白H1的结合。在酵母(S. cerevisiae)中,有一类组成型表达的蛋白质, 称为通用调节因子(General regulatory factors, GRFs), 它们在酵母细胞中的含量特别丰富, 每一种均与特定序列的DNA结合, 并使该区为无核小体区。当GRF结合点突变后,研究发现GRF与其结合点的结合为该无核小体区的形成所必需, 并对相邻区域核小体的形成产生重要影响。在his4基因的HSE区有RAP1(GRF中的一种)的结合位点, RAP1与其结合位点结合后产生无核小体区, 该无核小体区为诱导因子GCN4和BAS1与BAS2等基本因子(basal factors)在该区起作用时所必需〔13〕。
诱导去除是指基因在表达前其上游因子结合位点(Upstream factors-binding sites)以及核心启动子区均形成核小体, 经诱导后, 调节因子直接或间接地引起核小体的去除。
pho5基因在酵母中编码酸性磷酸酶, 当酵母生长的培养基中磷酸缺乏时, 该基因被诱导而表达。pho5启动子区在抑制状态下形成6个核小体, 在倒数第二个核小体上(从转录起始点算起)有PHO4和PHO2两个反式作用蛋白的结合位点, 第一个结合位点位于TATA框在倒数第一个核小体上, PHO4的另一结合点位于倒数第二和第三个核小体间的连接DNA上〔14〕。突变分析发现, PHO4和PHO2两蛋白为产生具有转录活性的染色质和pho5基因表达所必需, 在磷酸缺乏时, PHO4蛋白先与连接DNA的靶位点结合, 在PHO2的协同作用下, 引起第二位核小体的解体, 暴露PHO4的第二个结合点。Svaren(1997)等〔15〕的研究还表明, PHO4与其结合位点结合后, 其他蛋白复合体又与PHO4蛋白结合, 使该区的染色质的结构改变。后结合上的蛋白复合体具有ATP酶的活性, 说明染色质结构的改变是一个耗能的过程。PHO4的功能同时还受pho80和pho85 两个基因产物的调控, PHO80和PHO85两蛋白组成激酶复合体, 对PHO4蛋白进行磷酸化使其激活功能被抑制〔16〕。PHO5的表达需要倒数前四个核小体的解体。现在还不知道其他三个核小体被替代的具体方式, 但能量平衡是一个重要原因, 第二个核小体上DNA序列的变化, 将使启动子的诱导和核小体解体更容易。
与pho5基因相似, 鼠乳腺瘤病毒(mouse mammary tumor virus MMTV)长末端重复(long terminal repeat LTR)顺序的启动子受糖皮质激素受体(glucocorticoid receptor GR)的调控。该启动子也形成6个位置恒定的核小体,在倒数第二个核小体表面有四个糖皮质激素受体的识别位点, 分别位于-175, -119, -98和-83, 其中-175 和-83两结合位点DNA的主沟面对溶液, 可以与GR结合, 这两个位点相距92bp。由于核小体的形成,这两个位点正好位于同一核小体的同一侧, 有利于GR的结合和后来的激活过程。另两位点其主沟面对组蛋白,因受空间的阻碍与GR没有结合能力〔17〕。 在被诱导条件下, GR以二聚体形式与核小体上的识别位点结合, 但似乎并没有使其结合区核心组蛋白被去除, 而是形成了GR-核小体复合体。体内研究表明, 具有转录活性的启动子几乎无例外的为DNaseI超敏感区, 指示它们缺乏正常的核小体结构。GR结合后可能促进其他蛋白复合体(其中包括SWI1、2、3 蛋白在哺乳动物中的类似物)与核心组蛋白作用, 引起核小体的解体〔18〕。SWI1、2、3 是酵母基因编码的蛋白质,为GAL1和GAL10等基因的转录以及酵母GAL4蛋白起作用时所必需, 它们可能与另两种蛋白SNF5和SNF6形成多亚基复合体, 辅佐基因专一性蛋白起作用〔19〕。由GR诱导的转录是短暂的, 几个小时以后, 基本转录复合体, NF1, GR和八聚体因子又被核心组蛋白替代, 它们的结合位点又形成核小体, 启动子转录功能被抑制。转录因子被替代的机理还不清楚,组蛋白和转录因子与启动子区的结合很可能是一个动态过程。 Wong等〔20〕对甲状腺激素β-受体的突变研究发现, 该激素受体与甲状腺结合后, 可以引起其靶位点染色质的解体和调控基因转录起始, 但这两个过程分别与激素受体的不同功能区有关,染色质的解体不足以引起基因的转录起始。
4 组蛋白与基因的转录调控有关
核心组蛋白明显分为两个区域——羧基末端的疏水区和氨基末端的亲水区, 羧基端形成球形结构参与核小体核心的形成, 氨基端带正电荷位于核小体的外部, 与周围环境作用, 为基因的表达调控所必需, 并且不同核心组蛋白的氨基末端所起作用不同。
Kayne 等(1988)〔21〕发现, H4的N-末端的缺失可以解除酵母沉默交配位点 (silent mating loci)HMLα和HMRa的抑制作用,使邻近酵母β2启动子位置的核小体解体。H4末端的4个带正电荷氨基酸残基(Lys16, Arg17, His18或Arg19)的其中之一被甘氨酸替代, 均可以使交配频律降低5 000倍〔22〕。H3和H4的氨基末端对酵母端粒区沉默也有重要作用, 它们N-末端的缺失或一些氨基酸的替代均可以解除端粒沉默(telomeric silencing) 。H3和H4的N-末端对GAL1启动子也有重要的调节功能, 但它们所起的作用并不相同。H4组蛋白N-末端第4~23位氨基酸的缺失或该区段乙酰化位点的半胱氨酸残基(即第5、8、12和16位)被其他氨基酸替代, 将大大降低GAL1启动子的活性;与此相反, H3组蛋白N-末端4~15位氨基酸区段的缺失以及位于该区或邻近区域的半胱氨酸残基(即第9、14、18和23位)的替代, 可以使GAL1启动子活性增加。Fisher-Adams等(1995)〔23〕在H3和H4 N-末端缺失对GAL1启动子转录调控的影响方面进行了很精细的研究,也得到相似的结论。
组蛋白N-末端乙酰化对染色质结构和基因表达调控有重要影响。 Allfery等(1964)〔24〕认为, 染色质组蛋白N-末端半胱氨酸残基的乙酰化,可以作为该染色质具有转录活性的标志。组蛋白乙酰化减弱了染色质对DNA 的限制, 使核小体的构象发生变化, 转录因子接近核小体的机会增加,因而对基因的转录是有利的。当组蛋白的乙酰化去除后, 伴随着转录的关闭, 同时,组蛋白的乙酰化程度又决定于乙酰化酶和去乙酰化酶的平衡, 因此, Wolffe(1996)认为〔25〕, 对某一特定的基因而言, 其染色质的结构和基因转录与否决定于乙酰化酶和去乙酰化酶的比例。目前, 在多种生物中, 乙酰转移酶基因和去乙酰化酶基因均已被克隆。P/CAF为人类中被克隆的乙酰转移酶基因,研究表明, P/CAF基因产物与已知的转录共激活子(coactivator)CBP和p300结合, 后者与转录因子(例如CREB, c-JUN和FOS)结合, 因此, Sternglanz(1996)等〔26〕认为, 乙酰转移酶通过与转录因子和共激活子结合, 催化启动子区核心组蛋白的乙酰化。
5 核小体与基因调控
核小体是染色质的基本结构单位。体内外实验均证实, 核小体为基因转录的一个通用抑制子(general repressor)。由于缺少H4组蛋白,核小体不能形成的酵母细胞系中, 很多基因变为组成型表达,而在正常的细胞株系中, 它们均处于抑制状态。
但大量的实验结果证明, 核小体的形成和在染色质的精确定位为真核基因的表达所必需〔27〕。在MMTVLTR启动子中, 两个GR结合位点(第-175和-83)相距92bp, 由于核小体的形成, 它们正好位于核小体的同一侧面, 在空间上非常接近, 这显然有利于GR的结合, 因而对基因的诱导表达有利。
果蝇hsp26启动子中,第-51位和第-34位为热休克转录因子(heat shock transcription factor, HSTF) 的识别位点, 由于在第-330至-140区段形成一个核小体,也使得HSTF两个位点在空间上非常接近〔28〕。相似的情况也存在于果蝇ADH(alcohol dehydrogenase)基因和青蛙的卵黄原基因。卵黄原基因DNA的-300至-140区段与组蛋白结合形成核小体,转录促进因子(stimulatory transcription factors)、雌激素受体和NF1的结合点分别位于该核小体两侧的-300至-330 和-120至-110位, 核小体的形成或该区段的人工缺失, 均可以使该基因的转录量提高5~10倍。因此, Wolffe(1994)〔29〕认为, 核小体的形成及其在染色质上的精确定位有以下两方面的作用:(1) 提供一个框架(scaffold), 使转录因子之间的信息传递更有效; (2) 染色质结构的不均一性,即其某些区域不形成核小体, 从而保证了转录因子易于接近染色质模板。
染色质是真核生物遗传物质的载体, 真核基因的表达调控显然与染色质的结构改变有关。染色质的结构在具有转录活性的基因区域确实与基因活性惰性区域有很大的不同,同时, 其结构也存在不均一性,即在启动子或增强子区存在DNaseI超敏感点, 它们为基因的表达调控所必需。染色质的重要组成成分-组蛋白,在真核基因的表达调控中也起着非常重要的作用,核小体的形成和正确定位为基因的时空表达提供了重要保证。虽然这里只简单介绍染色质的结构和部分组成成分与基因表达的关系,但仍然可以领悟染色质结构和功能的高度一致。
作者单位:复旦大学遗传学研究所, 上海 200433
参考文献
[1] Eissenberg J C et al. Selected topics in chromatin structure. Annu. Rev. Genet., 1985, 19: 485~536
[2] Crane-Rodinson C. Where is the grobular domain if linking histone located in the nucleosome. TIBS, 1997, 22(3): 75~77
[3] Murray M G et al. #FSAltered chromatin structure of higher plant gene phaseolin.Biochemistry, 1984, 23: 4225~4232
[4] Steinmuller K et al. A simple and efficient procedure for isolating plant chromatin which is suitable for studies of DNaseI sensitive domains and hypersensitive sites. Plant Mol. Biol., 1986, 7: 87~94
[5] Fritton H P et al. Alterative sets of DNaseI hypersensitive sites characterize the various functional sites of the chicken lysozyme gene. Nature, 1984, 311: 163~165
[6] Elgin S C R. Anatony of hypersensitive sites. Nature, 1984, 309: 213~214
[7] Paul A L et al. Constitutive and anaerobically induced DNaseI hypersensitive sites in the 5' region of the maize Adh1 gene. Proc. Natl. Acad. Sci. USA, 1987, 84: 799~803
[8] Felsenfeld G. Chromatin as an essential part of the transcriptional mechanism. Nature, 1992, 355: 219~224
[9] Lorch Y et al. Nucleosomes inhibit the initiation of transcription but allow chain elongation with the displacement of histones. Cell, 1987, 49: 203~210
[10] Workman J et al. Multiple functions of nucleosomes and regulatory factors in transcription. TIBS, 1993, 18(3): 90~95
[11] Wolffe A P. Implication of DNA replication for eukaryotic gene. J. Cell Sci., 1991, 99(2): 201~206
[12] Gilmour D S et al. Drosophila nuclear proteins bind to regions of alteracting C and T residues in gene promoters. Science, 1989, 245(4944): 1487~1490
[13] Chasman D et al. A yeast protein that influences the chromatin structure of UASG and functions as a powerful auxiliary gene activator. Gene Dev., 1990, 4: 503~514
[14] Almer A et al. Remove of positioned nucleosomes from the yeast PHO5 induction release additional upstream activating DNA elements. EMBO J., 1986, 5: 2689~2696
[15] Svaren J et al. Transcription factors vs nucleosomes regulation of the PHO5 promoter in yeast. TIBS, 1997, 22(3): 93~97
[16] Kaffman A et al. Phosphorylation of the transcription pho4 by a cylin-CDK coplex pho80-pho85, Science, 1994, 263: 1153~1156
[17] Pina B et al. nucleosome positioning modulates accessibility of regulatory proteins to the mouse mammary tumor virus promoter. Cell, 1990, 60: 719~731
[18] Muchardt G et al. A human homologue of Saccharomyces cerevisiae SNF2/SWI2 and Drosophila brm genes potentiates transcriptional activation by the glucocortcoid receptor. EMBO J., 1993, 12: 4279~4293
[19] Peterson G L et al. Characterizaion of the yeast SwI1, Swl2 and Swl3 genes which encode a global activator of transcription. Cell, 1992, 68: 573~583
[20] Wong J et al. Determinants of chromatin disruption and transcriptional regulation instigated by the thyroid hormone receptor: hormone-regulated chromatin disruption is not sufficient for transcriptional activation. EMBO, J., 1997, 16(11): 3158~3171
[21] Thompson J S et al. Histone H3 amino terminus is required for telomeric and silent mating locus repression in yeast. Nature, 1994, 369: 245~247
[22] Fisher-Adams G et al. Yeast histone H4 and H3 N-termini have different effects on the chromatin structure of the GAL1 promoter. EMBO J., 1995, 14(7): 1468~1477
[23] Allfrey G et al. Acetylation and methylation of histone and their possible role in the regulation of RNA synthesis. Proc. Natl. Acad. Sci. USA, 1964, 51(5): 786~790
[24] Bauer W R et al. Nucleosome structural changes due to acetylation. J. Mol. Biol., 1994, 236: 685~690
[25] Wolffe A P. Histone deacetylase: A regulator of transcription. Science, 1996, 272: 371~372
[26] Sternglanz R. Histone acetylation: A gateway to transcriptional activation. TIBS, 1996, 21(10): 375~358
[27] Schild C et al. A nuclosome-dependent static loop potentiates estrogen-regulated transcription from the Xenops viteuogenin Bi promoter in vitro. EMBO J., 1993, 12: 423~433
[28] Jackson J R et al. DNA-histone interactions are sufficient to position a single nucleosome juxtaosing Drosophila Adh adult enhancer and distal promoter. Nucleic Acids Res., 1993, 21: 975~967
[29] Woffle A P. Nucleosome positioning and modification: Chromatin structure that potentiate transcription. TIBS, 1994, 19(6): 240~244