M13 噬菌体衣壳蛋白改造在改良噬菌体展示技术中的应用
丁香园
5036
1. 引言
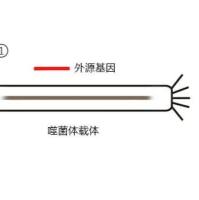
噬菌体展示是一项改造结合靶分子多肽的强大技术 [ 1 〜 3] 。蛋白质与噬菌体衣壳蛋白形成融合蛋白,可以在噬菌体表面表达,而相应的编码基因则包裹于噬菌体颗粒内 [4] 。包裹的 DNA 通过突变扩增构建噬菌体展示多肽库,为筛选各种变异体做准备。通过固定化靶分子体外结合实验,高亲和力的蛋白质可以从蛋白质展示库中筛选出来。随后,选定蛋白质的序列可以通过 DNA 测序推断出来。
靶蛋白可以通过构建噬菌粒载体与主要衣壳蛋白 [ 蛋白质-8 (P8) ] 或次要衣壳蛋白 [ 蛋白质 -3 ( P3 ) ] 形成的融合蛋白在 M13 噬菌体表面以低拷贝方式展示,这需要在野生型(wt) P8 和 P3 辅助噬菌体帮助的条件下进行 [5] 。由于融合蛋白的展示水平随蛋白质序列和长度的不同而不同,多价蛋白与 P8 融合进行展示难以实现 [6] 。例如,如果 P8 的每一个拷贝都与展示多肽形成融合蛋白(噬菌体系统,而不是噬菌粒系统),融合多肽长度超过6 个氨基酸残基,噬菌体一般就会不稳定 [7] 。即使在噬菌粒系统中,蛋白质展示也高度依赖于蛋白质的大小和性质 [8]。尽管用 P3 或 P8 进行单价噬菌体展示使许多不同蛋白质亲和力从中等提高到很高 [9],利用 P8 进行多价噬菌体展示从初级库中选择分子质量大的低亲和力受体蛋白尚未实现。
这里,我们描述一种通过突变 P8 锚定残基改进蛋白质展示的方法。利用这一方法,两个不同的蛋白质 [ ( 链霉亲和素和人生长激素(hGH ) ] 表达水平几乎增加了 100 倍,并且我们发现这一技术也适用于其他蛋白质。这一戏剧性的展示改进很可能由于融合蛋白在噬菌体表面定位更加恰当所致。理论上,高拷贝展示的应用应该可能使结合弱的蛋白质从初级天然库中选择出来,对于小肽这一方法已经成功可行。同样重要的是这一方法扩展了噬菌体展示研究蛋白质的范围,许多曾被认为难以展示的蛋白质用这一方法实现了展示。因此,这里描述的方法增加了噬菌体展示鉴定新蛋白质或修饰蛋白质功能的潜力。
2. 材料
2.1 P8 突变库的构建
2.1.1 制备含有尿嘧啶的单链 DNA 模板
( 1 ) 2YT 培养基:10 g 酵母抽提物、16 g 菌用胰蛋白胨和 5 g 氯化钠;加水到 1 L,用氢氧化钠调节 pH 到 7.0;高压灭菌。
( 2 ) 2YT/carb/cmp 培养基:2YT、50 μg/ml 羧苄青霉素(carbenicillin,carb) 和 5 μg/ml 氯霉素(chloramphenicol,cmp)。
( 3 ) 2YT/carb/kan/uridine 培养基:2YT、50 μg/ml 羧苄青霉素、25 μg/ml 卡那霉素和 0.25 μg/ml 尿嘧啶。
( 4 ) 羧苄青霉素:5 mg/ml,羧苄青霉素水溶液,超滤灭菌。
( 5 ) 氯霉素:50 mg/ml,氯霉素乙醇溶液。
( 6 ) 大肠杆菌 CJ236 ( NEB: New England Biolabs,Beverly,美国麻省)。
( 7 ) 卡那霉素:5 mg/ml,卡那霉素水溶液,超滤灭菌。
( 8 ) 生理磷酸缓冲液(PBS): 137 mmol/L 氯化钠、3 mmol/L 氯化钾、8 mmol/L 磷酸氢二钠以及 1.5 mmol/L 磷酸二氢钾;用盐酸(HCl ) 调节 pH 到 7.2;高压灭菌。
( 9 ) 聚乙二醇(polyethylene glycol,PEG) PEG/NaCl: 20% PEG-8000 ( m/V ) 和 2.5 mol/L 氯化钠;高压灭菌。
( 10 ) QIA prep Spin M13 试剂盒(Qiagen,Valencia,美国加州)。
( 11 ) M13KO7 辅助噬菌体(NEB,Beverly,美国麻省)。
( 12 ) Tris- acetate-EDIA (TAE) 缓冲液:40 mmol/L Tris-acetate 和 1 mmol/L EDTA;调节 pH 到 8.0;高压灭菌。
( 13 ) TAE/琼脂糖胶:TAE 缓冲液、1% 琼脂糖(m/V)以及 1 : 5000 ( V/V ) 10% 溴化乙锭(EB)。
( 14 ) 尿嘧啶:25 mg/ml,尿嘧啶水溶液,超滤灭菌。
2.1.2 体外合成异质共价闭环双链 DNA
( 1 ) 100 mmol/L 二硫苏糖醇(DTT )。
( 2 ) 25 mmol/L dNTP : 含有 dATP、dCTP、dGTP 和 dTTP 各 25 mmol/L 的溶液 ( Amersham-Pharmacia,Piscataway,美国新泽西州)。
( 3 ) 10 mmol/L ATP (Amersham-Pharmacia)。
( 4 ) 10 X Tris-Mg (TM) 缓冲液:0.1 mol/L MgCl2 以及 0.5 mol/L TrisHCl,pH 7.5。
( 5 ) QIAquick Gel Extraction 试剂盒(Qiagen,Valencia, 美国加州)。
( 6 ) T4 多聚核苷酸激酶(NEB: New England Biolabs) 。
( 7 ) T4 DNA 连接酶(Invitrogen, Carlsbad ,美国加州)。
( 8 ) TAE 琼脂糖凝胶(见 12.2.1.1 ) 。
( 9 ) USP ( United States Pharmacopeia ) 超纯水(Braun Medical In& , Irvine, 美国力口州)。
2.1.3 大肠杆菌电穿孔及噬菌体扩增
( 1 ) 2YT/carb 培养基:2YT 及 50 μg/ml 羧芐青霉素。
( 2 ) 羧芐青霉素(carbenicillin,carb) ( 见 12.2.1.1)。
( 3 ) 电转化感受态 E.coli SS320 ( 见 12.3.2.4)。
( 4 ) 0.2 cm 间隙电转化样品杯(BTX,圣地亚哥,美国加州)。
( 5 ) ECM-600 电转化仪(BTX )。
( 6 ) 卡那霉素 ( 见 12.2.1.1) 。
( 7 ) Luria-Bertani (LB) /carb 培养皿:LB 琼脂及 50 μg/ml 羧苄青霉素。
( 8 ) PBS ( 见 12.2.1.1)。
( 9 ) PEG/NaCl ( 见 12.2.1.1)。
( 10 ) SOC 培养基:5 g 细菌用酵母抽提物、20 g 细菌用胰蛋白胨和 0.5 g 氯化钠及 0.2 g 氯化钾;加水到大约1 L,用氢氧化钠调节 pH 到 7.0;高压灭菌;然后加入 5 ml 2.0 mol/L 高压灭菌过的氯化镁溶液及 20 ml 超滤灭菌过的 1.0 mol/L 葡萄糖溶液;并将最终体积调至 1 L。
( 11 ) M13KO7 辅助噬菌体(见 12.2.1.1)。
2.1.4 制备电穿孔法感受态细胞
( 1 ) 1.0 mmol/L HEPES,pH 7.4 : 将 4.0 ml 1.0 mol/L 的 HEPES,pH 7.4 加入 4.0 L 的 USP (U nited States Pharmacopeia) 超纯水,超滤灭菌。
( 2 ) 10% (V/V ) 的超纯甘油:100 ml 超纯甘油加入 900 ml USP 超纯水中,超滤灭菌。
( 3 ) 2YT/tet 培养基:2YT 及 5 μg/ml 四环素。
( 4 ) 磁性搅拌棒(约 5 cm 长),放入乙醇中浸泡。
( 5 ) Superbroth/tet 培养基:24 g 细菌用酵母抽提物、12 g 细菌用胰蛋白胨和 5 ml 甘油加水到 900 ml;高压灭菌;加入 100 ml 高压灭菌的 0.17 mol/L 磷酸二氢钾和 0.72 mol/L 的磷酸氢二钾混合液,最后加入 5 μg/ml 四环素。
( 6 ) 四环素(tetracycline ): 5 mg/ml 四环素水溶液,超滤灭菌。
( 7 ) 超纯甘油(Invitrogen )。
( 8 ) USP (United States Pharmacopeia) 超纯水(见 12.2.1.2)。
2.2 选择和分析增加融合蛋白展示水平的 P8 变异体
2.2.1 从 HGH- P8 库中选择噬菌体
( 1 ) 0.2% 牛血清白蛋白(BSA) 的 PBS 溶液。
( 2 ) 100 mmol/L HCl。
( 3 ) 1.0 mol/L Tris 碱。
( 4 ) 96 孔 maxisorp 免疫板(NUNC,Roskilde,丹麦)。
( 5 ) 大肠杆菌 E.coli XL-1 Blue 菌株(Stratagene,La Jolla,美国加州)。
( 6 ) PBS-T 缓冲液:PBS 及 0.05% Tween-20。
( 7 ) PBS-T-BSA 缓冲液:PBS,0.05% Tween-20 以及 0.2% BSA。
( 8 ) 2YT/carb/kan 培养基:2YT、50 μg/ml 羧苄青霉素以及 25 μg/ml 卡那霉素。
2.2.2 噬菌体酶联免疫筛选法测定 hGH 的展示水平
( 1 ) 1.0 mol/L H3PO4。
( 2 ) 2YT/carb/kan 培养基(见 12.2.2.1)。
( 3 ) 3,3',5,5'-四甲基联苯胺(TMB) /H2O2 过氧化物酶底物:(Kirkegaard&Perry La boratories Inc, Gaithersburg,MD)。
( 4 ) 96 孔 maxisorp 免疫板(见 12.2.2.1 )。
( 5 ) 羧芐青霉素(carbenicillin,carb) ( 见 12.2.1.1)。
( 6 ) 大肠杆菌 E.coli XL-1 Blue 菌株(见 12.2.2.1 )。
( 7 ) 辣根过氧化物酶(Horseradish peroxidase) /抗 M13 抗体络合物(AmershamPharmacia)。
( 8 ) 卡那霉素(见 12.2.1.1)。
( 9 ) LB/tet 培养皿:LB 琼脂及 5 μg/ml 四环素。
( 10 ) M13KO7 helper 噬菌体(见 12.2.1.1)。
( 11 ) PBS ( 见 12.2.1.1)。
( 12 ) PBS-T 缓冲液(见 12.2.2.1)。
( 13 ) PBS-T-BSA 缓冲液(见 12.2.2.1)。
( 14 ) PEG/NaCl ( 见 12.2.1.1)。
( 15 ) 四环素(见 12.2.1.4)。
4. 注
1. 通过突变引入终止密码子,方案可以缩减为 12.3.2.2 中的 2 ) 和 12.3.2.3 中的 3 ) 中的 1/10。反应产物可以直接用于转化 E.coli。
2. 这一方案可以通过过夜孵育大量产物以及用多次离心柱纯化 ssDNA 进行扩大化。
3. 除退火外,所有步骤都能相应进行扩大化。所述退火方案适用于 250 μl 体积左右。对更大体积也许依然适用,但我们没有测试过。
4. 在随后步骤中,磷酸化的寡核苷酸应该立即使用。
5. 可以通过多个 250 μl 体系退火反应来扩大化这一反应。
6. 洗脱时,加水后在 37°C 孵育 5 min 可以提高 DNA 的洗脱效率。
7. 环状 DNA 的电泳迁移率取决于盐浓度、pH 和 EB 的存在与否。观察图 12.2 中的相对迁移率,DNA 必须在直接加有 EB 的 TAE/琼脂糖胶上进行电泳,切忌将 EB 加到电泳缓冲液中。
8. 立即使用展示库或是加入 10% 甘油储存在 -80°C。冷冻能使有些展示蛋白变性,使之变得不稳定,不利于选择。一般而言,最好尽快使用展示库。
参考文献
1. Smith, G. P. and Petrenko, V. A. ( 1 997) Phage display. 97, 391-410.
2. Sidhu, S S. , Ix>wman, H. R , Cunningham, R C. , and Wells, J. A. (2000) Phage display for selection of novel binding peptides. Methods EnzymoL 328, 333-363.
3. Sidhu, S. S. (2000) Phage display in pharmaceutical biotechnology. Curr. Opin. Biotechnol. 11? Q10-616.
4. Smith, G. P. ( 1 98 5 ) Filamentous fusion phage: novel expression vectors that display cloned antigens on the virion surface. Science 228 ,1315-1317.
5. Bass, S . ,Greene,R . , and Wells,J. A. ( 1 990) Hormone phage: an enrichment method for variant proteins with altered binding properties. Proteins 8, 309-314.
6. Malik, P. , Terry, T. D. , Gowda, L. R. , et al. ( 1 99 6 ) Role of capsid structure and membrane protein pro-cessing in determining the size and copy number of peptides displayed on the major coat protein of filamentous bac-teriophage. J . Mol. Biol. 26 0, 9-21.
7. Iannolo, G. , Minenkova, O. , Petruzzelli, R. , and Cesareni, G. ( 1 99 5 ) Modifying filamentous phage cap-sid: limits in the size of the major capsid protein. J , Mol. Biol. 24 8, 835-844.
8. Kretzschmar, T. and Geiser, M. (1 99 5 ) Evaluation of antibodies fused to minor coat protein III and major coat protein VIII in bacteriophage M13. Gene 155 , 61-65.
9. Clackson, T. and Wells, J. A. ( 1 99 4 ) In vitro selection from protein and peptide libraries. Trends BiotechnoZ. 1 2 ,173-184.
10. Weiss, G. A. , Wells, J. A. , and Sidhu, S. S. (2000) Mutational analysis of the major coat protein of M13 identifies residues that control protein display. Protein Sci.9, 647-654.
11. Sidhu, S. S. , Weiss, G. A. , and Wells, J. A. (2000) High copy display of large proteins on phage for func-tional selections. J , Mol. Biol.296, 487-495.
12. Roth, T. A. , Weiss, G. A. , Eigenbrot, C. , and Sidhu, S. S. (2002) A minimized M l 3 coat protein de-fines the minimum requirements for assembly into the bacte-riophage particle. J . Mol. Biol. 322, 357-367.
13. Kunkel, T. A. , Roberts, J. D. , and Zakour, R. A. ( 1 987) Rapid and efficient site-specific mutagenesis without phe-notypic selection. Methods EnzymoL 154, 367-382.
14. Lechner, R. L. , Engler, M. J. , and Richardson, C. C. ( 1 98 3 ) Characterization of strand displacement synthesis catalyzed by bacteriophage T7 DNA pol3 mierase. J. Biol. Chem. 25 8, 11 7 4 - 118 4 .
15. Miller, J. H. ( 1 972) Experiments in Molecular Biology \st eA, , Cold Spring Harbor Laboratory Press, Cold Spring Harbor, NY, p. 190.
16. Fuh, G . ,Mulkerrin, ML G .,Bass,S , et al. (1 990) The human growth hormone receptor: secretion from Esche-richia coLi and disulfide bonding pattern of the extracellular binding domain. J. Biol. Cherru 265, 3111-3115.
17. Humphrey, W. , Dalke, A. , and Schulten, K. (1 99 6 ) VMD : visual molecular dynamics. J . Mol. Graph. 14 ,27-28,33-38.
18. Esnouf, R. M ( 1 9 9 7 ) An extensively modified version of MolScript that includes greatly enhanced coloring capabilities. J . MoL Graph. Model. 15, 11 2-113 , 13 2-134.
噬菌体展示是一项改造结合靶分子多肽的强大技术 [ 1 〜 3] 。蛋白质与噬菌体衣壳蛋白形成融合蛋白,可以在噬菌体表面表达,而相应的编码基因则包裹于噬菌体颗粒内 [4] 。包裹的 DNA 通过突变扩增构建噬菌体展示多肽库,为筛选各种变异体做准备。通过固定化靶分子体外结合实验,高亲和力的蛋白质可以从蛋白质展示库中筛选出来。随后,选定蛋白质的序列可以通过 DNA 测序推断出来。
靶蛋白可以通过构建噬菌粒载体与主要衣壳蛋白 [ 蛋白质-8 (P8) ] 或次要衣壳蛋白 [ 蛋白质 -3 ( P3 ) ] 形成的融合蛋白在 M13 噬菌体表面以低拷贝方式展示,这需要在野生型(wt) P8 和 P3 辅助噬菌体帮助的条件下进行 [5] 。由于融合蛋白的展示水平随蛋白质序列和长度的不同而不同,多价蛋白与 P8 融合进行展示难以实现 [6] 。例如,如果 P8 的每一个拷贝都与展示多肽形成融合蛋白(噬菌体系统,而不是噬菌粒系统),融合多肽长度超过6 个氨基酸残基,噬菌体一般就会不稳定 [7] 。即使在噬菌粒系统中,蛋白质展示也高度依赖于蛋白质的大小和性质 [8]。尽管用 P3 或 P8 进行单价噬菌体展示使许多不同蛋白质亲和力从中等提高到很高 [9],利用 P8 进行多价噬菌体展示从初级库中选择分子质量大的低亲和力受体蛋白尚未实现。
这里,我们描述一种通过突变 P8 锚定残基改进蛋白质展示的方法。利用这一方法,两个不同的蛋白质 [ ( 链霉亲和素和人生长激素(hGH ) ] 表达水平几乎增加了 100 倍,并且我们发现这一技术也适用于其他蛋白质。这一戏剧性的展示改进很可能由于融合蛋白在噬菌体表面定位更加恰当所致。理论上,高拷贝展示的应用应该可能使结合弱的蛋白质从初级天然库中选择出来,对于小肽这一方法已经成功可行。同样重要的是这一方法扩展了噬菌体展示研究蛋白质的范围,许多曾被认为难以展示的蛋白质用这一方法实现了展示。因此,这里描述的方法增加了噬菌体展示鉴定新蛋白质或修饰蛋白质功能的潜力。
2. 材料
2.1 P8 突变库的构建
2.1.1 制备含有尿嘧啶的单链 DNA 模板
( 1 ) 2YT 培养基:10 g 酵母抽提物、16 g 菌用胰蛋白胨和 5 g 氯化钠;加水到 1 L,用氢氧化钠调节 pH 到 7.0;高压灭菌。
( 2 ) 2YT/carb/cmp 培养基:2YT、50 μg/ml 羧苄青霉素(carbenicillin,carb) 和 5 μg/ml 氯霉素(chloramphenicol,cmp)。
( 3 ) 2YT/carb/kan/uridine 培养基:2YT、50 μg/ml 羧苄青霉素、25 μg/ml 卡那霉素和 0.25 μg/ml 尿嘧啶。
( 4 ) 羧苄青霉素:5 mg/ml,羧苄青霉素水溶液,超滤灭菌。
( 5 ) 氯霉素:50 mg/ml,氯霉素乙醇溶液。
( 6 ) 大肠杆菌 CJ236 ( NEB: New England Biolabs,Beverly,美国麻省)。
( 7 ) 卡那霉素:5 mg/ml,卡那霉素水溶液,超滤灭菌。
( 8 ) 生理磷酸缓冲液(PBS): 137 mmol/L 氯化钠、3 mmol/L 氯化钾、8 mmol/L 磷酸氢二钠以及 1.5 mmol/L 磷酸二氢钾;用盐酸(HCl ) 调节 pH 到 7.2;高压灭菌。
( 9 ) 聚乙二醇(polyethylene glycol,PEG) PEG/NaCl: 20% PEG-8000 ( m/V ) 和 2.5 mol/L 氯化钠;高压灭菌。
( 10 ) QIA prep Spin M13 试剂盒(Qiagen,Valencia,美国加州)。
( 11 ) M13KO7 辅助噬菌体(NEB,Beverly,美国麻省)。
( 12 ) Tris- acetate-EDIA (TAE) 缓冲液:40 mmol/L Tris-acetate 和 1 mmol/L EDTA;调节 pH 到 8.0;高压灭菌。
( 13 ) TAE/琼脂糖胶:TAE 缓冲液、1% 琼脂糖(m/V)以及 1 : 5000 ( V/V ) 10% 溴化乙锭(EB)。
( 14 ) 尿嘧啶:25 mg/ml,尿嘧啶水溶液,超滤灭菌。
2.1.2 体外合成异质共价闭环双链 DNA
( 1 ) 100 mmol/L 二硫苏糖醇(DTT )。
( 2 ) 25 mmol/L dNTP : 含有 dATP、dCTP、dGTP 和 dTTP 各 25 mmol/L 的溶液 ( Amersham-Pharmacia,Piscataway,美国新泽西州)。
( 3 ) 10 mmol/L ATP (Amersham-Pharmacia)。
( 4 ) 10 X Tris-Mg (TM) 缓冲液:0.1 mol/L MgCl2 以及 0.5 mol/L TrisHCl,pH 7.5。
( 5 ) QIAquick Gel Extraction 试剂盒(Qiagen,Valencia, 美国加州)。
( 6 ) T4 多聚核苷酸激酶(NEB: New England Biolabs) 。
( 7 ) T4 DNA 连接酶(Invitrogen, Carlsbad ,美国加州)。
( 8 ) TAE 琼脂糖凝胶(见 12.2.1.1 ) 。
( 9 ) USP ( United States Pharmacopeia ) 超纯水(Braun Medical In& , Irvine, 美国力口州)。
2.1.3 大肠杆菌电穿孔及噬菌体扩增
( 1 ) 2YT/carb 培养基:2YT 及 50 μg/ml 羧芐青霉素。
( 2 ) 羧芐青霉素(carbenicillin,carb) ( 见 12.2.1.1)。
( 3 ) 电转化感受态 E.coli SS320 ( 见 12.3.2.4)。
( 4 ) 0.2 cm 间隙电转化样品杯(BTX,圣地亚哥,美国加州)。
( 5 ) ECM-600 电转化仪(BTX )。
( 6 ) 卡那霉素 ( 见 12.2.1.1) 。
( 7 ) Luria-Bertani (LB) /carb 培养皿:LB 琼脂及 50 μg/ml 羧苄青霉素。
( 8 ) PBS ( 见 12.2.1.1)。
( 9 ) PEG/NaCl ( 见 12.2.1.1)。
( 10 ) SOC 培养基:5 g 细菌用酵母抽提物、20 g 细菌用胰蛋白胨和 0.5 g 氯化钠及 0.2 g 氯化钾;加水到大约1 L,用氢氧化钠调节 pH 到 7.0;高压灭菌;然后加入 5 ml 2.0 mol/L 高压灭菌过的氯化镁溶液及 20 ml 超滤灭菌过的 1.0 mol/L 葡萄糖溶液;并将最终体积调至 1 L。
( 11 ) M13KO7 辅助噬菌体(见 12.2.1.1)。
2.1.4 制备电穿孔法感受态细胞
( 1 ) 1.0 mmol/L HEPES,pH 7.4 : 将 4.0 ml 1.0 mol/L 的 HEPES,pH 7.4 加入 4.0 L 的 USP (U nited States Pharmacopeia) 超纯水,超滤灭菌。
( 2 ) 10% (V/V ) 的超纯甘油:100 ml 超纯甘油加入 900 ml USP 超纯水中,超滤灭菌。
( 3 ) 2YT/tet 培养基:2YT 及 5 μg/ml 四环素。
( 4 ) 磁性搅拌棒(约 5 cm 长),放入乙醇中浸泡。
( 5 ) Superbroth/tet 培养基:24 g 细菌用酵母抽提物、12 g 细菌用胰蛋白胨和 5 ml 甘油加水到 900 ml;高压灭菌;加入 100 ml 高压灭菌的 0.17 mol/L 磷酸二氢钾和 0.72 mol/L 的磷酸氢二钾混合液,最后加入 5 μg/ml 四环素。
( 6 ) 四环素(tetracycline ): 5 mg/ml 四环素水溶液,超滤灭菌。
( 7 ) 超纯甘油(Invitrogen )。
( 8 ) USP (United States Pharmacopeia) 超纯水(见 12.2.1.2)。
2.2 选择和分析增加融合蛋白展示水平的 P8 变异体
2.2.1 从 HGH- P8 库中选择噬菌体
( 1 ) 0.2% 牛血清白蛋白(BSA) 的 PBS 溶液。
( 2 ) 100 mmol/L HCl。
( 3 ) 1.0 mol/L Tris 碱。
( 4 ) 96 孔 maxisorp 免疫板(NUNC,Roskilde,丹麦)。
( 5 ) 大肠杆菌 E.coli XL-1 Blue 菌株(Stratagene,La Jolla,美国加州)。
( 6 ) PBS-T 缓冲液:PBS 及 0.05% Tween-20。
( 7 ) PBS-T-BSA 缓冲液:PBS,0.05% Tween-20 以及 0.2% BSA。
( 8 ) 2YT/carb/kan 培养基:2YT、50 μg/ml 羧苄青霉素以及 25 μg/ml 卡那霉素。
2.2.2 噬菌体酶联免疫筛选法测定 hGH 的展示水平
( 1 ) 1.0 mol/L H3PO4。
( 2 ) 2YT/carb/kan 培养基(见 12.2.2.1)。
( 3 ) 3,3',5,5'-四甲基联苯胺(TMB) /H2O2 过氧化物酶底物:(Kirkegaard&Perry La boratories Inc, Gaithersburg,MD)。
( 4 ) 96 孔 maxisorp 免疫板(见 12.2.2.1 )。
( 5 ) 羧芐青霉素(carbenicillin,carb) ( 见 12.2.1.1)。
( 6 ) 大肠杆菌 E.coli XL-1 Blue 菌株(见 12.2.2.1 )。
( 7 ) 辣根过氧化物酶(Horseradish peroxidase) /抗 M13 抗体络合物(AmershamPharmacia)。
( 8 ) 卡那霉素(见 12.2.1.1)。
( 9 ) LB/tet 培养皿:LB 琼脂及 5 μg/ml 四环素。
( 10 ) M13KO7 helper 噬菌体(见 12.2.1.1)。
( 11 ) PBS ( 见 12.2.1.1)。
( 12 ) PBS-T 缓冲液(见 12.2.2.1)。
( 13 ) PBS-T-BSA 缓冲液(见 12.2.2.1)。
( 14 ) PEG/NaCl ( 见 12.2.1.1)。
( 15 ) 四环素(见 12.2.1.4)。
4. 注
1. 通过突变引入终止密码子,方案可以缩减为 12.3.2.2 中的 2 ) 和 12.3.2.3 中的 3 ) 中的 1/10。反应产物可以直接用于转化 E.coli。
2. 这一方案可以通过过夜孵育大量产物以及用多次离心柱纯化 ssDNA 进行扩大化。
3. 除退火外,所有步骤都能相应进行扩大化。所述退火方案适用于 250 μl 体积左右。对更大体积也许依然适用,但我们没有测试过。
4. 在随后步骤中,磷酸化的寡核苷酸应该立即使用。
5. 可以通过多个 250 μl 体系退火反应来扩大化这一反应。
6. 洗脱时,加水后在 37°C 孵育 5 min 可以提高 DNA 的洗脱效率。
7. 环状 DNA 的电泳迁移率取决于盐浓度、pH 和 EB 的存在与否。观察图 12.2 中的相对迁移率,DNA 必须在直接加有 EB 的 TAE/琼脂糖胶上进行电泳,切忌将 EB 加到电泳缓冲液中。
8. 立即使用展示库或是加入 10% 甘油储存在 -80°C。冷冻能使有些展示蛋白变性,使之变得不稳定,不利于选择。一般而言,最好尽快使用展示库。
参考文献
1. Smith, G. P. and Petrenko, V. A. ( 1 997) Phage display. 97, 391-410.
2. Sidhu, S S. , Ix>wman, H. R , Cunningham, R C. , and Wells, J. A. (2000) Phage display for selection of novel binding peptides. Methods EnzymoL 328, 333-363.
3. Sidhu, S. S. (2000) Phage display in pharmaceutical biotechnology. Curr. Opin. Biotechnol. 11? Q10-616.
4. Smith, G. P. ( 1 98 5 ) Filamentous fusion phage: novel expression vectors that display cloned antigens on the virion surface. Science 228 ,1315-1317.
5. Bass, S . ,Greene,R . , and Wells,J. A. ( 1 990) Hormone phage: an enrichment method for variant proteins with altered binding properties. Proteins 8, 309-314.
6. Malik, P. , Terry, T. D. , Gowda, L. R. , et al. ( 1 99 6 ) Role of capsid structure and membrane protein pro-cessing in determining the size and copy number of peptides displayed on the major coat protein of filamentous bac-teriophage. J . Mol. Biol. 26 0, 9-21.
7. Iannolo, G. , Minenkova, O. , Petruzzelli, R. , and Cesareni, G. ( 1 99 5 ) Modifying filamentous phage cap-sid: limits in the size of the major capsid protein. J , Mol. Biol. 24 8, 835-844.
8. Kretzschmar, T. and Geiser, M. (1 99 5 ) Evaluation of antibodies fused to minor coat protein III and major coat protein VIII in bacteriophage M13. Gene 155 , 61-65.
9. Clackson, T. and Wells, J. A. ( 1 99 4 ) In vitro selection from protein and peptide libraries. Trends BiotechnoZ. 1 2 ,173-184.
10. Weiss, G. A. , Wells, J. A. , and Sidhu, S. S. (2000) Mutational analysis of the major coat protein of M13 identifies residues that control protein display. Protein Sci.9, 647-654.
11. Sidhu, S. S. , Weiss, G. A. , and Wells, J. A. (2000) High copy display of large proteins on phage for func-tional selections. J , Mol. Biol.296, 487-495.
12. Roth, T. A. , Weiss, G. A. , Eigenbrot, C. , and Sidhu, S. S. (2002) A minimized M l 3 coat protein de-fines the minimum requirements for assembly into the bacte-riophage particle. J . Mol. Biol. 322, 357-367.
13. Kunkel, T. A. , Roberts, J. D. , and Zakour, R. A. ( 1 987) Rapid and efficient site-specific mutagenesis without phe-notypic selection. Methods EnzymoL 154, 367-382.
14. Lechner, R. L. , Engler, M. J. , and Richardson, C. C. ( 1 98 3 ) Characterization of strand displacement synthesis catalyzed by bacteriophage T7 DNA pol3 mierase. J. Biol. Chem. 25 8, 11 7 4 - 118 4 .
15. Miller, J. H. ( 1 972) Experiments in Molecular Biology \st eA, , Cold Spring Harbor Laboratory Press, Cold Spring Harbor, NY, p. 190.
16. Fuh, G . ,Mulkerrin, ML G .,Bass,S , et al. (1 990) The human growth hormone receptor: secretion from Esche-richia coLi and disulfide bonding pattern of the extracellular binding domain. J. Biol. Cherru 265, 3111-3115.
17. Humphrey, W. , Dalke, A. , and Schulten, K. (1 99 6 ) VMD : visual molecular dynamics. J . Mol. Graph. 14 ,27-28,33-38.
18. Esnouf, R. M ( 1 9 9 7 ) An extensively modified version of MolScript that includes greatly enhanced coloring capabilities. J . MoL Graph. Model. 15, 11 2-113 , 13 2-134.