PEAKS蛋白质从头测序技术
北京百泰派克生物科技有限公司
2126
2003年,Ma等人开发了PEAKS。PEAKS是用于解析肽图谱的一种串联质谱蛋白质组学软件,可用于基于串联质谱的肽测序,蛋白质鉴定和蛋白质定量分析。他们在PEAKS方法中运用了新的从头测序模型和算法,分为四个步骤:
第一步将图谱进行预处理,包括图谱噪声过滤和图谱峰聚合;
第二步是在简化后的图谱中根据母离子质量列举出所有可能的候选肽段;
第三步和第四步为综合打分的差异分析以及分值正则化。
PEAKS蛋白质从头测序技术一般通过肽从头测序辅助的数据库搜索来进行肽鉴定。同时,它还通过基于肽序列标签的自动搜索(SPIDER)和PTM鉴定技术整合了PTM和突变表征。 PEAKS可提供各种肽的完整序列,各个氨基酸序列的可信度评分,简单的高通量分析报告,以及其他信息。
PEAKS可以对多个数据库搜索引擎的结果进行比较。PEAKS inChorus会自动将测试结果同其他蛋白质ID搜索引擎(例如Sequest,OMSSA,X!Tandem和Mascot)进行交叉检查,这样可防止肽鉴定结果的假阳性。
PEAKS Q是一种用于蛋白质定量的附加工具,支持标记(ICAT, iTRAQ, SILAC, TMT, 018等)和非标记两种定量法。
其中SPIDER是PEAKS中基于序列标签的搜索工具,可处理从头测序误差和同源突变之间可能出现的重叠部分。它可通过自动且有效地结合从头序列标签和同源物,重建正确的肽序列。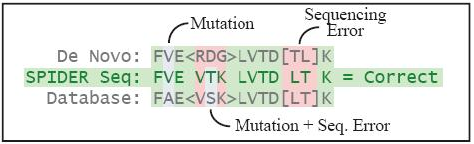
SPIDER 重建正确的肽序列过程(来源:百泰派克)
PEAKS软件中使用的一系列算法已经过修改配置到PEAKS AB软件中,成为用于自动单克隆抗体测序公认的首选方法。
第一步将图谱进行预处理,包括图谱噪声过滤和图谱峰聚合;
第二步是在简化后的图谱中根据母离子质量列举出所有可能的候选肽段;
第三步和第四步为综合打分的差异分析以及分值正则化。
PEAKS蛋白质从头测序技术一般通过肽从头测序辅助的数据库搜索来进行肽鉴定。同时,它还通过基于肽序列标签的自动搜索(SPIDER)和PTM鉴定技术整合了PTM和突变表征。 PEAKS可提供各种肽的完整序列,各个氨基酸序列的可信度评分,简单的高通量分析报告,以及其他信息。
PEAKS可以对多个数据库搜索引擎的结果进行比较。PEAKS inChorus会自动将测试结果同其他蛋白质ID搜索引擎(例如Sequest,OMSSA,X!Tandem和Mascot)进行交叉检查,这样可防止肽鉴定结果的假阳性。
PEAKS Q是一种用于蛋白质定量的附加工具,支持标记(ICAT, iTRAQ, SILAC, TMT, 018等)和非标记两种定量法。
其中SPIDER是PEAKS中基于序列标签的搜索工具,可处理从头测序误差和同源突变之间可能出现的重叠部分。它可通过自动且有效地结合从头序列标签和同源物,重建正确的肽序列。

SPIDER 重建正确的肽序列过程(来源:百泰派克)