想要发高分文章吗?分享一个来自 nature 的灵感
丁香园
2012 年,一篇 nature 文章,巧妙的整合了来自「Agilent mRNA 芯片、Illumina DNA 甲基化芯片、Affymetrix SNP 芯片、miRNA 测序、全外显子组测序以及反相蛋白阵列技术」六个不同技术的数据,对原发性乳腺癌进行了全面的分析[1]。
多组学整合分析:研究不能只满足于单一尺度
Nature 的这个案例,却是在肿瘤及其他复杂疾病研究中的一个常见研究思路,即用系统生物学的方法整合不同组学之间的数据,包括基因组、表观基因组、转录组、表观转录组、蛋白质组,来揭示肿瘤以及复杂疾病的发病机制、发生发展过程以及用药后的预后评估。鉴于复杂的多组学数据和甲基化测序的昂贵费用,高性价比的甲基化芯片(850 K),已然成为 TCGA 肿瘤研究中常用的 DNA 甲基化研究手段。

表观基因组全关联分析:站在 GWAS 肩膀上的新策略
对于复杂疾病,除了多组学整合分析,全基因组关联研究(GWAS)也为复杂疾病的致病基因定位提供了强有力的手段。但是越来越多的研究表明,复杂疾病的致病原因是多样化的,基因组遗传信息的变异是导致复杂疾病的重要因素之一,除此之外,表观遗传学的变异在复杂疾病的发生发展中也起到很重要的作用。
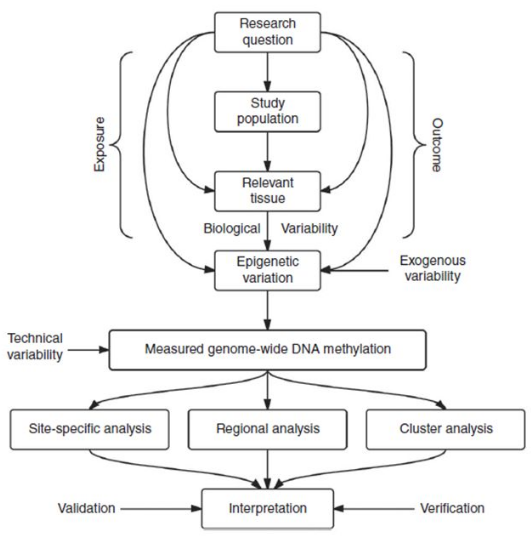
GWAS 所采用的研究策略仅仅局限于遗传信息的变异,而无法在表观遗传学层面进行复杂疾病的解读。表观基因组全关联分析(EWAS)技术的出现弥补了 GWAS 的不足,将表观遗传学变异和复杂疾病进行关联,在表观遗传学层面对复杂疾病的致病原因进行解读,找到与致病原因相关的表观遗传学变异位点。
EWAS 为研究者打开了一扇通往研究复杂疾病的新道路,将在患者全基因组范围内检测出的甲基化变异位点与对照组进行比较,找出所有甲基化位点的变异频率,从而避免了像候选基因策略一样需要预先假设致病基因。同时,EWAS 研究让我们找到了许多从前未曾发现的与疾病相关的甲基化位点,为复杂疾病的发病机制在表观层面上提供更多的线索。