做数据挖掘,就算发 20 几分的 CNS 子刊,也是垃圾?
丁香园
关于数据挖掘发表文章,我们知道很多人是看不上、瞧不起、嗤之以鼻的。大抵是因为这些人平时只发 CNS 主刊,所以才认为通过数据挖掘这种用「别人的数据」或者叫「干实验」来发文章是「垃圾」,没有什么价值。
真的是这样吗?今天我们要介绍的就是一篇做数据挖掘的 Cancer Cell 杂志的文章(IF: 27.4),大家来看看文章怎么样。

1 文章数据挖掘的情况
肿瘤类型:妇科肿瘤和乳腺癌;
数据来源:主要是 TCGA 数据库,1,087 例 BRCA(invasive breast carcinoma,乳腺癌), 308 例 CESC (cervical squamous cell carcinoma and endocervical adenocarcinoma, 宫颈癌 ), 579 例 OV(high-grade serous ovarian cystadenocarcinoma,卵巢癌), 548 例 UCEC(uterine corpus endometrial carcinoma,子宫内膜子宫内膜癌)和 57 例 UCS(uterine carcinosarcoma ,子宫癌肉瘤),共 2,579 例,统称为 “Pan-Gyn” 泛妇科肿瘤。
数据类型:临床信息(clinical), 拷贝数变异(somatic
copy-number alterations SCNAs), 突变(mutations), DNA甲基化(DNA methylation),mRNA,miRNA,lncRNA和蛋白的表达(expression of mRNA, microRNA, long non-coding RNA, and proteins)。
2 文章的研究工作
1. 找到了分子特征(molecular features),用以区分 “Pan-Gyn” 与 TCGA 中其它肿瘤;

2. 鉴定到高白细胞浸润(high leukocyte infiltration)这一免疫应答的肿瘤亚型;

3. 建立了基因和 lncRNA 的相互作用 network(interaction network );
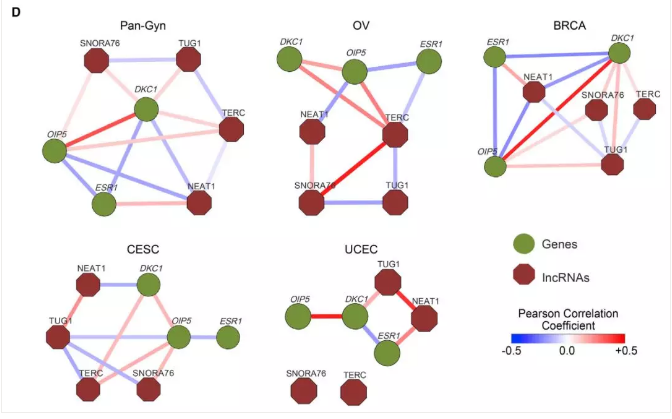
4. 建立了决策树(Decision tree),将临床相关预后的肿瘤亚型进行再分组;
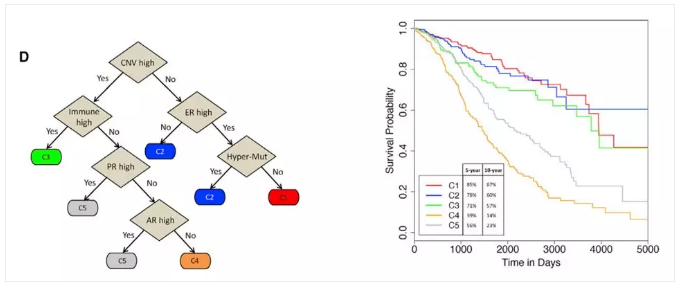
由于内容比较多,这篇文章我们就简单介绍到这里。
3 趋势文章
细心的同学会发现 Pubmed 的趋势文章(Trending Articles),最近有很多从各个角度分析 TCGA 数据的高分文章。
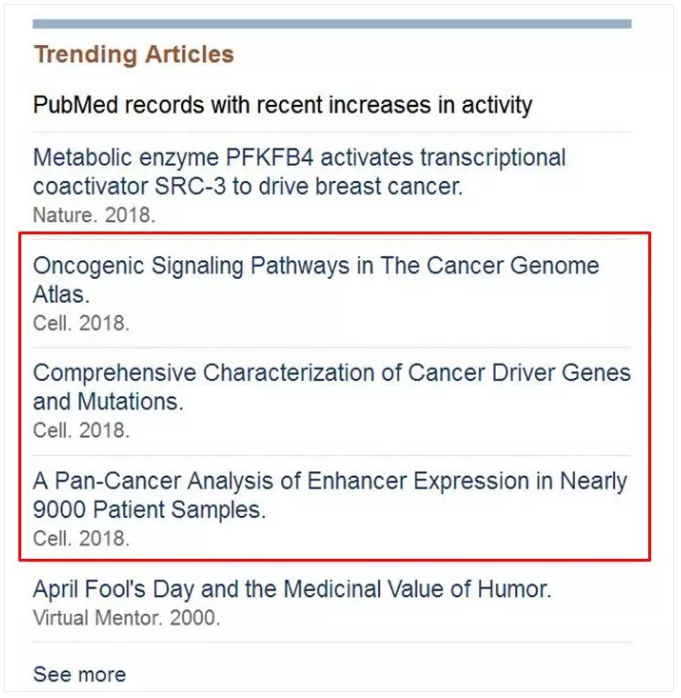
比如 4 月 5 日 Cell 主刊的六连发:
1. 分析泛肿瘤中增强子(Enhancer)表达:
A Pan-Cancer Analysis of Enhancer Expression in Nearly 9000 Patient Samples.Cell. 2018 Apr 5;173(2):386-399.e12.

2. 分析肿瘤驱动(Driver )基因和突变:
Comprehensive Characterization of Cancer Driver Genes and Mutations.Cell. 2018 Apr 5;173(2):371-385.e18.

3. 分析肿瘤信号通路:
Oncogenic Signaling Pathways in The Cancer Genome Atlas.Cell. 2018 Apr 5;173(2):321-337.e10.

4. 分析患者生存预后结果的
An Integrated TCGA Pan-Cancer Clinical Data Resource to Drive High-Quality Survival Outcome Analytics.Cell. 2018 Apr 5;173(2):400-416.e11.

5. 分析肿瘤发病生殖系变异(Pathogenic Germline Variants):
Pathogenic Germline Variants in 10,389 Adult Cancers.Cell. 2018 Apr 5;173(2):355-370.e14.

6. 分析细胞来源用于肿瘤分类:
Cell-of-Origin Patterns Dominate the Molecular Classification of 10,000 Tumors from 33 Types of Cancer.Cell. 2018 Apr 5;173(2):291-304.e6.

又比如 4 月 3 日 Cell Reports 的五连发:
1. 从DNA损伤修复角度分析基因组和分子图谱:
Genomic and Molecular Landscape of DNA Damage Repair Deficiency across The Cancer Genome Atlas.Cell Rep. 2018 Apr 3;23(1):239-254.e6.

2. 分析肾癌整体分子特性:
The Cancer Genome Atlas Comprehensive Molecular Characterization of Renal Cell Carcinoma.Cell Rep. 2018 Apr 3;23(1):313-326.e5.

3. 分析鳞癌的基因组、通路和免疫特性:
Genomic, Pathway Network, and Immunologic Features Distinguishing Squamous Carcinomas.Cell Rep. 2018 Apr 3;23(1):194-212.e6.

4. 从泛素化通路角度分析:
Integrated Genomic Analysis of the Ubiquitin Pathway across Cancer Types.Cell Rep. 2018 Apr 3;23(1):213-226.e3.

5. 从lncRNA角度分析,并通过实验验证:
Pan-Cancer Analysis of lncRNA Regulation Supports Their Targeting of Cancer Genes in Each Tumor Context.Cell Rep. 2018 Apr 3;23(1):297-312.e12.