学会这个方法,5 分钟找到合适的 qPCR 引物
丁香园
师姐说:师妹啊,你帮我找几个基因的 qPCR 引物吧。你问怎么找?师姐说查文献啊!于是你一头雾水的开始查文献:打开 Pubmed,输入基因名,ATG7,enter,哇,有几万条记录,点下 best match,再选下近 5 年的文献,筛选一下,哇,还有几万篇相关文章。
好吧,那就选第一篇看吧,点进去,找全文,往下拖,找到材料和方法,找啊找,终于找到了,那就 copy。还挺幸运的,可是,一看时间,半个小时过去了,这才查了一个基因的,还有好几个,一个晚上又没了,心里默念:师姐真坑!
终于在今天结束前将所有引物都发给了师姐,明天就可以跟师姐一起愉快的跑 qPCR 了。
过了两天,师姐一脸不高兴的说:师妹你在哪找的引物啊?跑出来的都是什么鬼?
你一看溶解曲线是双峰,心里也很委屈,嘀咕到:你不是说在文献里面找吗?我就是在文献里面找的。
师姐问:多少分的文献?你默默的打开 pubmed,找到昨天找的文献,一查 IF 3 分不到,你默默的不敢说话了!
设计 qPCR 引物,一种方法就够了,省时省力,关键还靠谱,再也不会让师姐失望了!
1. 首先还是打开 pubmed,选择 gene, 输入基因名 ATG7,点 search。

2. 选择正确的基因名和物种,记住基因名下面的那串数字,也就是 gene ID,也可以直接 copy 这串数字。

3. 打开 primer bank。网址是:
https://pga.mgh.harvard.edu/primerbank/,
在 search by 的地方选择 NCBIGene ID,Species 那里选择相应的物种,将上一步 copy 的数字放在 For text 里面,然后点 Submit。
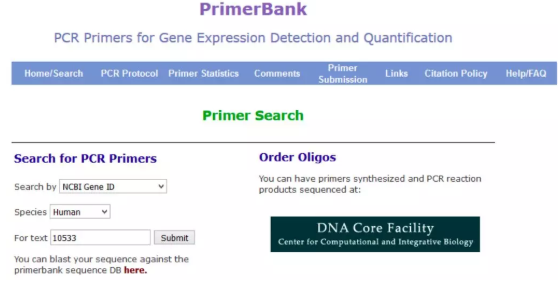
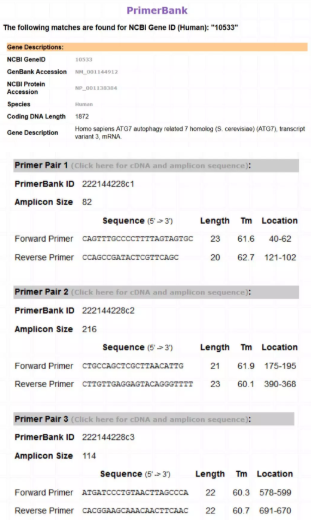
4. 出来了,在 Gene Description 那里再对一下基因信息,确定是自己想找的基因后,在几对引物中选择自己看着顺眼的那对拿去合成就好了。
开玩笑啦,还是有选择标准的:首先看看 Amplicon Size,也就是扩增长度,建议在 100~300bp 间比较好扩增;引物长度在 20~25bp 较好,而且上下游引物间不要相差超过 5bp;再就是看看 Tm 值,60℃左右,相差不要超过 1℃。
综上,将选择好的引物(这里我选择 Primer Pair 3),进一步验证,这里很友好,支持复制粘贴。
5. 还是回到 Pubmed,打开里面的 primerblast。网址是:
https://www.ncbi.nlm.nih.gov/tools/primer-blast/index.cgi?LINK_LOC=BlastHome) 。
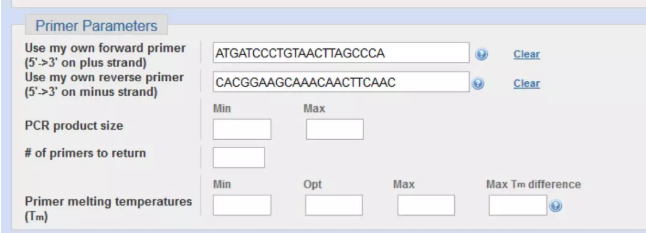
将选中的引物直接 copy 到相应的位置。
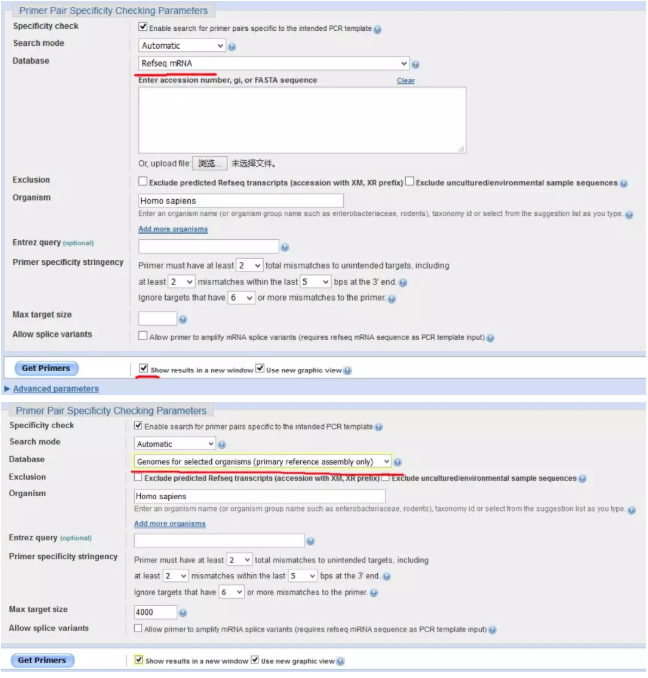
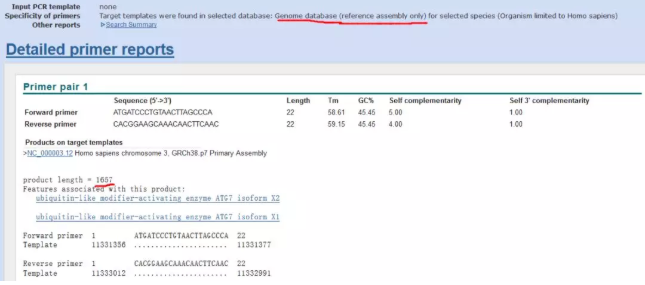
在 Database 里选 Refseq mRNA,在 Get Primer 后的第一个小方框上也打个勾,再点 Get Primer,这样结果就会显示在新的标签页。
接着再在 Database 里选 Genomes for selected organisms(primary referenceassembly only),点击 Get Primer。
得到结果:在 Refseq mRNA 的数据库里面我们得到的就是这对引物能扩增出什么产物来,一看结果都是 ATG7,只是有不同的转录本,扩增产物长度也一样,没关系,反正扩出来都是 ATG7。
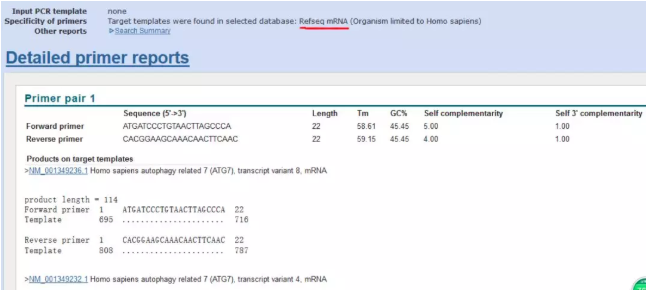
在 Genomes for selected organisms(primary referenceassembly only)里查找的目的是看看是否用该对引物会扩增出非特异性的产物,没有是最好的,说明引物很特异;如果有,也不要着急,看看到底扩增出来什么?
诶,好像不是我想要的基因,是不是不好啊?不要着急,看看 Product length:1657bp,放心吧,这么长的产物,qPCR 是扩增不出来的(一般 500bp 就比较难扩增出来了)。
所以结合以上判定方法,你选的引物基本上就能跑出漂亮的溶解曲线了,为什么不说百分之一百,因为这还取决于你提取的 RNA 合格不!
上面介绍的这种查找 qPCR 的引物的方法在我这里是屡试不爽,介绍给实验室的同学和师弟师妹们用也是深受好评,primer bank 里面同一个基因引物基本不会超过 3 对,只要你不是选择困难症患者,整个操作过程 5 分钟就能搞定。
但是友情提醒下,blast 真的很慢,所以等你对 primer bank 有了十足的信心之后,你基本不会再借用 blast 来判断你选的引物好不好,结合扩增产物长度、引物长度和 Tm 值就能愉快的三选一或二选一了,这又为你节约了 2 分钟,是不是很开心!