如何使用 GSEA 进行数据分析?
丁香园
在本篇文章中,将利用 GSEA 官网提供的数据集,介绍如何使用该软件进行分析数据。
一、打开软件,在软件左上角 Download Example datasets,或者直接去该网站进行下载。
http://www.gsea-msigdb.org/gsea/datasets.jsp
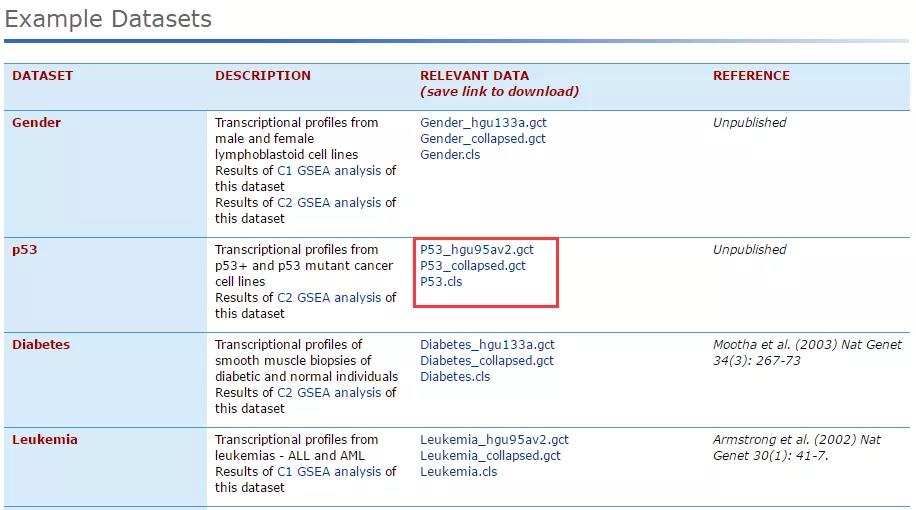
在这里,我们将使用 p53 的数据进行分析,将图中红框的三个文件都下载到本地,这三个文件为携带 p53 突变与野生型表达谱、表型文件。在下载数据的时候,首先需要先进行注册,注册只需要邮箱,很简单就能完成。
二、下载完成之后,再回到软件首页,在软件左上角,点击 load data,将刚才下载的三个文件上传。
注意:上传的数据在格式上是有要求的,这个在 GSEA 官网上有说明
点击 load data 之后,右边的界面会出现下图所示画面:
1. 通过浏览电脑本地文件夹上传文件;
2. 上传最近使用过的数据(第一次使用时不会用到这个方法上传数据);
3. 直接将数据直接拖过来;
4. 对上传的数据类型的具体格式有详细的要求,因此要按照软件要求格式上传数据;

我是直接将下载的数据直接拖进来,然后点击「Load these files!」,之后软件会对将要上传的数据进行检验,除非按照软件要求的格式,否则会报错。

上传成功之后,就会在该界面看到导入的数据:

三、数据准备好之后,下一步就是要进行分析本批次数据了。点击 Run GSEA,此时右边界面会有很多选项,然后根据你的实际情况进行填写就好了。
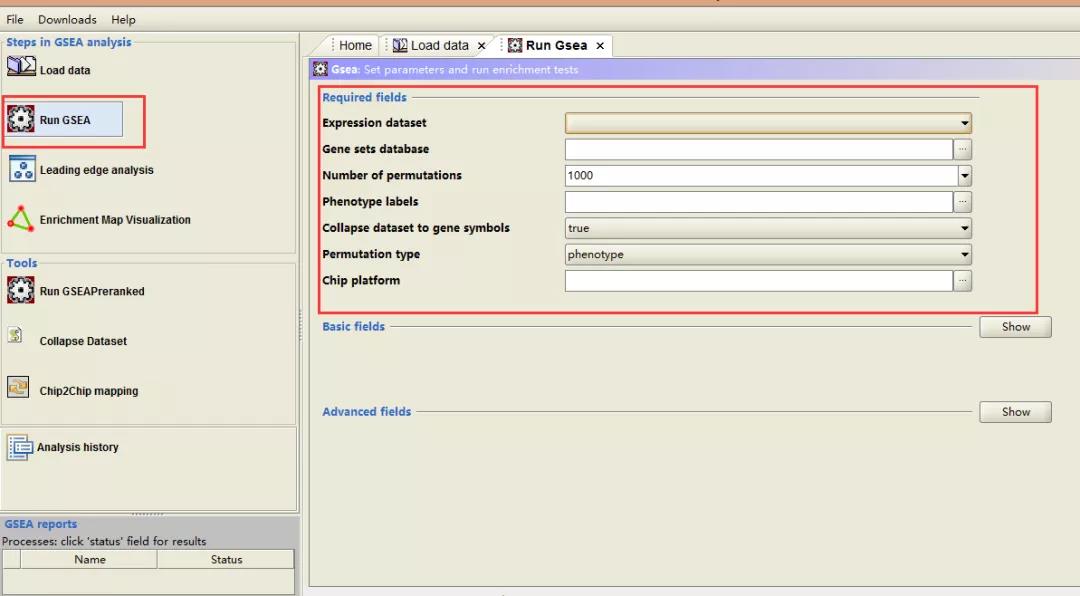

具体参数设置根据提示进行选择:
1. 表达数据集,在这里为之前下载的 p53_hgu95av2 dataset ;
2. 根据自己数据的情况,点击右边小红框,选择合适的数据集(网络或者本地),这里选择的是 Hallmark;
3. 第一次运行的时候,先用一个小的数,这里选用 5,等成功运行之后可以换成较大的数字;
4. 根据实际情况,点击右边小红框选择想要比较的表型,这里选择 MUT VS WT;
5. 如果上传的表达数据集基因 ID 为测序平台原始的 ID,这里就选择「true」;如果表达数据集为基因名称(gene symbols),那就不需要进行转换,此时选择「false」;
6. 在上传的数据集中,如果每组样本数较多(>= 8),此处选择「phenotype」;如果样本数较少,选择「gene_set」;
7. 如果上传的表达数据集基因 ID 为测序平台原始的 ID,如果第 5 项选择的「true」,那么在此处需要点击右边的小红框选择对应的 chip platform,使得原始 ID 转换为 gene symbols,后者为软件可识别的 ID。
在这些参数设置好之后就可以点击下方的「run」运行。
四、运行过程中,在软件的左下角会显示运行状态,屏幕中间显示完成程度:
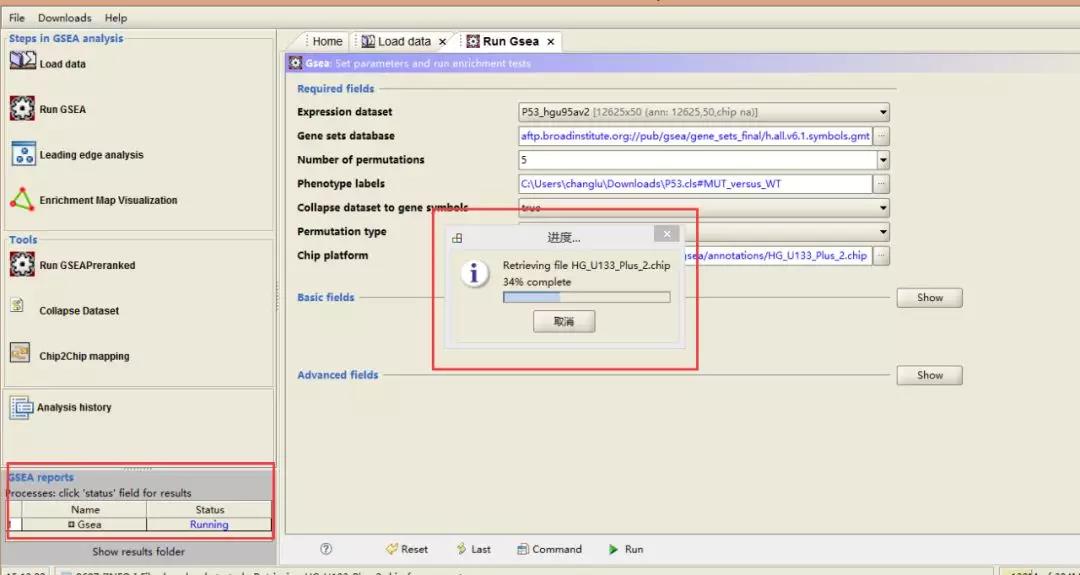
五、运行成功之后,点击左下角状态一栏的「success」,然后进入结果分析页面。当出现「success」之后,说明运行成功,但是也会出现「Error」,此时,你就要根据自己数据特点检查各个参数选择是否正确。
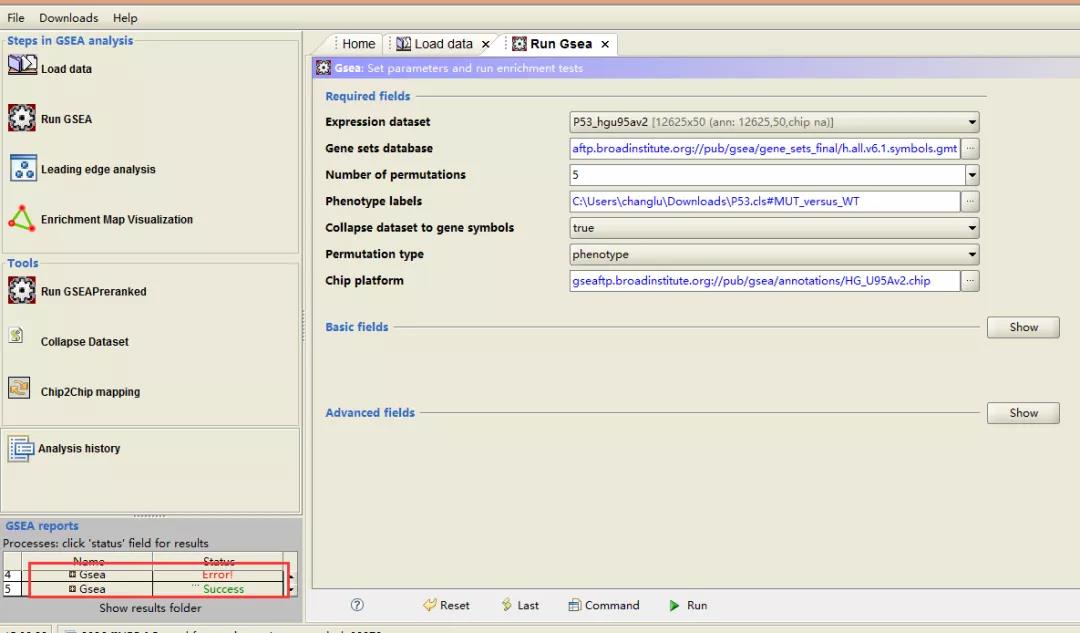
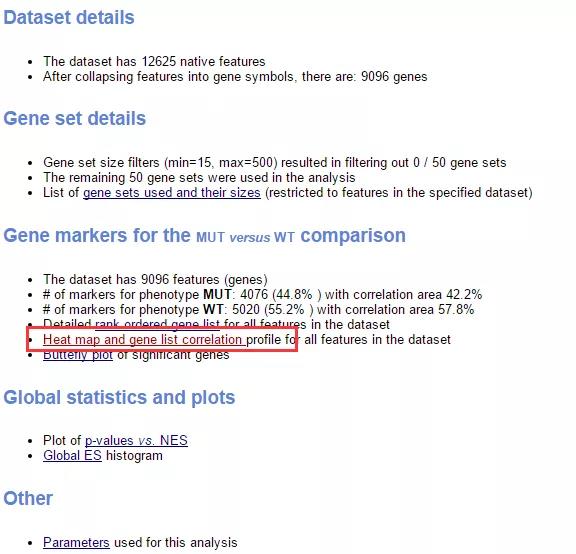
六、联网状态下,点击 success 就会转到结果报告页面,在这里有详细的解读,其中 Snapshot 超链接是图形,点开之后就会看到想要的画面了,可以分别点开每一个图进行查看是否为预期中的结果。
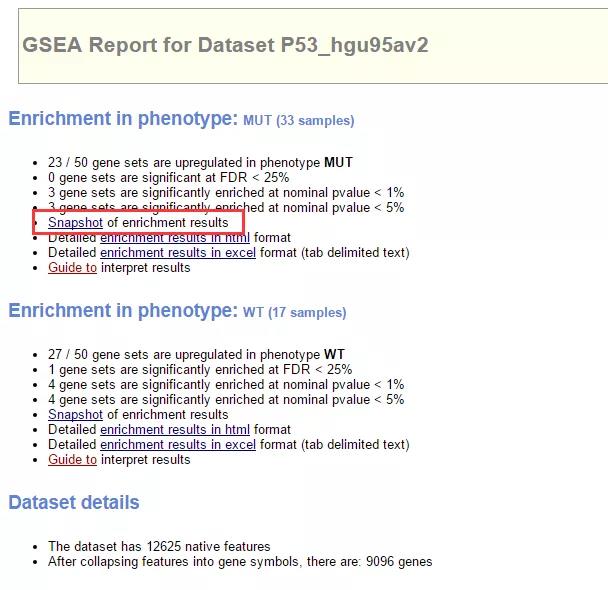
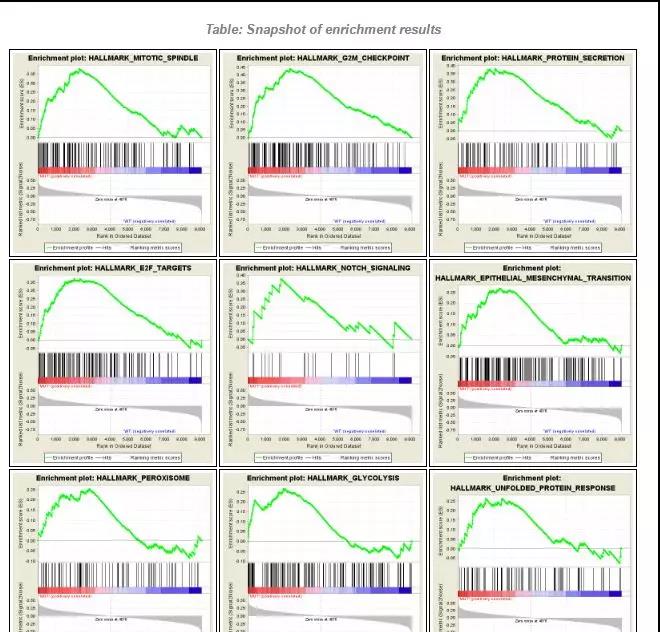
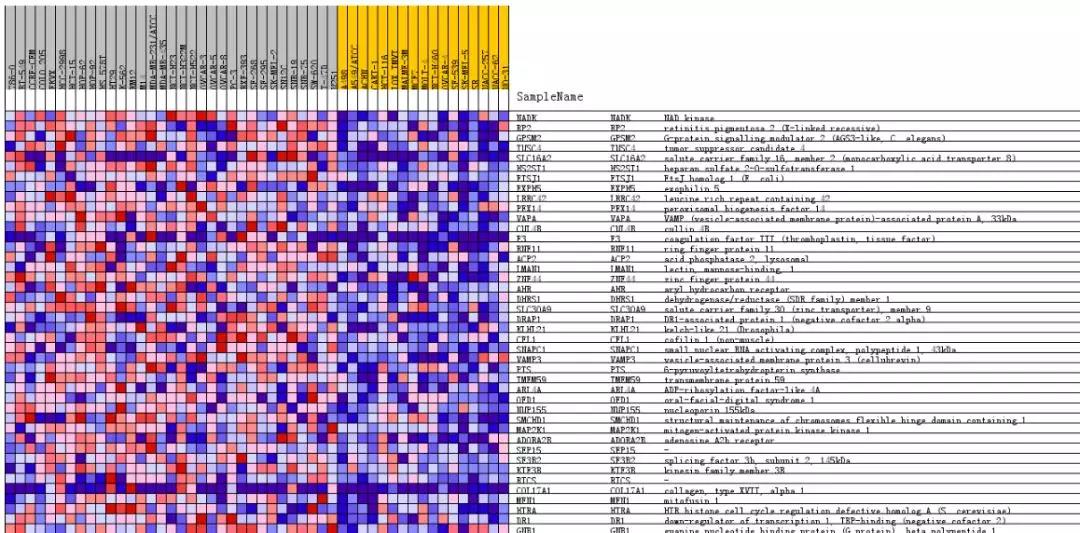
结果报告页面的下半部分,还会看到 MUT 与 WT 两者 marker 的热图和基因列表:
到这里,GSEA 的基本分析就完成了,希望可以对你有所帮助!