3 种方法注释你的甲基化探针
生信技能树
甲基化芯片背景
甲基化芯片原理:https://www.jianshu.com/p/c4f758e0399d
芯片主要分为 EPIC 和 450k 两种,EIPC 也就是 850k,两种探针的都是以 cg 开头的数字编号,所谓注释也就是提取这些探针的所对应的信息,例如,探针序列的 CpG 位置信息,对应的基因信息,染色体上的位置信息等等。很多包在安装的时候都会自动下载这些注释信息,并包装在一起,如果我们想要自己注释这些探针,就要考虑如何获取独立的注释信息。而所需要注释数据的,大部分都来自于两个数据库,GEO 和 TCGA。
下面介绍三种提取注释信息的方法
方法一:从 UCSC Xena 下载
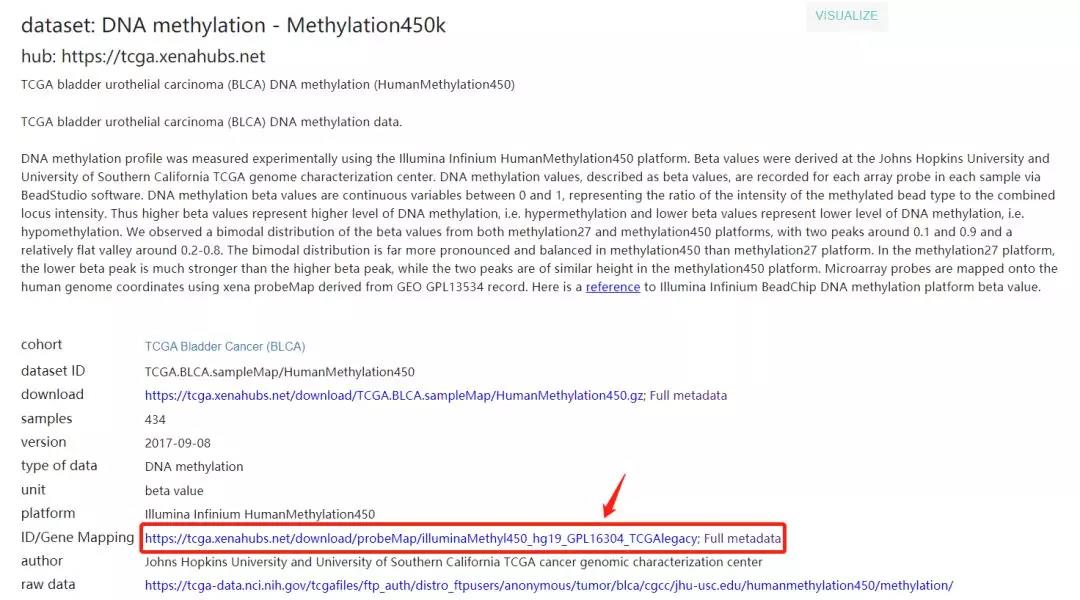
直接从 UCSC Xena 相应的癌症甲基化数据库里下载对应的文件。可以看到是来自 GPL16304 平台的芯片,其实和下面要介绍的从 GEO 下载注释信息是一样的,不过 TCGA 的探针数可能会少于 45w,大约 39w,因为提前过滤了一些低质量的探针。
方法二:从 GEO 下载对应平台的注释文件
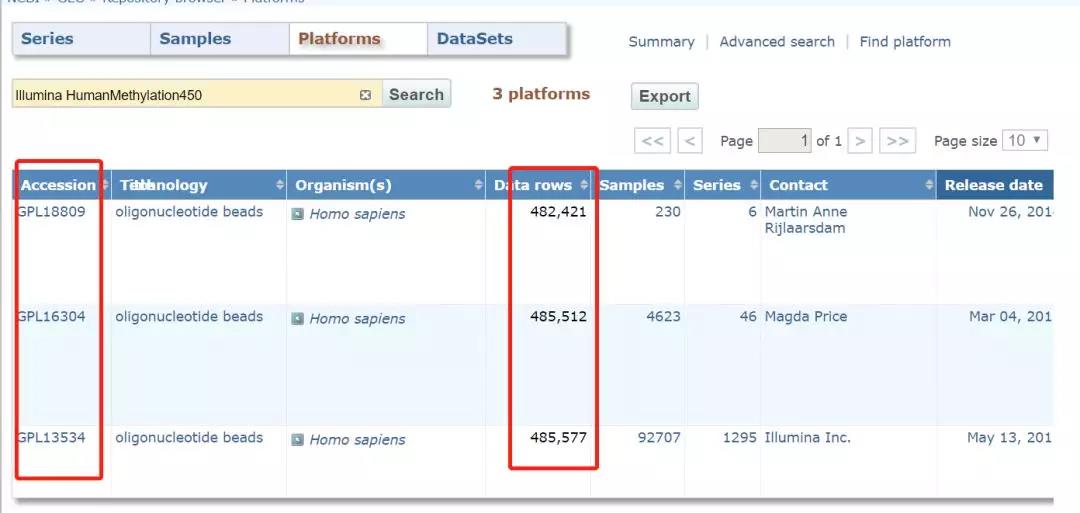
在 GEO 的官网 platform 下搜索 Illumina HumanMethylation450,可以看到 450k 的芯片主要来自三个平台,探针数也是不一样的,TCGA 中下载时一般都会标明来自那个平台,从 GEO 中下载数据都会得知平台的信息。直接进入对应平台的介绍就可以了。
看一下最常见的 GPL13534 平台的内容
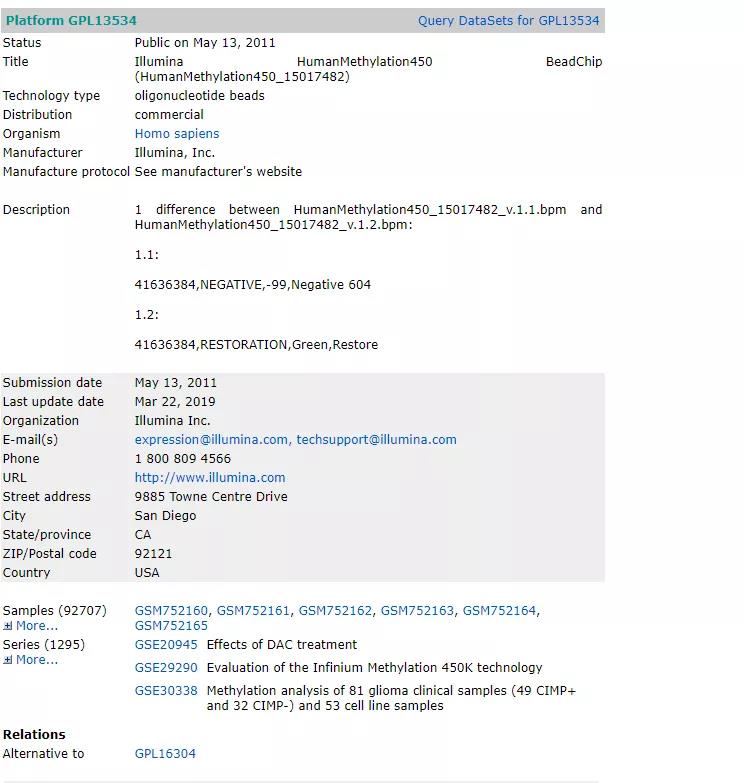
可以看到有 1295 个 GSE 数据集来自这个平台,可以利用的数据相当多,这里给出了一部分数据的概览

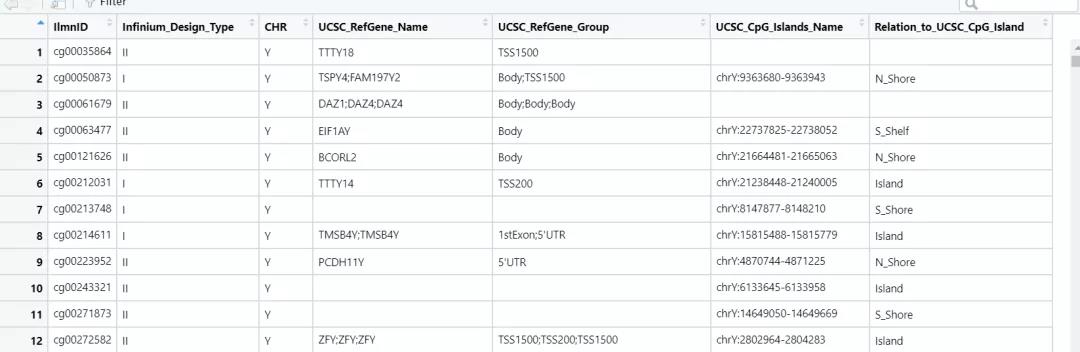
表格中展示了部分信息,直接下载然后就可以提取我们需要的注释信息了,485577 个探针一个不差,可能是因为我网速的问题,只有下载 CSV 这个的时候速度比较快,其他速度都非常感人

可以看到信息非常全面了,但实际上我们并用不到这么多,有下面这些就够了
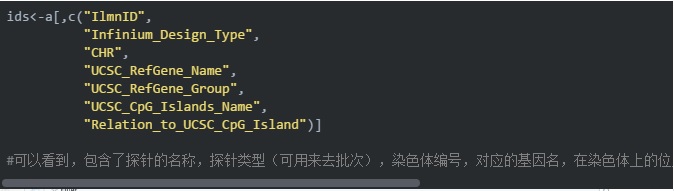
方法三:从 ChAMP 包中提取
这个方法严格来说其实是从 ChAMP 依赖的两个注释包中提取的,但是我又懒又笨,懒得看原始的包里数据藏在哪里了,ChAMP 包在做甲基化分析的时候也很方便,而其中 champ.filter 函数直接就提取好了
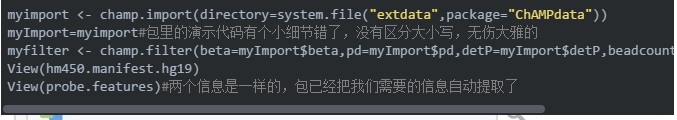
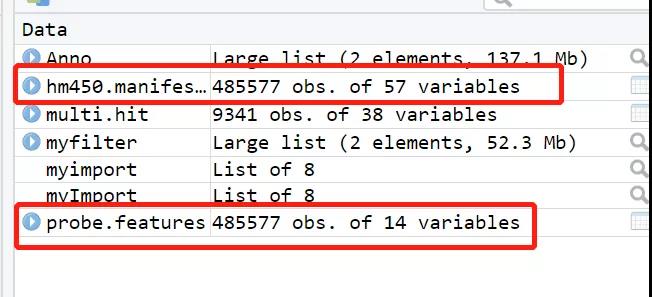
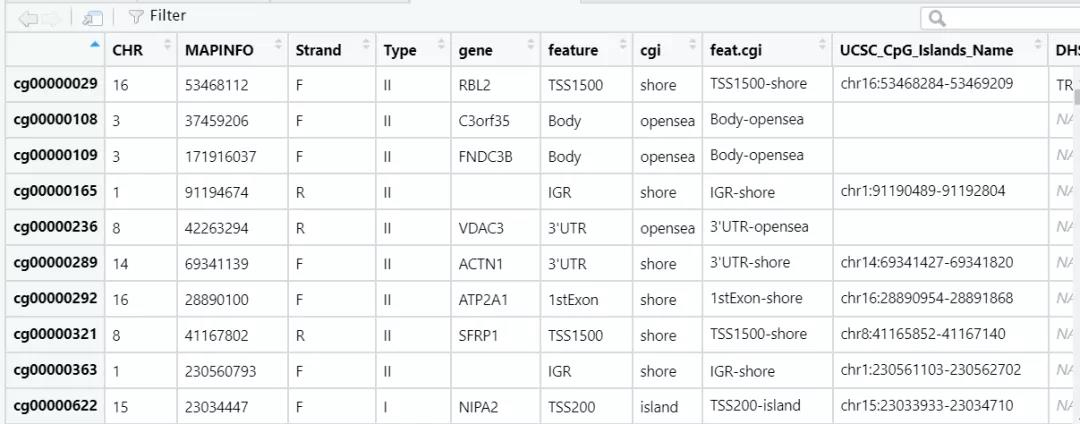
850k 和 450k 本质上没有什么区别,所以方法都是通用的。